Mehr Grafiken mit ggplot2
Erweiterte Plots mit ggplot2
Weil auch ich mich in dieser Phase der eingeschränkten Freizeitmöglichkeiten beschäftigen muss, habe ich im Folgenden ein paar zusätzliche Abbildungen erstellt, die die Grundlagen aus dem Post zu ggplot2 erweitern. Für die Grafiken hier benutze ich den gleichen Datensatz, weswegen ich hier das Erstellen und Umstrukturieren der Daten hier nicht noch einmal explizit behandele. Für die Abbildungen unten gehen wir also davon aus, dass die aktuellen COVID-Zahlen im aggregierten langen Format vorliegen. Die Abbildungen in diesem Post gehen über das Ausmaß dessen hinaus, was normalerweise in psychologischen Studien oder Berichten genutzt wird, aber falls Sie die grafische Darstellung von Daten interessiert, sollten Sie aber von diesen Möglichkeiten gehört haben.
Animationen
Was sich bei Verlaufsdaten anbietet ist es, diese auch so zu animieren, dass der Verlauf deutlich wird. Für Animationen gibt es für ggplot2 das Erweiterungspaket gganimate, das Ihnen die Möglichkeit bietet, normale ggplots um eine Achse - die Zeit - zu erweitern. Zuerst aber müssen wir gganimate laden:
library(gganimate)## Loading required package: ggplot2Damit die Darstellung nicht so unübersichtlich wird, beschränken wir die Abbildung mal auf Frankreich, Deutschland, Italien, Spanien und das Vereinigte Königreich:
covid_sel <- covid[covid$Country.Region %in% c('France', 'Germany', 'Italy', 'Spain', 'United Kingdom'), ]Nehmen wir wieder den Plot der Verläufe der bestätigten Fälle. Dieser stellt jetzt den “statischen” Anteil unserer Abbildung dar, daher können wir ihn einfach static nennen:
static <- ggplot(covid_sel, aes(x = Day, y = Confirmed, color = Country.Region)) +
geom_line() + geom_point() +
theme_light() + scale_color_discrete('Country')Im Gegensatz zu den bisherigen Abbildungen ändern wir außerdem den Titel der Legende zu “Country”, um das Ganze ein wenig aufzuhübschen. In static ist jetzt zunächst der gleiche Plot enthalten, wie bisher:
static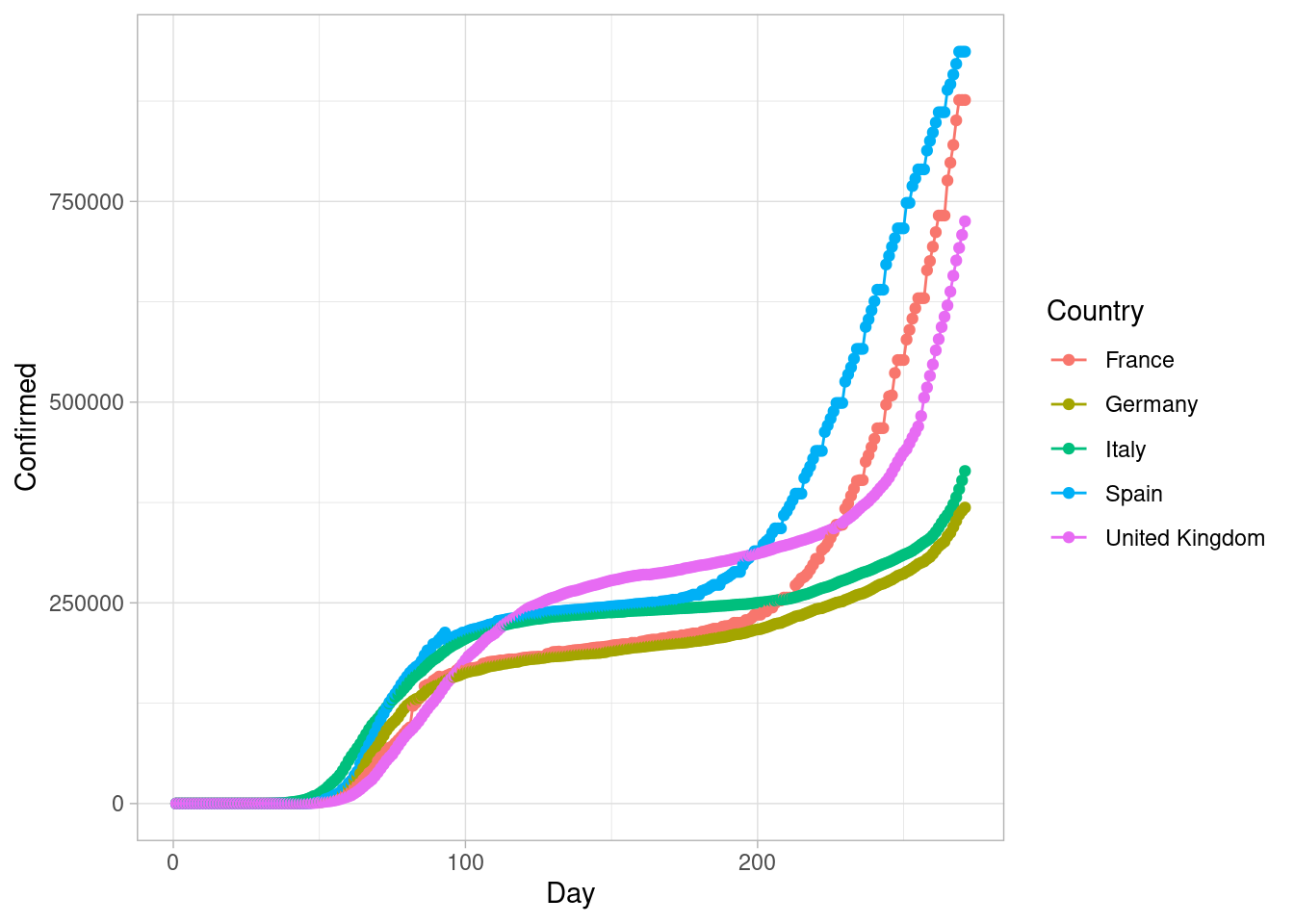
Um Animationen zu generieren, können wir mit den Funktionen von gganimate eine neue Schicht - die der Übergänge - erzeugen. Alle Funktionen, mit denen man durch das Pakte Animationen erzeugen kann beginnen mit transition_ - ähnlich wie im Kern-ggplot2 alle Geometrien mit geom_ beginnen.
Um in einer Animation nach und nach die Werte der einzelnen Tage anzuzeigen, können wir die Daten schrittweise “aufdecken” - der dazugehörige Befehl heißt also transition_reveal. Um das mit unserem bisher statistischen Plot zu verbinden, benutzen wir in ggplot Tradition weiterhin das +:
fluid <- static + transition_reveal(Day)Die Funktion transition_reveal möchte als Argument wissen, anhand welche Variable des Datensatzes wir die Zeitachse kodieren. Bei uns ist das der Tag, also Day. Wenn Sie jetzt fluid aufrufen würden, würde nach ca. 20 Sekunden renderzeit eine Animation in RStudio erscheinen. Wir fügen der Abbildung aber zunächst noch einen Titel und Untertitel hinzu. Dabei soll der Untertitel den jeweiligen Tag angeben, sich also über die Zeit ebenfalls verändern. In der Hilfe zu transition_reveal finden Sie den Abschnitt Label variables, der Ihnen verrät, welche Werte von der Animation ausgegeben werden und z.B. in die Benennung mit eingebunden werden können. In diesem Fall heißt diese Variable frame_along. Diese können wir in den Untertitel einbinden:
fluid <- static + transition_reveal(Day) +
ggtitle('Confirmed COVID-19 Cases', subtitle = 'Day {frame_along}')Somit wird in der Animation im Untertitel immer “Day X” stehen, wobei X sich im Verlauf der Animation ändert. Um dann die Abbildung zu animieren gibt es den Befehl animate, der ein paar, für uns relevante, Argumente entgegennimmt:
plot: Welche Plot soll animiert werden? Hierfluidnframes: Wieviele Frames sollen insgesamt animiert werden?fps: Wieviele Frames per Second soll die finale Animation haben?duration: Wie lang soll die Animation (in Sekunden) sein?
Von den letzten drei sind immer nur zwei nötig, weil die dritte sich dann aus den beiden Angaben ergibt. In unserem Fall mach es Sinn jedem Tag einen eigenen Frame zuzuweisen und 20 Frames pro Sekunde zu benutzen:
animate(fluid,
nframes = max(covid_sel$Day),
fps = 20)Die Animation sollte erneut ca. 20 Sekunden dauern und in folgendem gif resultieren (ich habe die Animation am 19. Oktober durchgeführt, Ihre Animation wird sich also dementsprechend unterscheiden):
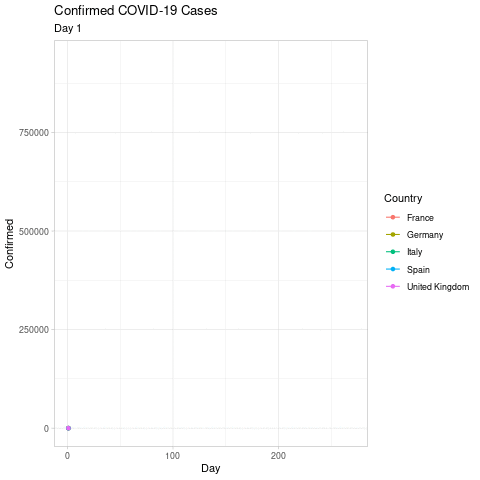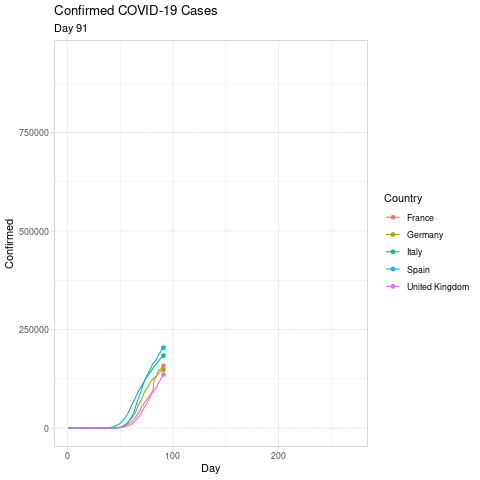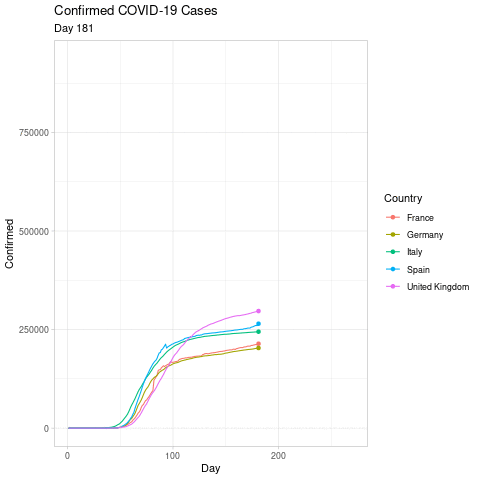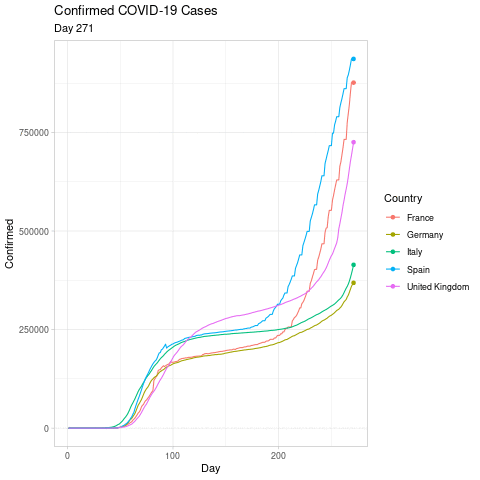
Karten
Bei psychologischen Daten eher selten, aber mit dem COVID-19 Datensatz natürlich sehr naheliegend, ist die Datenvisualisierung auf Karten. Für komplexere Karten (z.B. mit Google Maps) gibt es das ggmap Paket. Für unsere Zwecke reichen allerdings die von ggplot2 mitgelieferten Karten aus.
Karten benötigen eine sehr eigene Art der Datenaufbereitung, die häufig nicht gerade platzsparend ist. Daher sind die meisten Karten in R nicht als Datensätze vorhanden, sondern müssen erst einmal in solche überführt werden. Dafür gibt es die map_data Funktion. Um die Weltkarte in einen Datensatz zu übertragen, z.B.:
welt <- map_data('world')
head(welt)## long lat group order region subregion
## 1 -69.89912 12.45200 1 1 Aruba <NA>
## 2 -69.89571 12.42300 1 2 Aruba <NA>
## 3 -69.94219 12.43853 1 3 Aruba <NA>
## 4 -70.00415 12.50049 1 4 Aruba <NA>
## 5 -70.06612 12.54697 1 5 Aruba <NA>
## 6 -70.05088 12.59707 1 6 Aruba <NA>Was Sie in den Daten sehen sind Länge- und Breitengrade von Landesgrenzen. Außerdem bestimmt die Variable group das Land (anhand dessen die Landesgrenzen gruppiert werden sollten). Damit Linie der Grenzen nicht hin und her springt gibt es außerdem die Variable order die angibt, welcher Punkt in der Grenze als nächstes kommt. Anhand dieser Punkte werden in ggplot2 mit der allgemeinen geom_polygon Funktion Karten gezeichnet. Um eine leere Weltkarte zu erzeugen reicht Folgendes aus:
ggplot(welt, aes(x = long, y = lat, group = group)) +
geom_polygon()
Wie Sie sehen, hat dieser Plot die gleichen Eigenschaften wie normale ggplots - weil es ein ganz normaler Plot ist. Um einzelne Länder erkennen zu können, sollten wir z.B. die Länder weiß und nicht schwarz füllen. Außerdem brauchen wir nicht unbedingt x- und y-Achse, sodass wir das komplett leere Theme theme_void nutzen können:
ggplot(welt, aes(x = long, y = lat, group = group)) +
geom_polygon(fill = 'white', color = 'black', lwd = .25) +
theme_void()
Um die Karten-Daten mit den COVID Daten in Verbindung zu bringen steht uns leider - wie so häufig - im Weg, dass die Daten nicht einheitlich kodiert wurden. In diesem Fall sind es die Benennungen der Länder, die uneinheitlich sind. Um herauszufinden, wo Unterschiede bestehen, können wir die normalen Operatoren der Mengenvergleiche in R nutzen:
setdiff(unique(welt$region), unique(covid$Country.Region))## [1] "Aruba" "Anguilla"
## [3] "American Samoa" "Antarctica"
## [5] "French Southern and Antarctic Lands" "Antigua"
## [7] "Barbuda" "Saint Barthelemy"
## [9] "Bermuda" "Ivory Coast"
## [11] "Democratic Republic of the Congo" "Republic of Congo"
## [13] "Cook Islands" "Cape Verde"
## [15] "Curacao" "Cayman Islands"
## [17] "Czech Republic" "Canary Islands"
## [19] "Falkland Islands" "Reunion"
## [21] "Mayotte" "French Guiana"
## [23] "Martinique" "Guadeloupe"
## [25] "Faroe Islands" "Micronesia"
## [27] "UK" "Guernsey"
## [29] "Greenland" "Guam"
## [31] "Heard Island" "Isle of Man"
## [33] "Cocos Islands" "Christmas Island"
## [35] "Chagos Archipelago" "Jersey"
## [37] "Siachen Glacier" "Kiribati"
## [39] "Nevis" "Saint Kitts"
## [41] "South Korea" "Saint Martin"
## [43] "Marshall Islands" "Macedonia"
## [45] "Myanmar" "Northern Mariana Islands"
## [47] "Montserrat" "New Caledonia"
## [49] "Norfolk Island" "Niue"
## [51] "Bonaire" "Sint Eustatius"
## [53] "Saba" "Nauru"
## [55] "Pitcairn Islands" "Palau"
## [57] "Puerto Rico" "North Korea"
## [59] "Madeira Islands" "Azores"
## [61] "Palestine" "French Polynesia"
## [63] "South Sandwich Islands" "South Georgia"
## [65] "Saint Helena" "Ascension Island"
## [67] "Saint Pierre and Miquelon" "Swaziland"
## [69] "Sint Maarten" "Turks and Caicos Islands"
## [71] "Turkmenistan" "Tonga"
## [73] "Trinidad" "Tobago"
## [75] "Taiwan" "USA"
## [77] "Vatican" "Grenadines"
## [79] "Saint Vincent" "Virgin Islands"
## [81] "Vanuatu" "Wallis and Futuna"
## [83] "Samoa"setdiff(unique(covid$Country.Region), unique(welt$region))## [1] "Antigua and Barbuda" "Burma"
## [3] "Cabo Verde" "Congo (Brazzaville)"
## [5] "Congo (Kinshasa)" "Cote d'Ivoire"
## [7] "Czechia" "Diamond Princess"
## [9] "Eswatini" "Holy See"
## [11] "Korea, South" "MS Zaandam"
## [13] "North Macedonia" "Saint Kitts and Nevis"
## [15] "Saint Vincent and the Grenadines" "Taiwan*"
## [17] "Trinidad and Tobago" "United Kingdom"
## [19] "US" "West Bank and Gaza"Im Folgenden werden die Namen der Länder mit dem recode Befehl des car-Pakets umkodiert. Diesen hatten wir im letzten Semester zum Rekodieren negativ formulierter Items genutzt.
# Recodes
covid$Country.Region <- car::recode(covid$Country.Region,
"'Burma' = 'Myanmar';
'Cabo Verde' = 'Cape Verde';
'Congo (Brazzaville)' = 'Republic of Congo';
'Congo (Kinshasa)' = 'Democratic Republic of the Congo';
'Czechia' = 'Czech Republic';
'Eswatini' = 'Swaziland';
'Holy See' = 'Vatican';
'Korea, South' = 'South Korea';
'North Macedonia' = 'Macedonia';
'Saint Kitts and Nevis' = 'Saint Kitts';
'Saint Vincent and the Grenadines' = 'Saint Vincent';
'Taiwan*' = 'Taiwan';
'United Kingdom' = 'UK';
'US' = 'USA';
'West Bank and Gaza' = 'Palestine'")Darüber hinaus brauchen zwei Inselstaaten eine gesonderte Behandlung, weil sie auf der Karte als separate Inseln, im COVID Datensatz aber als ein Land geführt werden:
# Antigua & Barbuda
covid$Country.Region <- car::recode(covid$Country.Region, "'Antigua and Barbuda'='Antigua'")
tmp <- covid[covid$Country.Region == 'Antigua', ]
tmp$Country.Region <- 'Barbuda'
covid <- rbind(covid, tmp)
# Trinidad & Tobago
covid$Country.Region <- car::recode(covid$Country.Region, "'Trinidad and Tobago'='Trinidad'")
tmp <- covid[covid$Country.Region == 'Trinidad', ]
tmp$Country.Region <- 'Tobago'
covid <- rbind(covid, tmp)Zu guter Letzt muss die Elfenbeinküste einzeln umkodiert werden, weil der Apostroph im französischen Namen ein Problem bereitet:
levels(covid$Country.Region) <- c(levels(covid$Country.Region), 'Ivory Coast')
covid$Country.Region[covid$Country.Region == "Cote d'Ivoire"] <- 'Ivory Coast'Diese Umkodierung ist nicht auf andere Datensätze übertragbar - Sie müssen immer in den Daten, die Sie vorliegen haben nachgucken, welche Schritte zum Angleichen verschiedener Datensätze notwendig sind.
Um die Daten für heute anzuzeigen, können wir den Datensatz wieder erst einmal auf heute beschränken:
covid_today <- covid[covid$Day == max(covid$Day), ]Anschließend können wir den Datensatz mit der Weltkarte zusammenführen. Dafür verwenden wir wieder den merge Befehl. Damit nach dem merge die Grenzen richtig gezeichnet werden, müssen wieder die Reihenfolge der Daten wiederherstellen. Dazu wird mit order nach Land (group) und dann nach Reihenfolge der Grenzpunkte (order) sortiert.
covid_map <- merge(welt, covid_today, by.x = 'region', by.y = 'Country.Region', all.x = TRUE, all.y = FALSE)
covid_map <- covid_map[order(covid_map$group, covid_map$order), ]Mit den neuen Daten können wir unsere vorherige Karte jetzt so ergänzen, dass wir die Länder nach der Anzahl der Fälle einfärben:
ggplot(covid_map, aes(x = long, y = lat, group = group)) +
geom_polygon(color = 'black', lwd = .25, aes(fill = Confirmed)) +
theme_void()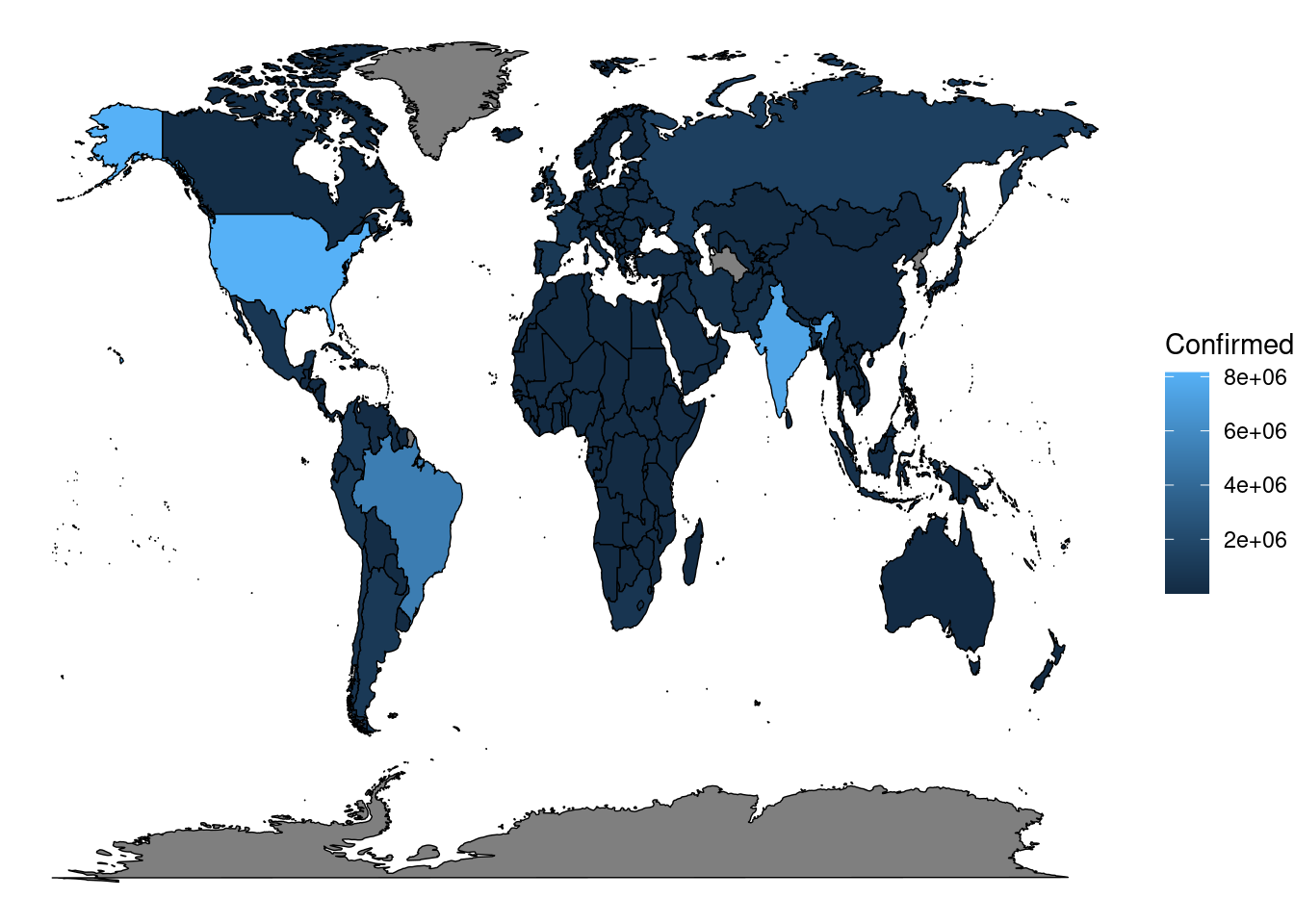
Wegen der exponentiellen Art und Weise mit der die Anzahl der Fälle in betroffenen Ländern zunimmt, sind mit dieser Skalierung nur wenige Länder überhaupt unterscheidbar. Das liegt besonders an der exorbitanten Anzahl bestätigter Fälle in den USA. Um solche Situationen zu umgehen wird in der Datenvisualisierung häufig mit logarithmischen Skalen gearbeitet. Das Gleiche können wir hier auch mit dem Argument trans für Funktionen tun, die mit scale_ beginnen. Darüber hinaus gefällt mir die Farbgebung nicht, sodass ich mit scale_fill_gradient2 drei Farben aussuchen werden, die Untergrenze, Mittelpunkt und Obergrenze der bestätigten Fälle kodieren:
ggplot(covid_map, aes(x = long, y = lat, group = group)) +
geom_polygon(color = 'black', lwd = .25, aes(fill = Confirmed)) +
theme_void() +
scale_fill_gradient2(low = '#737c45', mid = '#e3ba0f', high = '#ad3b76',
trans = 'log2',
midpoint = log(median(covid_map$Confirmed, na.rm = TRUE), 2))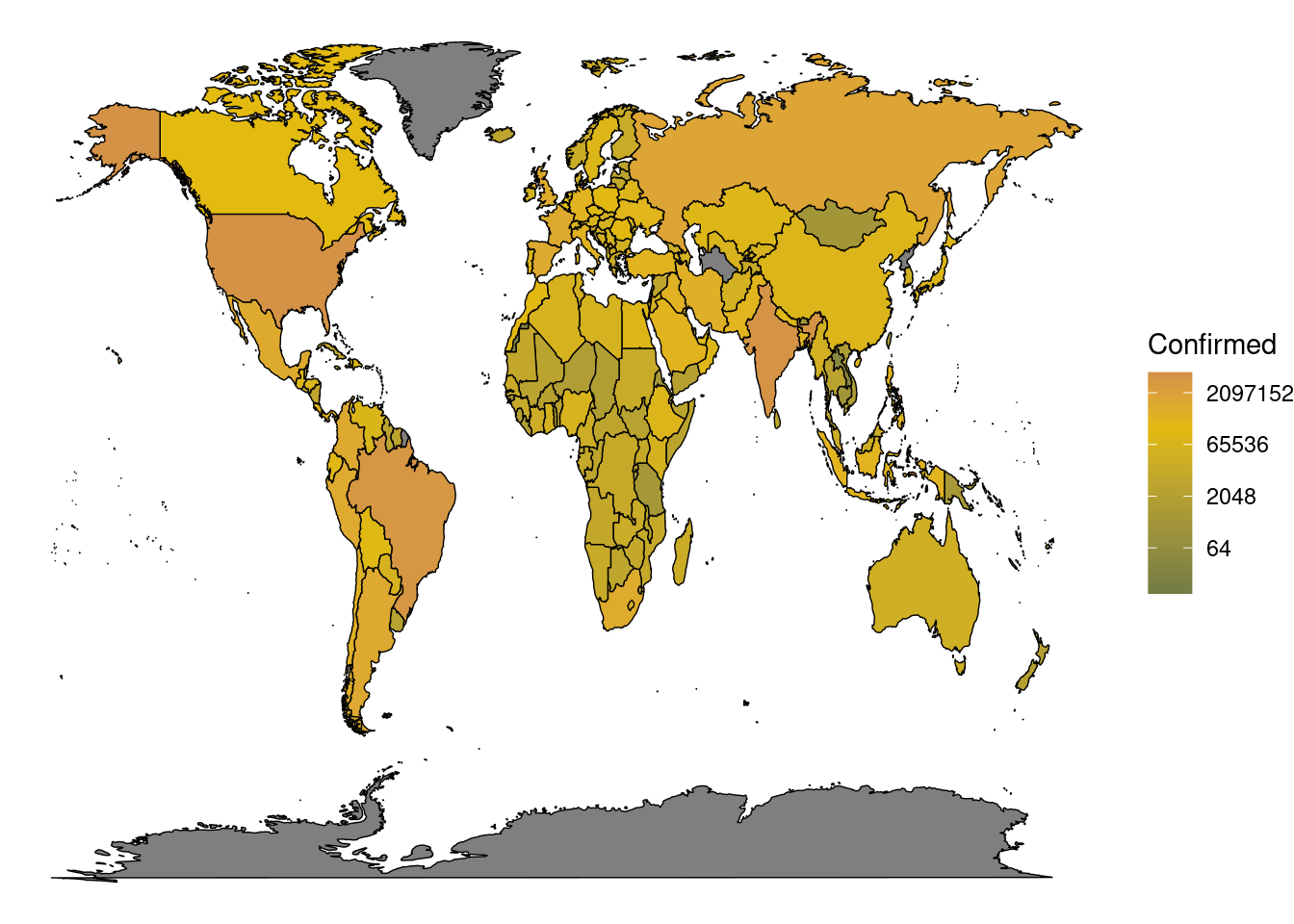
Per Voreinstellung ist der Mittelpunkt der Skala bei 0 - was in unserem Fall nicht sonderlich sinnvoll ist. Stattdessen wird hier über midpoint der aktuelle Median als Mittelpunkt der Skala definiert.
Im letzten Schritt werden die Farbe, die bei fehlenden Werten vergeben wird, etwas angepasst und die Skala wird von der etwas befremdlich wirkenden Beschriftung in 10, 1000 und 100000 überführt. Darüber hinaus wird die Legende nach unten Verschoben, um der Karte genug Platz in die Breite zu geben. Mit scipen wird außerdem vorübergehend die wissenschaftliche Notation der Zahlen ausgesetzt:
options(scipen = 3)
ggplot(covid_map, aes(x = long, y = lat, group = group)) +
geom_polygon(color = 'black', lwd = .25, aes(fill = Confirmed)) +
theme_void() +
scale_fill_gradient2(trans = 'log2', low = '#737c45', mid = '#e3ba0f', high = '#ad3b76', na.value = 'grey95', midpoint = log(median(covid_map$Confirmed, na.rm = TRUE), 2),
breaks = c(10, 1000, 100000), name = 'Confirmed\n(Log-Scale)') +
theme(legend.position = 'bottom')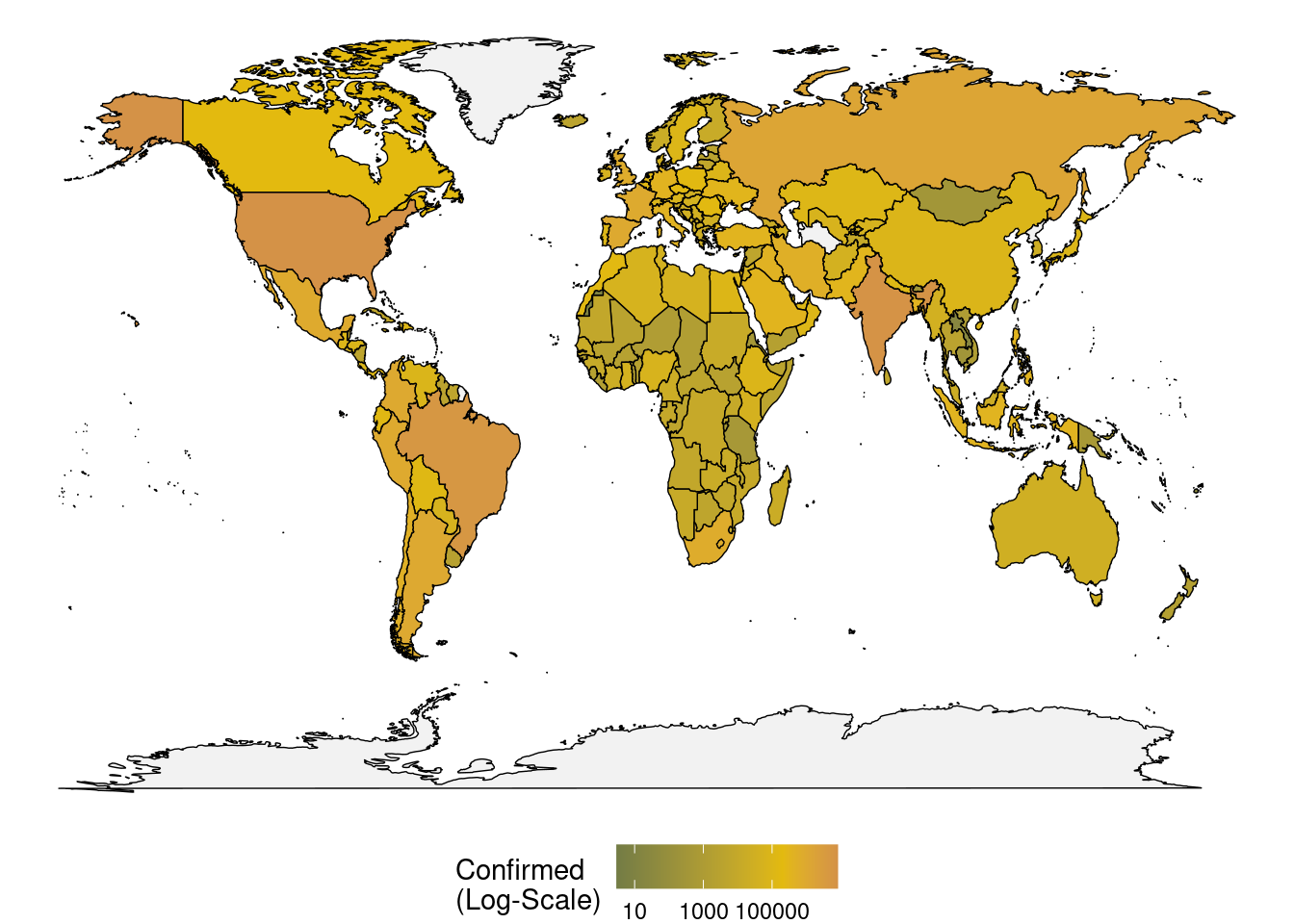
options(scipen = 1)