Korrelation
Kernfragen dieser Lehreinheit
- Wie können Kreuztabellen in R erstellt werden? Welche Varianten gibt es, relative Häufigkeitstabellen zu erstellen?
- Wie kann ein gemeinsames Balkendiagramm für zwei Variablen erstellt werden?
- Welche zwei Varianten gibt es, Varianzen und Kovarianzen zu bestimmen?
- Wie kann die Produkt-Moment-Korrelation, die Rang-Korrelation nach Spearman und Kendalls \(\tau\) bestimmt werden?
- Wie wird bei der Berechnung von Korrelationen mit fehlenden Werten umgegangen?
Vorbereitende Schritte
Zu Beginn laden wir wie gewohnt den Datensatz und verteilen die relevanten Labels. Beachten Sie, dass diese Befehle bereits angewendet wurden. Wenn Sie die veränderten Daten abgespeichert oder noch aktiv haben, sind die folgenden Befehle natürlich nicht nötig.
#### Was bisher geschah: ----
# Daten laden
load(url('https://pandar.netlify.app/post/fb22.rda'))
# Nominalskalierte Variablen in Faktoren verwandeln
fb22$geschl_faktor <- factor(fb22$geschl,
levels = 1:3,
labels = c("weiblich", "männlich", "anderes"))
fb22$fach <- factor(fb22$fach,
levels = 1:5,
labels = c('Allgemeine', 'Biologische', 'Entwicklung', 'Klinische', 'Diag./Meth.'))
fb22$ziel <- factor(fb22$ziel,
levels = 1:4,
labels = c("Wirtschaft", "Therapie", "Forschung", "Andere"))
fb22$wohnen <- factor(fb22$wohnen,
levels = 1:4,
labels = c("WG", "bei Eltern", "alleine", "sonstiges"))
fb22$ort <- factor(fb22$ort, levels=c(1,2), labels=c("FFM", "anderer"))
fb22$job <- factor(fb22$job, levels=c(1,2), labels=c("nein", "ja"))
# Skalenbildung
fb22$prok2_r <- -1 * (fb22$prok2 - 5)
fb22$prok3_r <- -1 * (fb22$prok3 - 5)
fb22$prok5_r <- -1 * (fb22$prok5 - 5)
fb22$prok7_r <- -1 * (fb22$prok7 - 5)
fb22$prok8_r <- -1 * (fb22$prok8 - 5)
# Prokrastination
fb22$prok_ges <- fb22[, c('prok1', 'prok2_r', 'prok3_r',
'prok4', 'prok5_r', 'prok6',
'prok7_r', 'prok8_r', 'prok9',
'prok10')] |> rowMeans()
# Naturverbundenheit
fb22$nr_ges <- fb22[, c('nr1', 'nr2', 'nr3', 'nr4', 'nr5', 'nr6')] |> rowMeans()
fb22$nr_ges_z <- scale(fb22$nr_ges) # Standardisiert
# Weitere Standardisierungen
fb22$nerd_std <- scale(fb22$nerd)
fb22$neuro_std <- scale(fb22$neuro)Häufigkeitstabellen
Die Erstellung von Häufigkeitstabellen zur Darstellung univariater Häufigkeiten haben Sie schon kennengelernt. Dies funktioniert mit einfachen Befehlen für die Häufigkeiten und die zugehörigen relativen Prozentzahlen.
tab <- table(fb22$fach) #Absolut
tab##
## Allgemeine Biologische Entwicklung Klinische Diag./Meth.
## 19 27 37 57 7prop.table(tab) #Relativ##
## Allgemeine Biologische Entwicklung Klinische Diag./Meth.
## 0.12925170 0.18367347 0.25170068 0.38775510 0.04761905Die Erweiterung für den bivariaten Fall ist dabei nicht schwierig und wird als Kreuztabelle bezeichnet. Sie liefert die Häufigkeit von Kombinationen von Ausprägungen in mehreren Variablen. In den Zeilen wird die erste Variable abgetragen und in den Spalten die zweite. Im Unterschied zum univariaten Fall muss im table()-Befehl nur die zweite interessierende Variable zusätzlich genannt werden. Tabellen können beliebig viele Dimensionen haben, werden dann aber sehr unübersichtlich.
tab<-table(fb22$fach,fb22$ziel) #Kreuztabelle
tab##
## Wirtschaft Therapie Forschung Andere
## Allgemeine 4 3 8 4
## Biologische 6 6 11 4
## Entwicklung 8 18 6 5
## Klinische 2 50 1 4
## Diag./Meth. 0 1 6 0In eine Kreuztabelle können Randsummen mit dem addmargins() Befehl hinzugefügt werden. Randsummen erzeugen in der letzten Spalte bzw. Zeile die univariaten Häufigkeitstabellen der Variablen.
addmargins(tab) #Randsummen hinzufügen##
## Wirtschaft Therapie Forschung Andere Sum
## Allgemeine 4 3 8 4 19
## Biologische 6 6 11 4 27
## Entwicklung 8 18 6 5 37
## Klinische 2 50 1 4 57
## Diag./Meth. 0 1 6 0 7
## Sum 20 78 32 17 147Auch für die Kreuztabelle ist die Möglichkeit der Darstellung der Häufigkeiten in Relation zur Gesamtzahl der Beobachtungen gegeben.
prop.table(tab) #Relative Häufigkeiten##
## Wirtschaft Therapie Forschung Andere
## Allgemeine 0.027210884 0.020408163 0.054421769 0.027210884
## Biologische 0.040816327 0.040816327 0.074829932 0.027210884
## Entwicklung 0.054421769 0.122448980 0.040816327 0.034013605
## Klinische 0.013605442 0.340136054 0.006802721 0.027210884
## Diag./Meth. 0.000000000 0.006802721 0.040816327 0.00000000050 von insgesamt 147 (34.01%) wollen therapeutisch arbeiten und interessieren sich bisher am meisten für die klinische Psychologie.
prob.table() kann allerdings nicht nur an der Gesamtzahl relativiert werden, sondern auch an der jeweiligen Zeilen- oder Spaltensumme. Dafür gibt man im Argument margin für Zeilen 1 oder für Spalten 2 an.
prop.table(tab, margin = 1) #relativiert an Zeilen##
## Wirtschaft Therapie Forschung Andere
## Allgemeine 0.21052632 0.15789474 0.42105263 0.21052632
## Biologische 0.22222222 0.22222222 0.40740741 0.14814815
## Entwicklung 0.21621622 0.48648649 0.16216216 0.13513514
## Klinische 0.03508772 0.87719298 0.01754386 0.07017544
## Diag./Meth. 0.00000000 0.14285714 0.85714286 0.00000000Von 57 Personen, die sich am meisten für klinische Psychologie interessieren, wollen 87.72% (nämlich 50 Personen) später therapeutisch arbeiten.
prop.table(tab, margin = 2) #relativiert an Spalten##
## Wirtschaft Therapie Forschung Andere
## Allgemeine 0.20000000 0.03846154 0.25000000 0.23529412
## Biologische 0.30000000 0.07692308 0.34375000 0.23529412
## Entwicklung 0.40000000 0.23076923 0.18750000 0.29411765
## Klinische 0.10000000 0.64102564 0.03125000 0.23529412
## Diag./Meth. 0.00000000 0.01282051 0.18750000 0.00000000Von 78 Personen, die später therapeutisch arbeiten wollen, interessieren sich 64.1% (nämlich 50 Personen) für die klinische Psychologie.
addmargins()und prop.table() können beliebig kombiniert werden.
prop.table(addmargins(tab)) behandelt die Randsummen als eigene Kategorie (inhaltlich meist unsinnig!).
addmargins(prop.table(tab)) liefert die Randsummen der relativen Häufigkeiten.
addmargins(prop.table(tab)) # als geschachtelte Funktion##
## Wirtschaft Therapie Forschung Andere Sum
## Allgemeine 0.027210884 0.020408163 0.054421769 0.027210884 0.129251701
## Biologische 0.040816327 0.040816327 0.074829932 0.027210884 0.183673469
## Entwicklung 0.054421769 0.122448980 0.040816327 0.034013605 0.251700680
## Klinische 0.013605442 0.340136054 0.006802721 0.027210884 0.387755102
## Diag./Meth. 0.000000000 0.006802721 0.040816327 0.000000000 0.047619048
## Sum 0.136054422 0.530612245 0.217687075 0.115646259 1.000000000prop.table(tab) |> addmargins() # als Pipe##
## Wirtschaft Therapie Forschung Andere Sum
## Allgemeine 0.027210884 0.020408163 0.054421769 0.027210884 0.129251701
## Biologische 0.040816327 0.040816327 0.074829932 0.027210884 0.183673469
## Entwicklung 0.054421769 0.122448980 0.040816327 0.034013605 0.251700680
## Klinische 0.013605442 0.340136054 0.006802721 0.027210884 0.387755102
## Diag./Meth. 0.000000000 0.006802721 0.040816327 0.000000000 0.047619048
## Sum 0.136054422 0.530612245 0.217687075 0.115646259 1.000000000Balkendiagramme
Grafisch kann eine solche Kreuztabelle durch gruppierte Balkendiagramme dargestellt werden. Das Argument beside sorgt für die Anordnung der Balken (bei TRUE nebeneinander, bei FALSE übereinander). Das Argument legend nimmt einen Vektor für die Beschriftung entgegen. Die Zeilen des Datensatzes bilden dabei stets eigene Balken, während die Spalten die Gruppierungsvariable bilden. Deshalb müssen als Legende die Namen der Reihen rownames() unserer Tabelle tab ausgewählt werden.
barplot (tab,
beside = TRUE,
col = c('mintcream','olivedrab','peachpuff','steelblue','maroon'),
legend = rownames(tab))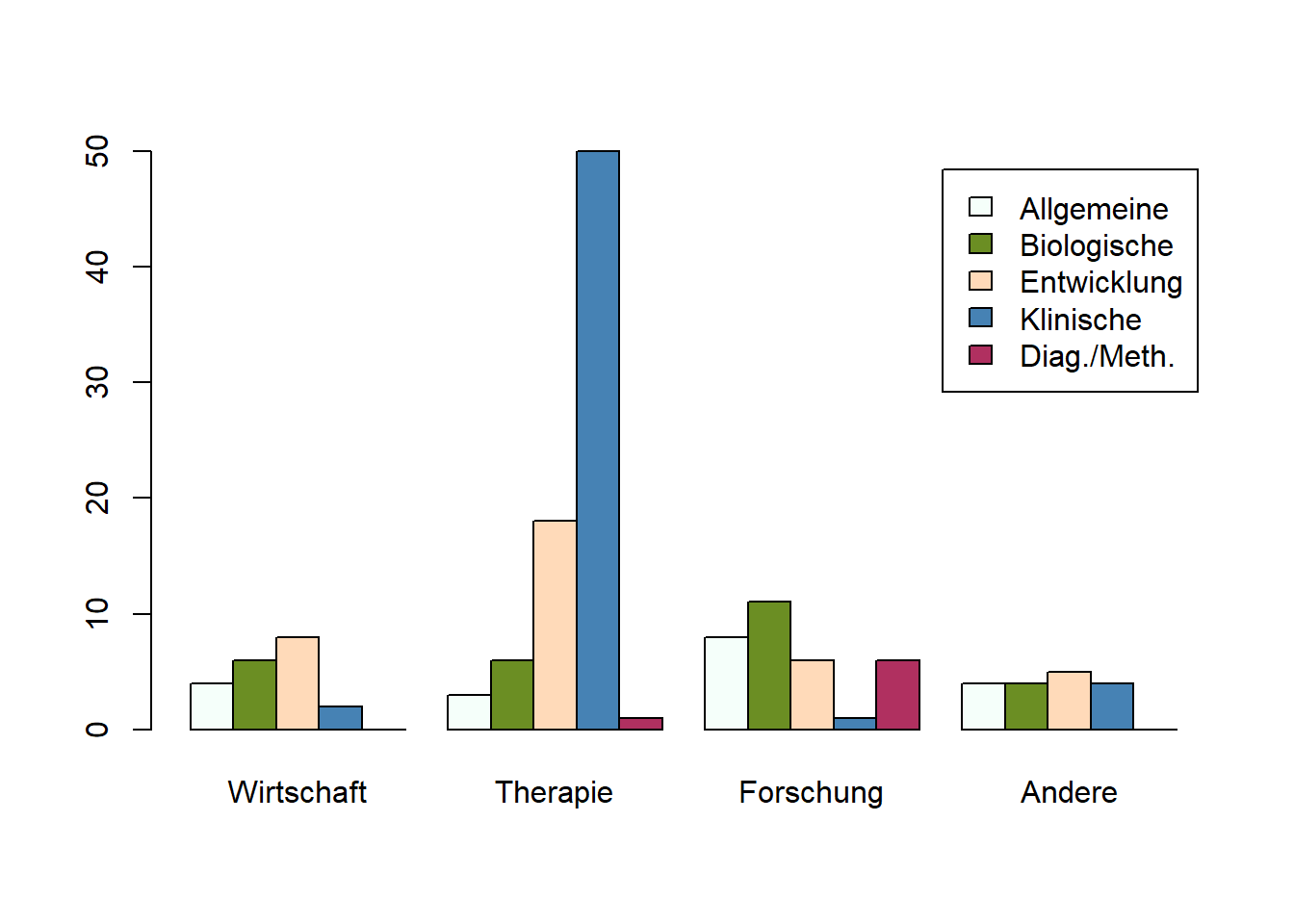
Varianz, Kovarianz und Korrelation
In der Vorlesungen haben Sie gelernt, dass es für Kovarianzen und Varianzen empirische und geschätzte Werte gibt. R berechnet standardmäßig für die Varianz und Kovarianz die Populationsschätzer, verwendet also folgende Formeln für Varianz
\[\hat{\sigma}^2_{X} = \frac{\sum_{m=1}^n (y_m - \bar{y})^2}{n-1}\]
und Kovarianz.
\[\hat{\sigma}_{XY} = \frac{\sum_{m=1}^n (x_m - \bar{x}) \cdot (y_m - \bar{y})}{n-1}\]
Die Funktionen für die Varianz ist dabei var(). Im Folgenden wird diese für die Variablen vertr (Verträglichkeit) und gewis (Gewissenhaftigkeit) aus dem Datensatz bestimmt. Als Argumente müssen jeweils die Variablennamen verwendet werden.
Wie bereits in vergangenen Sitzungen gesehen, führen fehlende Werte zu der Ausgabe NA. Um dies vorzubeugen, wird im univariaten Fall na.rm = TRUE zum Ausschluss verwendet.
var(fb22$vertr, na.rm = TRUE) #Varianz Verträglichkeit## [1] 0.3337015var(fb22$gewis, na.rm = TRUE) #Varianz Gewissenhaftigkeit## [1] 0.4389081Die Funktion cov() wird für die Kovarianz verwendet und benötigt als Argumente die Variablen.
cov(fb22$vertr, fb22$gewis) #Kovarianz Verträglichkeit und Gewissenhaftigkeit## [1] 0.07689475Da Kovarianzen unstandardisierte Kennzahlen sind, können wir Kovarianzen nicht pauschal nach ihrer Höhe beurteilen. Die Höhe hängt beispielsweise von der Antwortskala ab.
Natürlich können auch bei der Kovarianzberechnung fehlende Werte zu einem Problem werden. Zur Bewältigung des Problems gibt es das Argument use. Bei Zusammenhangsmaßen gibt es in R mehrere Möglichkeiten für den Umgang mit fehlenden Werten, die sich nur unterscheiden, wenn mehr als zwei Variablen korreliert werden:
- Paarweiser Fallausschluss: Personen, die auf (mindestens) einer von zwei Variablen
NAhaben, werden von der Berechnung ausgeschlossen. - Listenweiser Fallausschluss: Personen, die auf (mindestens) einer von allen Variablen
NAhaben, werden von der Berechnung ausgeschlossen. - na.or.complete: Zeilen, die einen fehlenden Wert (
NA) enthalten, werden bei den Berechnungen ignoriert. Das entspricht der Angabe vonna.rm = TRUEbei der Betrachtung von lediglich zwei Variablen.
Am besten lässt sich der Unterschied in einer Kovarianzmatrix veranschaulichen. Hier werden alle Varianzen und Kovarianzen von einer Menge an Variablen berechnet und in einer Tabelle darstellt. Dafür muss ein Datensatz erstellt werden, der nur die interessierenden Variablen enthält. Zu unseren beiden Variablen nehmen wir als drittes noch die Lebenszufriedenheit (lz) auf.
drei <- fb22[, c('vertr','gewis','lz')] #Datensatzreduktion
cov(drei) #Kovarianzmatrix ## vertr gewis lz
## vertr 0.33370154 0.07689475 NA
## gewis 0.07689475 0.43890813 NA
## lz NA NA NADa die fehlenden Werte nicht entfernt wurden, gibt R NA aus.
Nun folgt die Gegenüberstellung der beiden betrachteten Möglichkeiten zum Ausschluss.
Zu Illustrationszwecken setzen wir nun den Wert in Verträglichkeit in Zeilen 50 und 72 auf fehlend:
fb22$vertr_neu <- fb22$vertr # erstelle neue Variable vertr_neu
fb22[c(50,72), 'vertr_neu'] <- NA # setze vertr_neu in den Zeilen 50 und 72 auf fehlend
drei_neu <- fb22[, c('vertr_neu','gewis','lz')] #Datensatzreduktion
cov(drei_neu) #Kovarianzmatrix ## vertr_neu gewis lz
## vertr_neu NA NA NA
## gewis NA 0.4389081 NA
## lz NA NA NAVergleichen wir nun dieses Ergebnis mit dem Ergebnis nach paarweisem Fallausschluss und listenweisem Fallausschluss:
cov(drei_neu, use = 'pairwise') #Paarweiser Fallausschluss## vertr_neu gewis lz
## vertr_neu 0.33465519 0.07675721 0.08361123
## gewis 0.07675721 0.43890813 0.21902866
## lz 0.08361123 0.21902866 1.15304916cov(drei_neu, use = 'complete') #Listenweiser Fallausschluss## vertr_neu gewis lz
## vertr_neu 0.33083892 0.06784667 0.08361123
## gewis 0.06784667 0.43618035 0.22378090
## lz 0.08361123 0.22378090 1.15291831Wie wir sehen, unterscheiden sich die Werte voneinander, da beim listenweisen Fallausschluss noch mehr Personen von Beginn an von der Berechnung ausgeschlossen werden (es werden hier auch die Personen in Zeilen 50 und 72 für die Berechnung der Kovarianz von Gewissenhaftigkeit und Lebenszufriedenheit ausgeschlossen - obwohl diese beiden Personen auf diesen beiden Variablen eigentlich gültige Werte besitzen). Anmerkung: Die Kovarianz einer Variablen mit sich selbst (zu finden in der Hauptdiagonalen) entspricht ihrer Varianz.
Der Zusammenhang zwischen zwei Variablen kann in einem Scatterplot bzw. Streupunktdiagramm dargestellt werden. Dafür kann man die plot() Funktion nutzen. Als Argumente können dabei x für die Variable auf der x-Achse, y für die Variable auf der y-Achse, xlim, ylim für eventuelle Begrenzungen der Achsen und pch für die Punktart angegeben werden.
plot(x = fb22$vertr, y = fb22$gewis, xlim = c(1,5) , ylim = c(1,5))
Wie in der Vorlesung besprochen, sind für verschiedene Skalenniveaus verschiedene Zusammenhangsmaße verfügbar, die im Gegensatz zur Kovarianz auch eine Vergleichbarkeit zwischen zwei Zusammenhangswerten sicherstellen. Für zwei metrisch skalierte Variablen gibt es dabei die Produkt-Moment-Korrelation. In der Funktion cor() werden dabei die Argumente x und y für die beiden betrachteten Variablen benötigt. use beschreibt weiterhin den Umgang mit fehlenden Werten.
cor(x = fb22$vertr, y = fb22$gewis, use = 'pairwise')## [1] 0.2009235Bei einer positiven Korrelation gilt „je mehr Variable x… desto mehr Variable y” bzw. umgekehrt, bei einer negativen Korrelation „je mehr Variable x… desto weniger Variable y” bzw. umgekehrt. Korrelationen sind immer ungerichtet, das heißt, sie enthalten keine Information darüber, welche Variable eine andere vorhersagt - beide Variablen sind gleichberechtigt. Korrelationen (und Regressionen, die wir später in einem Tutorial kennen lernen werden) liefern keine Hinweise auf Kausalitäten. Sie sagen beide etwas über den (linearen) Zusammenhang zweier Variablen aus.
In R können wir uns auch eine Korrelationsmatrix ausgeben lassen. Dies geschieht äquivalent zu der Kovarianzmatrix mit dem Datensatz als Argument in der cor() Funktion. In der Diagonale stehen die Korrelationen der Variable mit sich selbst - also 1 - und in den restlichen Feldern die Korrelationen der Variablen untereinander.
cor(drei, use = 'pairwise')## vertr gewis lz
## vertr 1.0000000 0.2009235 0.1384518
## gewis 0.2009235 1.0000000 0.3104911
## lz 0.1384518 0.3104911 1.0000000Die Stärke des korrelativen Zusammenhangs wird mit dem Korrelationskoeffizienten ausgedrückt, der zwischen -1 und +1 liegt.
Die default-Einstellung bei cor()ist die Produkt-Moment-Korrelation, also die Pearson-Korrelation.
cor(fb22$vertr, fb22$gewis, use = "pairwise", method = "pearson")## [1] 0.2009235Achtung! Die inferenzstatistische Testung der Pearson-Korrelation hat gewisse Voraussetzungen, die vor der Durchführung überprüft werden sollten!
Voraussetzungen Pearson-Korrelation:
- Skalenniveau: intervallskalierte Daten \(\rightarrow\) ok (Ratingskalen werden meist als intervallskaliert aufgefasst, auch wenn das nicht 100% korrekt ist)
- Linearität: Zusammenhang muss linear sein \(\rightarrow\) grafische Überprüfung (siehe Scatterplot)
- Normalverteilung: Variablen müssen normalverteilt sein \(\rightarrow\) QQ-Plot, Histogramm oder Shapiro-Wilk-Test
zu 3. Normalverteilung
\(\rightarrow\) QQ-Plot, Histogramm & Shapiro-Wilk-Test
#QQ
qqnorm(fb22$vertr)
qqline(fb22$vertr)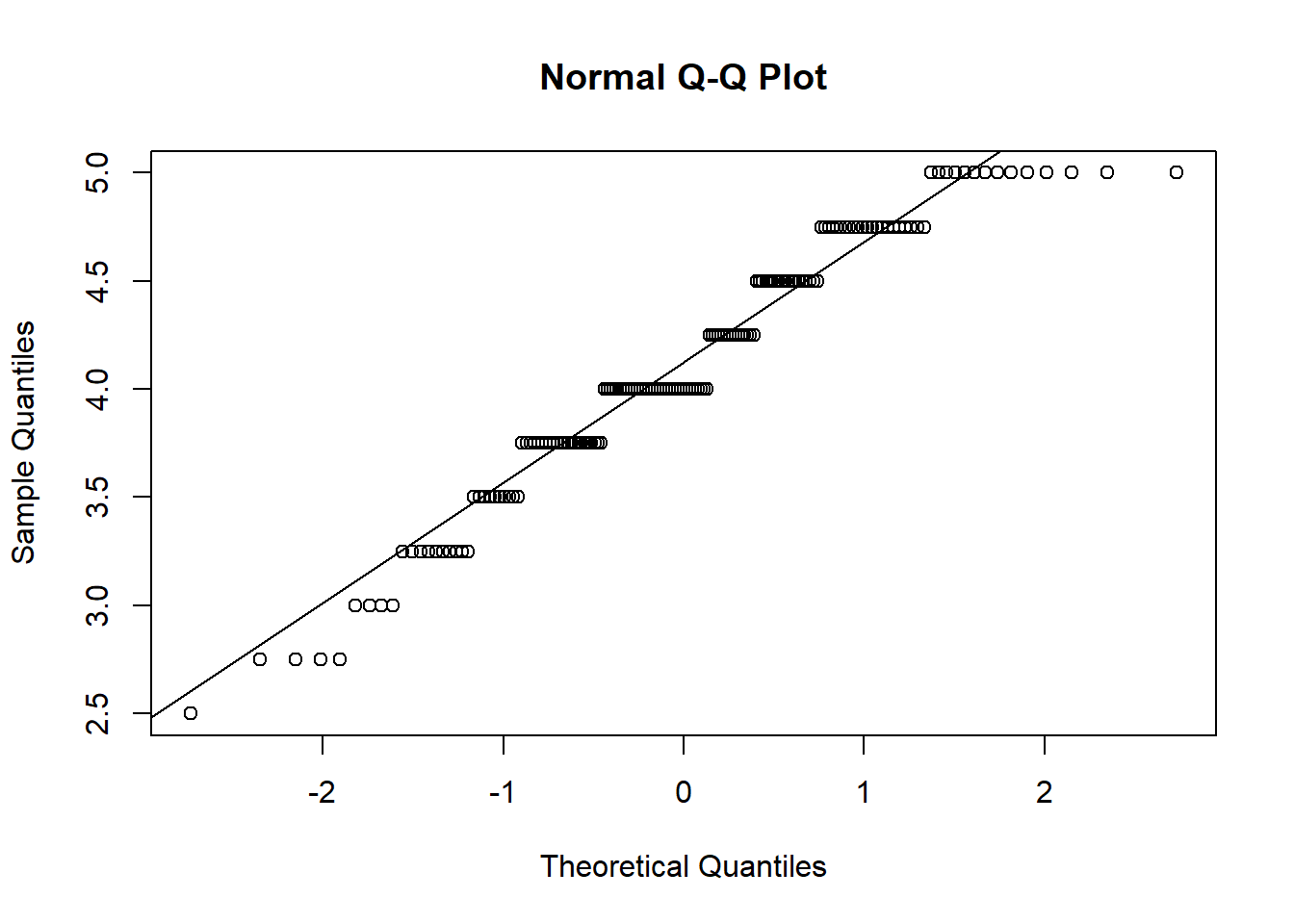
qqnorm(fb22$gewis)
qqline(fb22$gewis)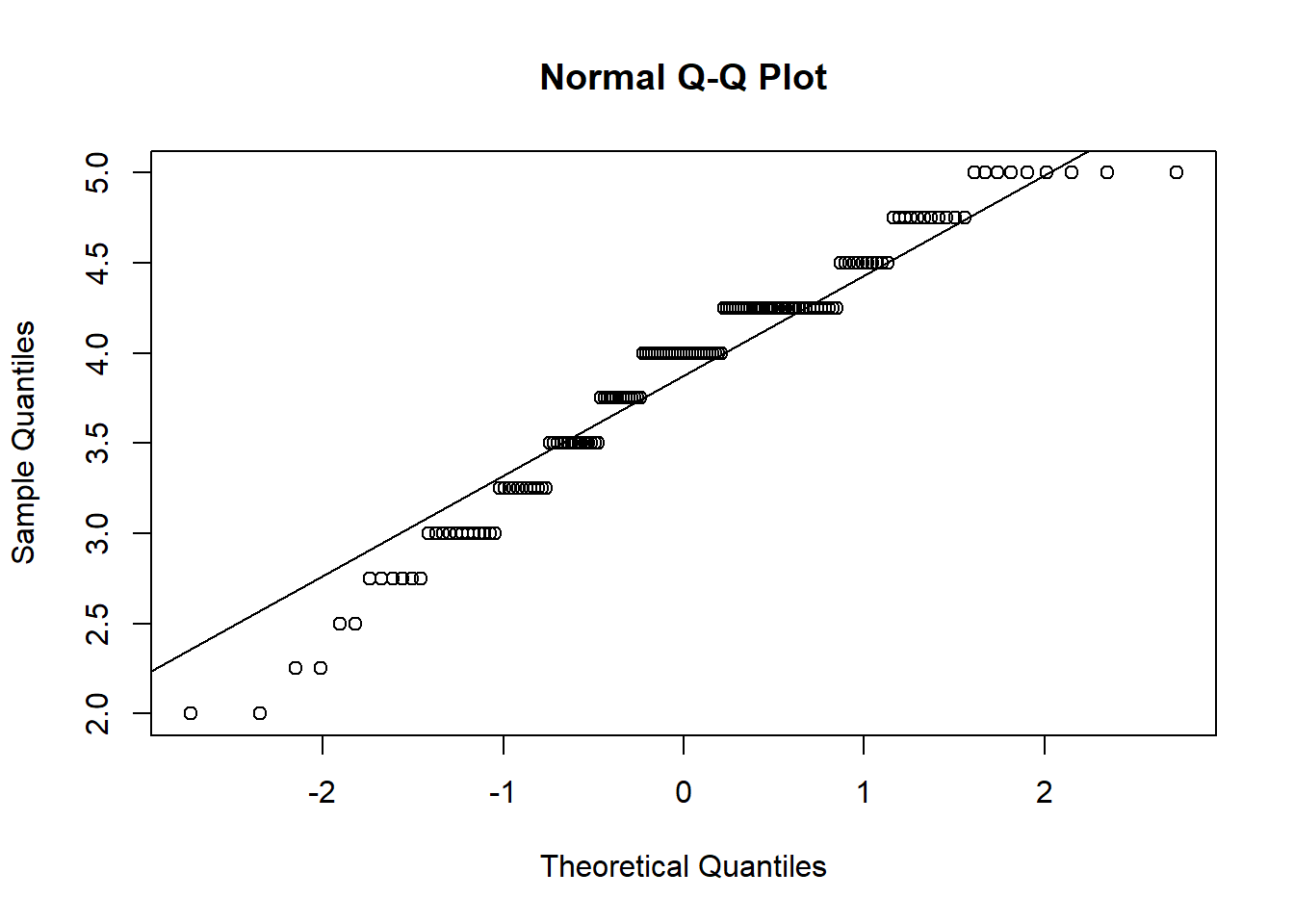
#Histogramm
hist(fb22$vertr, prob = T, ylim = c(0, 1))
curve(dnorm(x, mean = mean(fb22$vertr, na.rm = T), sd = sd(fb22$vertr, na.rm = T)), col = "blue", add = T) 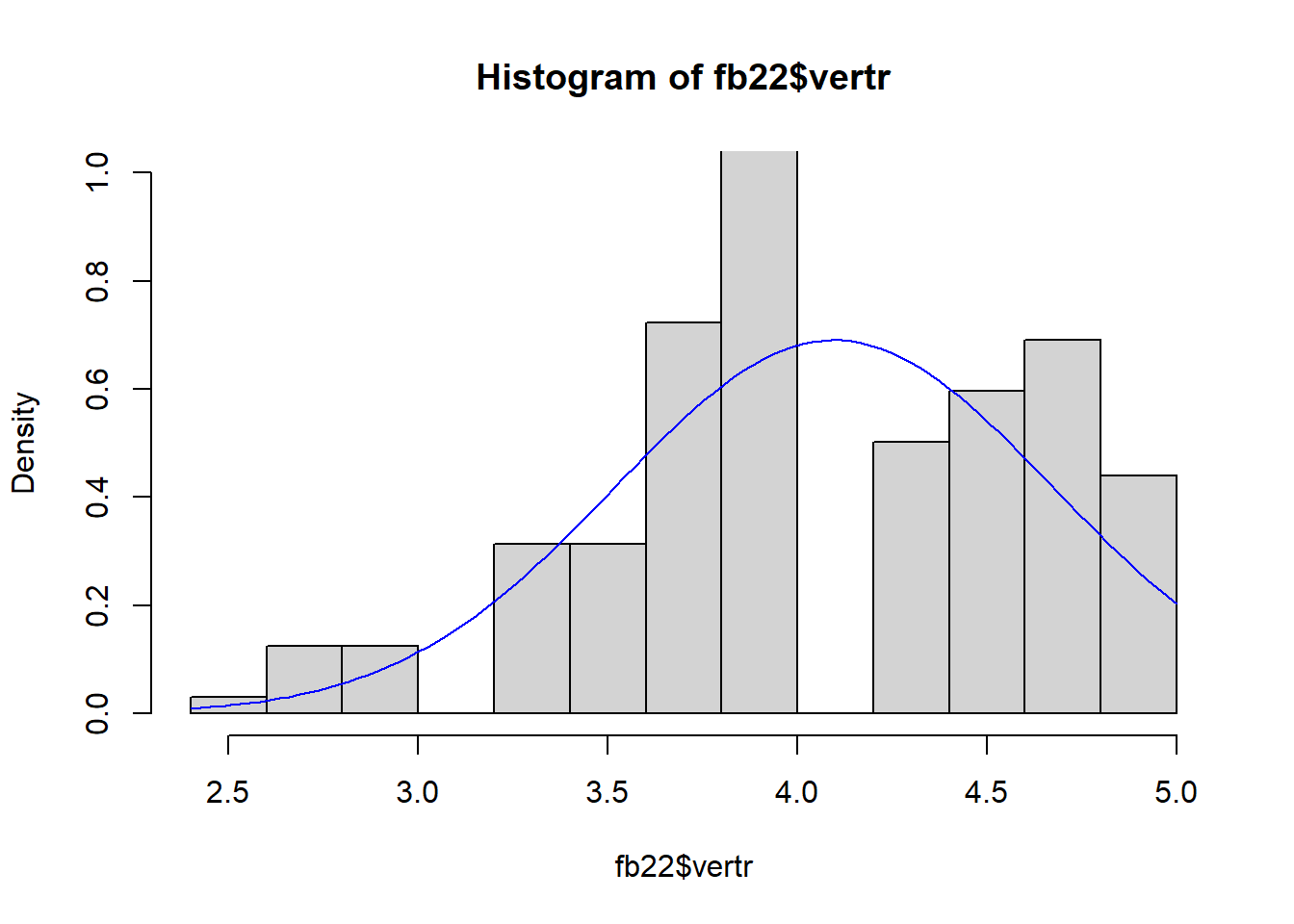
hist(fb22$gewis, prob = T, ylim = c(0,1))
curve(dnorm(x, mean = mean(fb22$gewis, na.rm = T), sd = sd(fb22$gewis, na.rm = T)), col = "blue", add = T)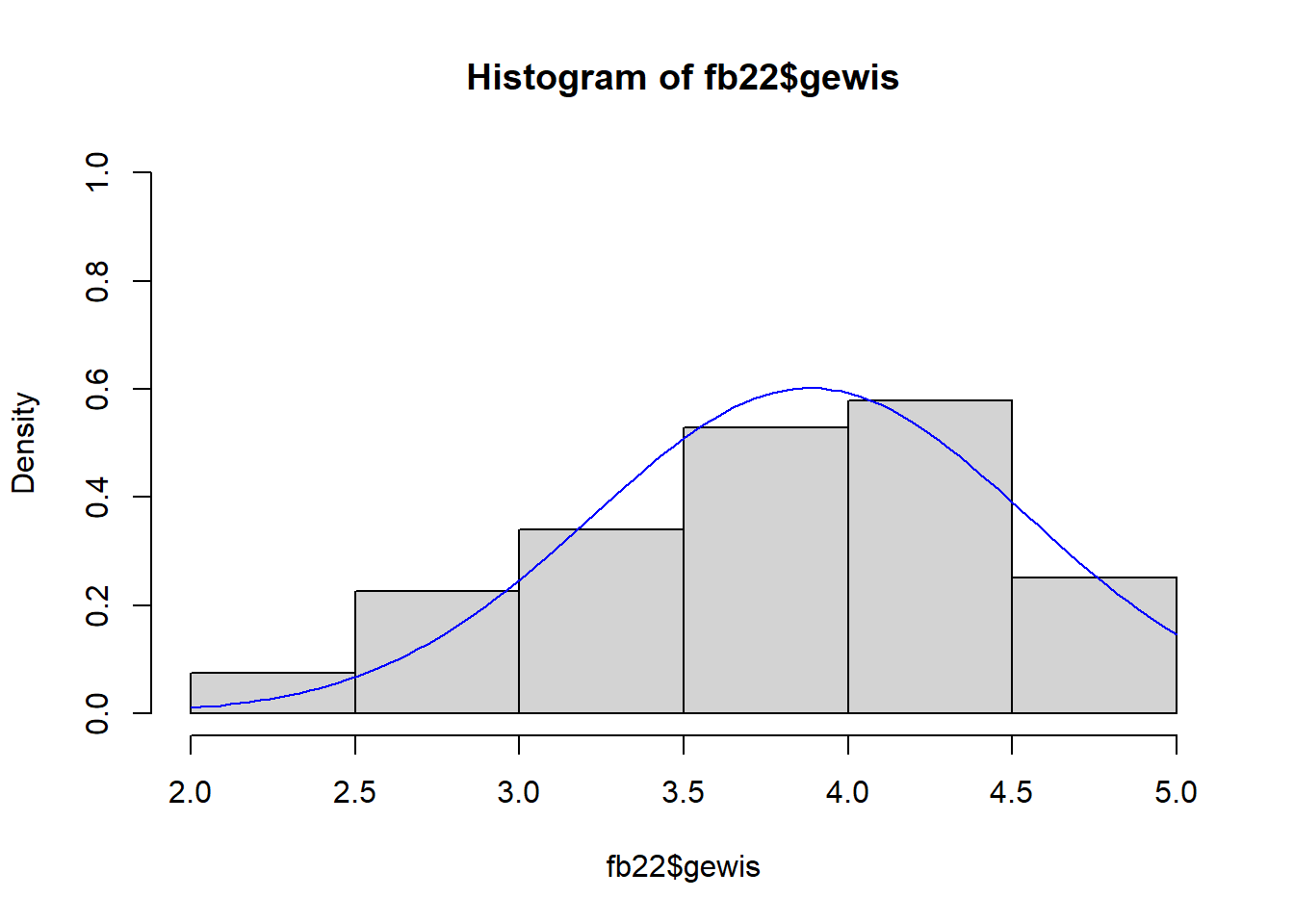
#Shapiro
shapiro.test(fb22$vertr)##
## Shapiro-Wilk normality test
##
## data: fb22$vertr
## W = 0.95611, p-value = 6.624e-05shapiro.test(fb22$gewis)##
## Shapiro-Wilk normality test
##
## data: fb22$gewis
## W = 0.95665, p-value = 7.423e-05\(p < \alpha\) \(\rightarrow\) H1: Normalverteilung kann nicht angenommen werden. Somit ist diese Voraussetzung verletzt. Eine Möglichkeit damit umzugehen, ist die Rangkorrelation nach Spearman. Diese ist nicht an die Voraussetzung der Normalverteilung gebunden. Das Verfahren kann über method = "spearman" angewendet werden.
Rangkorrelation in R
r1 <- cor(fb22$vertr,fb22$gewis,
method = "spearman", #Pearson ist default
use = "complete")
r1## [1] 0.2477728Interpretation des deskriptiven Zusammenhangs:
Es handelt sich um eine positive Korrelation von r = 0.25. Der Effekt ist nach Cohens (1988) Konvention als schwach bis mittelstark zu bewerten. Je höher die Ausprägung in Verträglichkeit, desto höher ist die Ausprägung in der Gewissenhaftigkeit und anders herum.
Cohens (1988) Konvention zur Interpretation von \(|r|\):
- ~ .10: schwacher Effekt
- ~ .30: mittlerer Effekt
- ~ .50: starker Effekt
Als weitere Variante der Rangkorrelation gibt es noch Kendalls \(\tau\). Diese kann man mit method = "kendall" angesprochen werden.
cor(fb22$vertr, fb22$gewis, use = 'complete', method = 'kendall')## [1] 0.1882259Die Interpretation erfolgt wie bei Spearman’s Rangkorrelation.
Signifikanztestung des Korrelationskoeffizienten:
Nachdem der Korrelationskoeffizient berechnet wurde, kann dieser noch auf Signifikanz geprüft werden. Dazu verwenden wir die cor.test()-Funktion.
- H0: \(\rho = 0\) \(\rightarrow\) es gibt keinen Zusammenhang zwischen Verträglichkeit und Gewissenhaftigkeit
- H1: \(\rho \neq 0\) \(\rightarrow\) es gibt einen Zusammenhang zwischen Verträglichkeit und Gewissenhaftigkeit
cor <- cor.test(fb22$vertr, fb22$gewis,
alternative = "two.sided",
method = "spearman", #Da Voraussetzungen für Pearson verletzt
use = "complete")## Warning in cor.test.default(fb22$vertr, fb22$gewis, alternative = "two.sided", :
## Kann exakten p-Wert bei Bindungen nicht berechnencor$p.value #Gibt den p-Wert aus## [1] 0.001638895Anmerkung: Bei der Rangkorrelation kann der exakte p-Wert nicht berechnet werden, da gebundene Ränge vorliegen. Das Ergebnis ist allerdings sehr eindeutig: \(p > \alpha\) \(\rightarrow\) H1. Die Korrelation ist mit einer Irrtumswahrscheinlichkeit von 5% signifikant von 0 verschieden. Daraus würde sich die folgende Interpretation ergeben:
Ergebnisinterpretation: Es wurde untersucht, ob Verträglichkeit und Gewissenhaftigkeit miteinander zusammenhängen. Der spearman-Korrelationskoeffizient beträgt 0.25 und ist statistisch signifikant (p = 0.002). Folglich wird die Nullhypothese hier verworfen: Verträglichkeit und Gewissenhaftigkeit weisen einen signifikanten Zusammenhang auf.
Modifikation
Wir haben in der Funktion cor.test() als Argument method = "spearman" eingegeben, da die Voraussetzungen für die Pearson-Korrelation nicht erfüllt waren. Wenn dies der Fall gewesen wäre, müsste man stattdessen method = "pearson" angeben:
cor.test(fb22$vertr, fb22$gewis,
alternative = "two.sided",
method = "pearson",
use = "complete")##
## Pearson's product-moment correlation
##
## data: fb22$vertr and fb22$gewis
## t = 2.57, df = 157, p-value = 0.0111
## alternative hypothesis: true correlation is not equal to 0
## 95 percent confidence interval:
## 0.04673796 0.34575779
## sample estimates:
## cor
## 0.2009235Vertiefung: Wie können Zusammenhangsmaße für ordinalskalierte Daten berechnet werden?
In diesem Abschnitt wird vertiefend die Bestimmung von Zusammenhangsmaßen für ordinalskalierte Variablen besprochen. Den dazugehörigen Auszug aus den Vorlesungsfolien, der in diesem Jahr herausgekürzt wurde, finden Sie hier.
Ordinalskalierte Daten können aufgrund der Verletzung der Äquidistanz zwischen bspw. Antwortstufen eines Items eines Messinstrumentes nicht schlicht mittels Pearson-Korrelation in Zusammenhang gesetzt werden. Zudem sind oft Verteilungsannahmen bei ordinalskalierten Variablen verletzt. Der Koeffizient \(\hat{\gamma}\) ist zur Betrachtung solcher Zusammenhänge am besten geeignet (sogar besser als Spearman’s und Kendalls’s Rangkorrelation). Er nimmt - ähnlich wie Spearman’s und Kendall’s Koeffizenten - weder eine gewisse Verteilung der Daten an, noch deren Äquidistanz.
Zur Berechnung dieses Koeffizienten müssen wir das Paket rococo installieren, welches verschiedene Konkordanz-basierte Zusammenhangsmaße enthält. Die Installation muss dem Laden des Paketes logischerweise vorausgestellt sein. Wenn R einmal geschlossen wird, müssen alle Zusatzpakete neu geladen, jedoch nicht neu installiert werden.
install.packages('rococo') #installierenlibrary(rococo) #laden## Warning: Paket 'rococo' wurde unter R Version 4.2.2 erstelltÜbersichten über Pakete kann man mit ?? erhalten.
??rococoDie Funktion heißt hier zufälligerweise genau gleich wie das Paket. Wenn man nur Informationen über die Funktion statt dem Paket sucht, geht das anhand von ?.
?rococoDank des neuen Pakets können wir nun den Koeffizienten \(\hat{\gamma}\) berechnen und damit den Zusammenhang zwischen Items betrachten. Schauen wir uns nun mal den Zusammenhang der beiden Prokrastinationsitems prok1 und prok9 an, um zu überprüfen, ob die beiden Items auch (wie beabsichtigt) etwas Ähnliches messen (nähmlich Prokrastionationstendenz). Die beiden Variablen wurden ursprünglich auf einer Skala von 1 (stimmt nicht) bis 4 (stimmt genau) (also auf Ordinalskalenniveau) erfasst.
rococo(fb22$prok1, fb22$prok9)## [1] 0.7466895Um zu überprüfen, ob zwei ordinalskalierte Variablen signifikant miteinander zusammenhängen, können wir die rococo.test()-Funktion anwenden.
rococo.test(fb22$prok1, fb22$prok9)##
## Robust Gamma Rank Correlation:
##
## data: fb22$prok1 and fb22$prok9 (length = 159)
## similarity: linear
## rx = 0.1 / ry = 0.2
## t-norm: min
## alternative hypothesis: true gamma is not equal to 0
## sample gamma = 0.7466895
## estimated p-value = 0.006 (6 of 1000 values)Betrachten wir nun den Koeffizienten \(\hat{\gamma}\) für zwei andere Items (prok1 mit prok2)
##
## Robust Gamma Rank Correlation:
##
## data: fb22$prok1 and fb22$prok2 (length = 159)
## similarity: linear
## rx = 0.1 / ry = 0.1
## t-norm: min
## alternative hypothesis: true gamma is not equal to 0
## sample gamma = -0.3303587
## estimated p-value = 0.006 (6 of 1000 values)Der Koeffizient von -0.33 zeigt uns, dass die Items zwar miteinander korrelieren, allerdings negativ. Ist hier etwas schief gelaufen? Nein, prok2 ist lediglich ein invertiertes Item. Mit der rekodierten Variante der prok2 Variable würde das - nicht da stehen, aber die Höhe der Korrelation gleich bleiben. Wir sehen daher, dass prok1 mit prok2 signifikant zusammenhängt. Die beiden Items messen demnach ein ähnliches zugrundeliegendes Konstrukt (Prokrastination).
