Lösungen
Vorwarnung
Achtung! Im Folgenden werden die Lösungen für das achte Projekt präsentiert. Falls du das Projekt noch nicht vollständig bearbeitet hast, nutze zunächst die Tipps. Sofern dir die Tipps für einen Teil nicht geholfen haben, kannst du die Lösungen dafür benutzen, einen Schritt weiterzukommen und beim nächsten Abschnitt weiterzumachen.
Abschnitt 1 - Daten herunterladen
Variable auswählen
Abschnitt anzeigen
Bevor du anfängst, lädst du am besten die Pakete, welche du für dieses Projekt benötigst:
#install.packages("httr")
#install.packages("jsonlite")
#install.packages('OData')
#install.packages('data.table')
library(data.table)
library(OData)
library(httr)
library(jsonlite)Im ersten Schritt musst du dir dann eine Variable aussuchen. Dafür lädst du zunächst die Daten der Indikatoren herunter.
Indikatoren <- retrieveData('https://ghoapi.azureedge.net/api/Indicator')Da wir uns die verschachtelte Liste nur schwer anzeigen lassen können, wandeln wir sie in ein Dataframe um.
Indi <- as.data.frame(Indikatoren)Somit bekommen wir ein sehr langes Dataframe, bei dem in der ersten Zeile der Variablenname steht, in der zweiten Zeile die Beschreibung zu der Variable und in der dritten Zeile die Sprache.
Wenn du dir die Indikatoren lieber als Matrix anzeigen lassen willst, musst du zunächst den Befehl unlist anwenden.
a <- unlist(Indikatoren)
ma <- as.matrix(a)Hier hast du nun eine Spalte, in der erst der Vairablenname steht, darunter steht die Beschreibung zu der Variable und wiederum darunter steht die Sprache zu der Variable, welche meistens Englisch ist.
Ich werde im Folgenden mit der VariableWHOSIS_000001 zur Lebenserwartung arbeiten, aber es sollte mit jeder anderen Variable genauso funktionieren.Daten über die API herunterladen
Abschnitt anzeigen
Die Daten, mit denen wir arbeiten wollen, können wir auf die gleiche Art und Weise herunterladen, wie die Indikatoren: mit dem Befehl retrieveData. Das Einzige, was wir für den Befehl benötigen, ist die richtige URL.
Für die Daten der WHO ist der grundlegende Befehl https://ghoapi.azureedge.net/api. Da ich die Variable mit den Daten zur Lebenserwartung laden möchte, füge ich dem Link ein / + WHOSIS_000001 an. Damit sieht der vollständige Link folgendermaßen aus: https://ghoapi.azureedge.net/api/WHOSIS_000001 und kann zum Laden der Daten verwendet werden.
data <- retrieveData('https://ghoapi.azureedge.net/api/WHOSIS_000001')Diese Daten liegen nun wieder in einer verschachtelten Liste vor. Wenn wir uns die Variable value anschauen, sehen wir jedoch, dass es sich diesmal nicht um character, sondern um weitere Listen in der Liste handelt. Für diese Art von verschachtelten Listen gibt es einen einfachen Befehl aus dem data.table-Paket, welches mit dem Befehl rbindlist verschachtelte Listen in einfache Dataframes konvertiert. Dabei musst du nur beachten, dass du fill= TRUE setzt.
data1 <- rbindlist(data$value, fill = T)Abschnitt 2 - Interaktive Grafiken
Übertragen der Daten von Excel in R Studio
Hier findest du eine kurze Erklärung, wie man Datensätze von Excel in R Studio überführt. Wenn du diesen Schritt erledigt hast, kehre zurück zu Abschnitt 2 dieses Projektes und probiere dich an der Erstellung von Grafiken mit plotly.
Abschnitt anzeigen
Bevor du mit dem Einlesen der Daten beginnen kannst, solltest du dir das Dokument anschauen. Es enthält 6 Tabellen: “Tabelle1” bis “Tabelle6”.
- Tabelle 1 ist leer.
- Tabelle 2 beinhaltet Variablennamen und deren Erläuterung
- Tabelle 3 führt alle Indikatoren und deren Bedeutung (z.B. WHOSIS_000001 = ‘Life expectancy at birth (years)’)
- Tabelle 4 stellt den gesamten Datensatz dar
- Tabelle 5 nur die Daten von Männern aus 2011 und
- Tabelle 6 nur die Daten von Männern
Das heißt, dass du hier ausschließlich Tabelle 4 in R laden solltest. Dafür kann man das Paket readxl mit dem Befehl read_excel verwenden. Mit den Argumenten innerhalb dieses Befehls musst du dann nur noch den Dateipfad der Excel-Tabellen, die Behandlung von fehlenden Werten und den Namen des gewünschten Sheets (“Tabelle4”) angeben und das Ergebnis einem Objekt zuweisen. Achte hierbei darauf, dass sich das Excel-Dokument in deiner Working Directory befindet.
#install.packages("readxl")
library (readxl)
data1 <- read_excel("GHO-Daten.xlsx", na = "NA", sheet = "Tabelle4")Anpassung des Datensatzes
Hier wird der geladene Datensatz von unbrauchbaren Spalten bereinigt, die Variablen neu benannt und zwei neue Variablen hinzugefügt. Prinzipiell kannst du dadurch den Datensatz übersichtlicher machen aber auch die Struktur der Daten kennenlernen.
Abschnitt anzeigen
Natürlich kann man sich mithilfe der Standard-Pakete von R bereits einen guten Überblick über die Struktur des Datensatzes verschaffen. So könnten beispielsweise folgende Befehle verwendet werden:
str(data1) # gibt den Typ jeder Variable aus ("character", "string", "integer", "numeric")
table(data1$IndicatorCode) # Tabellen zu den Ausprägungen der gewählten Variablen
table(data1$SpatialDimType) # ermöglicht Überblick über Anzahl der Ausprägungen und deren Häufigkeit
table(data1$TimeDimType)
table(data1$TimeDim)
View(data1) # öffnet den gesamten Datensatz in einem neuen FensterWir haben uns im ersten Schritt jedoch dafür entschieden, mit dem tidyverse-Paket zu arbeiten. Dieses bietet einige Vorteile im Umgang mit Datensätzen. So kann man den Datensatz in ein neues Format (tibble) umwandeln, das die Darstellung des Datensatzes standardmäßig übersichtlicher gestaltet.
#install.packages("tidyverse")
library(tidyverse)## ── Attaching packages ────────────────────────────────────────────────────── tidyverse 1.3.0 ──## ✓ ggplot2 3.3.2 ✓ purrr 0.3.4
## ✓ tibble 3.0.3 ✓ dplyr 1.0.2
## ✓ tidyr 1.1.2 ✓ stringr 1.4.0
## ✓ readr 1.3.1 ✓ forcats 0.5.0## ── Conflicts ───────────────────────────────────────────────────────── tidyverse_conflicts() ──
## x dplyr::between() masks data.table::between()
## x dplyr::filter() masks stats::filter()
## x dplyr::first() masks data.table::first()
## x purrr::flatten() masks jsonlite::flatten()
## x dplyr::lag() masks stats::lag()
## x dplyr::last() masks data.table::last()
## x purrr::transpose() masks data.table::transpose()data1 <- as_tibble(data1)
data1## # A tibble: 9,486 x 23
## Id IndicatorCode SpatialDimType SpatialDim TimeDimType TimeDim Dim1Type
## <dbl> <chr> <chr> <chr> <chr> <dbl> <chr>
## 1 1.56e7 WHOSIS_000001 COUNTRY RWA YEAR 2000 SEX
## 2 1.56e7 WHOSIS_000001 COUNTRY RWA YEAR 2000 SEX
## 3 1.56e7 WHOSIS_000001 COUNTRY RWA YEAR 2000 SEX
## 4 1.56e7 WHOSIS_000001 COUNTRY RWA YEAR 2001 SEX
## 5 1.56e7 WHOSIS_000001 COUNTRY RWA YEAR 2001 SEX
## 6 1.56e7 WHOSIS_000001 COUNTRY RWA YEAR 2001 SEX
## 7 1.56e7 WHOSIS_000001 COUNTRY RWA YEAR 2002 SEX
## 8 1.56e7 WHOSIS_000001 COUNTRY RWA YEAR 2002 SEX
## 9 1.56e7 WHOSIS_000001 COUNTRY RWA YEAR 2002 SEX
## 10 1.56e7 WHOSIS_000001 COUNTRY RWA YEAR 2003 SEX
## # … with 9,476 more rows, and 16 more variables: Dim1 <chr>, Dim2Type <lgl>,
## # Dim2 <lgl>, Dim3Type <lgl>, Dim3 <lgl>, DataSourceDimType <lgl>,
## # DataSourceDim <lgl>, Value <chr>, NumericValue <dbl>, Low <lgl>,
## # High <lgl>, Comments <chr>, Date <dttm>, TimeDimensionValue <chr>,
## # TimeDimensionBegin <dttm>, TimeDimensionEnd <dttm>Jetzt können wir mit einem Befehl und ohne Zwischenschritte direkt alle Spalten entfernen, die nicht weiter von Nutzen sind. Das ermöglicht der Pipe-Befehl %>% aus dem dplyr-Paket. %>% umgeht Zwischenschritte, indem es die zuvor erstellten “Zwischenwerte” direkt als erstes Argument in die darauf folgende Funktion einsetzt. Hier wird bspw. zuerst das Objekt data1 ausgewählt; in der folgenden Funktion select_if wird data1 als erstes Argument eingesetzt und modifiziert; das Ergebnis davon wird schließlich in der letzten Funktion (select) weiter verwertet und abschließend verändert. (Für die Bedeutung und Funktionsweise der einzelnen Funktionen hierin, nutze die R interne Hilfe oder das Internet.)
data2 <- data1 %>%
select_if(function(col) !is.logical(col)) %>%
select(-c(TimeDimType, Dim1Type, Date, TimeDimensionValue, TimeDimensionBegin, TimeDimensionEnd))
data2## # A tibble: 9,486 x 9
## Id IndicatorCode SpatialDimType SpatialDim TimeDim Dim1 Value
## <dbl> <chr> <chr> <chr> <dbl> <chr> <chr>
## 1 1.56e7 WHOSIS_000001 COUNTRY RWA 2000 MLE 43.0
## 2 1.56e7 WHOSIS_000001 COUNTRY RWA 2000 FMLE 48.4
## 3 1.56e7 WHOSIS_000001 COUNTRY RWA 2000 BTSX 45.7
## 4 1.56e7 WHOSIS_000001 COUNTRY RWA 2001 MLE 43.5
## 5 1.56e7 WHOSIS_000001 COUNTRY RWA 2001 FMLE 49.5
## 6 1.56e7 WHOSIS_000001 COUNTRY RWA 2001 BTSX 46.5
## 7 1.56e7 WHOSIS_000001 COUNTRY RWA 2002 MLE 46.0
## 8 1.56e7 WHOSIS_000001 COUNTRY RWA 2002 FMLE 51.5
## 9 1.56e7 WHOSIS_000001 COUNTRY RWA 2002 BTSX 48.8
## 10 1.56e7 WHOSIS_000001 COUNTRY RWA 2003 MLE 47.4
## # … with 9,476 more rows, and 2 more variables: NumericValue <dbl>,
## # Comments <chr>Nun hast du den Datensatz erfolgreich auf 9 Spalten reduziert. Diese kannst du zur besseren Übersicht umbenennen:
names(data2) <- c('DatID', 'VarID', 'SpatialDimType', 'COUNTRYCODE', 'YEAR',
'SEX', 'LIFE_EXPECTANCY','L_E2EXACT', 'COMMENT')Dir ist vielleicht aufgefallen, dass zwei Variablen die Lebenserwartung beinhalten: LIFE_EXPECTANCY und L_E2EXACT. Aus diesem Grund lohnt sich ein genauerer Blick auf beide Variablen - Kann man beide für ein Diagramm verwenden oder ist irgendeine Variable besser/schlechter geeignet oder gar ungeeignet? Ein Blick auf die Variablenart könnte darüber Aufschluss liefern:
str(data2$LIFE_EXPECTANCY)## chr [1:9486] "43.0" "48.4" "45.7" "43.5" "49.5" "46.5" "46.0" "51.5" ...str(data2$L_E2EXACT)## num [1:9486] 43 48.4 45.7 43.5 49.5 ...range(data2$L_E2EXACT)## [1] 33.22747 87.14502Wie sich zeigt, ist die Variable LIFE_EXPECTANCY ein character. Das heißt, dass R diese Variable nicht als Zahl behandelt, weshalb diese für Abbildungen ungeeignet ist. L_E2EXACT hingegen liegt im Format numeric vor. Im weiteren Verlauf müssen wir deshalb diese Variable zur Erstellung der Abbildungen verwenden.
Jetzt kannst du dich daran setzen, die zwei neuen Variablen zu erstellen. Im aktuellen Zustand stehen unter der Variable COUNTRYCODE nur die Codes der verschiedenen Länder. Diese sind jedoch teilweise nicht intuitiv zuzuordnen, weshalb wir noch eine neue Variable (COUNTRY) erstellen, die die ausgeschriebenen Ländernamen beinhaltet. Glücklicherweise sind die Ländercodes weit verbreitet und können deshalb einfach mit dem ISOcodes-Paket in ihre zugehörigen Ländernamen umgewandelt werden.
#install.packages("ISOcodes")
library(ISOcodes)(Anmerkung: Da die for-Schleife einen Fehler hervorruft, können wir die folgenden Befehle an dieser Stelle nicht ausführen. Wenn du das selbst in deiner R-Sitzung ausführst, wirst du merken, dass die neue Variable COUNTRY trotz des Fehlers vollständig mit Werten befüllt wurde. Hiernach gilt es für dich, die Gründe für den Fehler zu ermitteln. Dabei helfen die darauf folgenden 4 Befehle.)
for (i in 1:9438){
data2$COUNTRY[i] <- ISO_3166_1$Name[ISO_3166_1$Alpha_3 == data2$COUNTRYCODE[i]]
}
table(table(data2$COUNTRY)) #Warum gibt es zu allen Ländern 51 Einträge und zu einem 204?
#A: Welches Land hat 204 Einträge? --> Ruanda
data2 %>% .$COUNTRY %>% table() %>% which.max()
#B: Woran liegt das? Ein Fehler? --> JA!
data2 %>% filter(COUNTRY == "Rwanda") %>% select(COUNTRYCODE) %>% table()
#C: Wie viele Daten gibt es wirkich zu Ruanda?
data2 %>% filter(COUNTRYCODE == "RWA") %>% select(COUNTRY) %>% table()Wie sich zeigt, ist bei der Umwandlung der Ländercodes ein Fehler aufgetreten - und ab diesem Punkt im Datensatz wurde in jeder Zeile “der erste Wert” eingesetzt (siehe A). Schaut man sich die falsch formatierten Ländercodes (bzw. die zugehörigen Zeilen) an, so fällt auf, dass es sich hierbei um selbstkreierte Regionen der WHO handelt (siehe B). Zudem war der erste Wert aus irgendeinem Grund Ruanda, obwohl die Länder ansonsten alphabetisch geordnet vorliegen. Es zeigt sich, dass zu Ruanda mehr Daten zur Verfügung stehen, als zu allen anderen Ländern (normal: 17 Jahre * 3 Geschlechter = 51 Datenpunkte)(siehe C).
Daraus lässt sich schließen, dass die ersten 48 Zeilen zu Ruanda irgendwie fehl am Platz sind und gelöscht werden können. Außerdem müssen die Ländercodes zu den WHO-Regionen händisch umgewandelt werden:
data2 <- data2[-c(1:48), ] #um die ersten Ruanda-Zeilen zu löschen
for (i in 1:9438){
if (data2$COUNTRYCODE[i] == "AFR"){data2$COUNTRY[i] <- "African Region"}
else if (data2$COUNTRYCODE[i] == "AMR"){data2$COUNTRY[i] <- "Region of the Americas"}
else if (data2$COUNTRYCODE[i] == "EMR"){data2$COUNTRY[i] <- "Eastern Mediterranean Region"}
else if (data2$COUNTRYCODE[i] == "EUR"){data2$COUNTRY[i] <- "European Region"}
else if (data2$COUNTRYCODE[i] == "GLOBAL"){data2$COUNTRY[i] <- "GLOBAL"}
else if (data2$COUNTRYCODE[i] == "SEAR"){data2$COUNTRY[i] <- "South-East Asian Region"}
else if (data2$COUNTRYCODE[i] == "WPR"){data2$COUNTRY[i] <- "Western Pacific Region"}
else {data2$COUNTRY[i] <- ISO_3166_1$Name[ISO_3166_1$Alpha_3 == data2$COUNTRYCODE[i]]}
}
table(table(data2$COUNTRY))##
## 15 51
## 7 183Wie die Tabelle zeigt, wurden die Ländercodes in COUNTRYCODE nun erfolgreich in die vollständigen Länder-/Regionenbezeichnungen in der Variable COUNTRY umgewandelt. Das sieht man daran, dass nun zu jedem Land 51 Datenpunkte vorliegen und zu allen Regionen 15.
Jetzt können wir uns der zweiten neuen Variable widmen: den Kontinenten. Auch hierfür gibt es ein passendes Paket in R, das anhand der Länderbezeichnungen (COUNTRY) den Kontinent ausgeben kann. Dieses Paket heißt genau wie die benötigte Funktion countrycode.
#install.packages('countrycode')
library(countrycode)
data2 <- data2 %>% mutate(
CONTINENT = countrycode(
sourcevar = data2$COUNTRY,
origin = "country.name",
destination = "continent",
nomatch = NA))Zur weiteren Überprüfung kann man nun noch folgende Befehle ausführen:
table(data2$CONTINENT) #Gibt Auskunft darüber, wie häufig welcher Kontinent eingefügt wurde##
## Africa Americas Asia Europe Oceania
## 2754 1683 2397 1989 510table(data2$COUNTRY[data2$CONTINENT == "Oceania"]) #Zeigt alle Länder vom Kontinent Ozeanien an##
## Australia Fiji
## 51 51
## Kiribati Micronesia, Federated States of
## 51 51
## New Zealand Papua New Guinea
## 51 51
## Samoa Solomon Islands
## 51 51
## Tonga Vanuatu
## 51 51table(data2$CONTINENT)/51 #So erhält man die Anzahl der Länder pro Kontinent, zu denen Daten der WHO vorliegen##
## Africa Americas Asia Europe Oceania
## 54 33 47 39 10Somit ist dieser Abschnitt abgeschlossen. Der Datensatz wurde erfolgreich von unnötigen Zeilen und Spalten bereinigt, die Variablen wurden neu benannt und die zwei neuen Variablen wurden erstellt. Hiernach solltest du außerdem einen weitreichenden Überblick über den Datensatz haben, ohne ihn dir Zeile für Zeile anschauen zu müssen.
Grafiken erstellen mit plotly
Jetzt, wo die Daten geladen und vorbereitet wurden, können wir uns dem Erstellen der Grafiken widmen. In der Problemstellung haben wir hierfür fünf verschiedene Vorschläge gegeben. Diese steigern sich in ihrer Komplexität und werden im Folgenden nach und nach durchgegangen. Grafik 1 soll hierbei eine generelle Einführung in die Struktur von plotly-Abbildungen darstellen und die Möglichkeit aufzeigen, Abbildungen auf Basis von ggplot2 nur mit einer Funktion (ggplotly) in plotly-Abbildungen umzuwandeln.
Lade dir dafür zunächst einmal das plotly-Paket:
#install.packages("plotly")
library(plotly)Grafik 1 - Ein Standard-Plot
Das Ziel des ersten Plots ist es, ein Liniendiagramm mit drei Linien zu erstellen. Diese drei Linien sollen dabei die drei Ausprägungen der Gendervariable repräsentieren (MLE, FMLE und BTSX). Das bedeutet im Umkehrschluss, dass wir den Datensatz auf ein Land reduzieren müssen. Hierfür verwenden wir weiterhin dplyr-Funktionen aus dem tidyverse:
filter(data2, COUNTRY == "Germany")## # A tibble: 51 x 11
## DatID VarID SpatialDimType COUNTRYCODE YEAR SEX LIFE_EXPECTANCY L_E2EXACT
## <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> <dbl>
## 1 1.82e7 WHOS… COUNTRY DEU 2000 MLE 75.0 75.0
## 2 1.82e7 WHOS… COUNTRY DEU 2001 MLE 75.4 75.4
## 3 1.82e7 WHOS… COUNTRY DEU 2002 MLE 75.6 75.6
## 4 1.82e7 WHOS… COUNTRY DEU 2003 MLE 75.7 75.7
## 5 1.82e7 WHOS… COUNTRY DEU 2004 MLE 76.4 76.4
## 6 1.82e7 WHOS… COUNTRY DEU 2005 MLE 76.5 76.5
## 7 1.82e7 WHOS… COUNTRY DEU 2006 MLE 76.9 76.9
## 8 1.82e7 WHOS… COUNTRY DEU 2007 MLE 77.1 77.1
## 9 1.82e7 WHOS… COUNTRY DEU 2008 MLE 77.3 77.3
## 10 1.82e7 WHOS… COUNTRY DEU 2009 MLE 77.4 77.4
## # … with 41 more rows, and 3 more variables: COMMENT <chr>, COUNTRY <chr>,
## # CONTINENT <chr>Wie man sehen kann, wurde der Datensatz nun auf 51 Zeilen reduziert. Alle diese Zeilen enthalten Daten aus Deutschland. Um mit diesem modifizierten Datensatz weiterarbeiten zu können, müssen wir diesen jedoch NICHT in einem neuen Objekt speichern. Hier können wir einfach wieder den Pipe-Operator %>% verwenden. Zur Erstellung der Abbildung verwendet man nun den plot_ly-Befehl (siehe ?plot_ly für weitere Hilfe). Ohne weitere Anpassungen sieht der Code dann folgendermaßen aus:
filter(data2, COUNTRY == "Germany") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "lines+markers")## Warning: `arrange_()` is deprecated as of dplyr 0.7.0.
## Please use `arrange()` instead.
## See vignette('programming') for more help
## This warning is displayed once every 8 hours.
## Call `lifecycle::last_warnings()` to see where this warning was generated.Wenn man möchte kann man hier jetzt noch mit dem colors-Argument die Farben der drei Linien anpassen. Das kann man bspw. manuell machen, indem man einfach drei Farben nennt. In diesem Fall ist das auch noch nicht zu aufwändig - bei mehr als 10 benötigten Farben könnte das jedoch problematisch werden. In solchen Fällen kann man beispielsweise die colorRampPalette-Funktion verwenden. Diese Funktion funktioniert derart, dass man 2 oder mehr Farben angibt, aus denen die Funktion dann einen Farbverlauf bildet (diese Funktion speichert man in einem Objekt ab, bspw. CRP). Aus diesem Farbverlauf kann man dann eine beliebige Anzahl an Farbabstufungen ziehen, indem man hinter dem Farbobjekt (CRP) in Klammern angibt, wie viele Farben benötigt werden. Das könnte jetzt erst einmal etwas verwirrend sein, deshalb hier ein Beispiel:
CRP <- colorRampPalette(c('red', 'blue'))
filter(data2, COUNTRY == "Germany") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "lines+markers",
colors = CRP(3))Das Objekt bzw. die Funktion CRP() hat in diesem Fall dafür gesorgt, dass aus einem Farbverlauf von rot und blau eine dritte Farbe gezogen wurde. Diese eine Farbe liegt ‘genau zwischen rot und blau’ und ist somit eine 50:50-Mischung aus diesen beiden Farben - deshalb ist die dritte Farbe violett.
Eine weitere Möglichkeit zur Erstellung dieses Plots ist das ggplot2-Paket. Die daraus entstandene Abbildung kann dann mit der ggplotly-Funktion direkt in eine plotly-Abbildung umgewandelt werden. Dafür bedarf es keiner weiteren Pakete, da ggplot2 bereits im tidyverse enthalten ist. Ein ähnliches Liniendiagramm mit ggplot2 ließe sich demnach folgendermaßen erstellen:
#install.packages("ggthemes")
library(ggthemes) # aus diesem Paket kommt das Color-Theme `theme_stata`
ggplot1 <- filter(data2, COUNTRY == "Germany") %>%
ggplot(aes(x = YEAR, y = L_E2EXACT, group = SEX)) +
geom_line(aes(colour = SEX)) + # Liniendiagramm
xlab('TIME') + # Beschriftung x-Achse
ylab('LIFE EXPECTANCY') + # Beschriftung y-Achse
ggtitle('LIFE EXPECTANCY DEVELOPMENT IN GERMANY - GENDER COMPARISON') + # Überschrift
scale_color_manual(values = CRP(3)) + # Zuweisung Farbe-Geschlecht
theme_stata()
ggplot1 # Abruf des gespeicherten Plots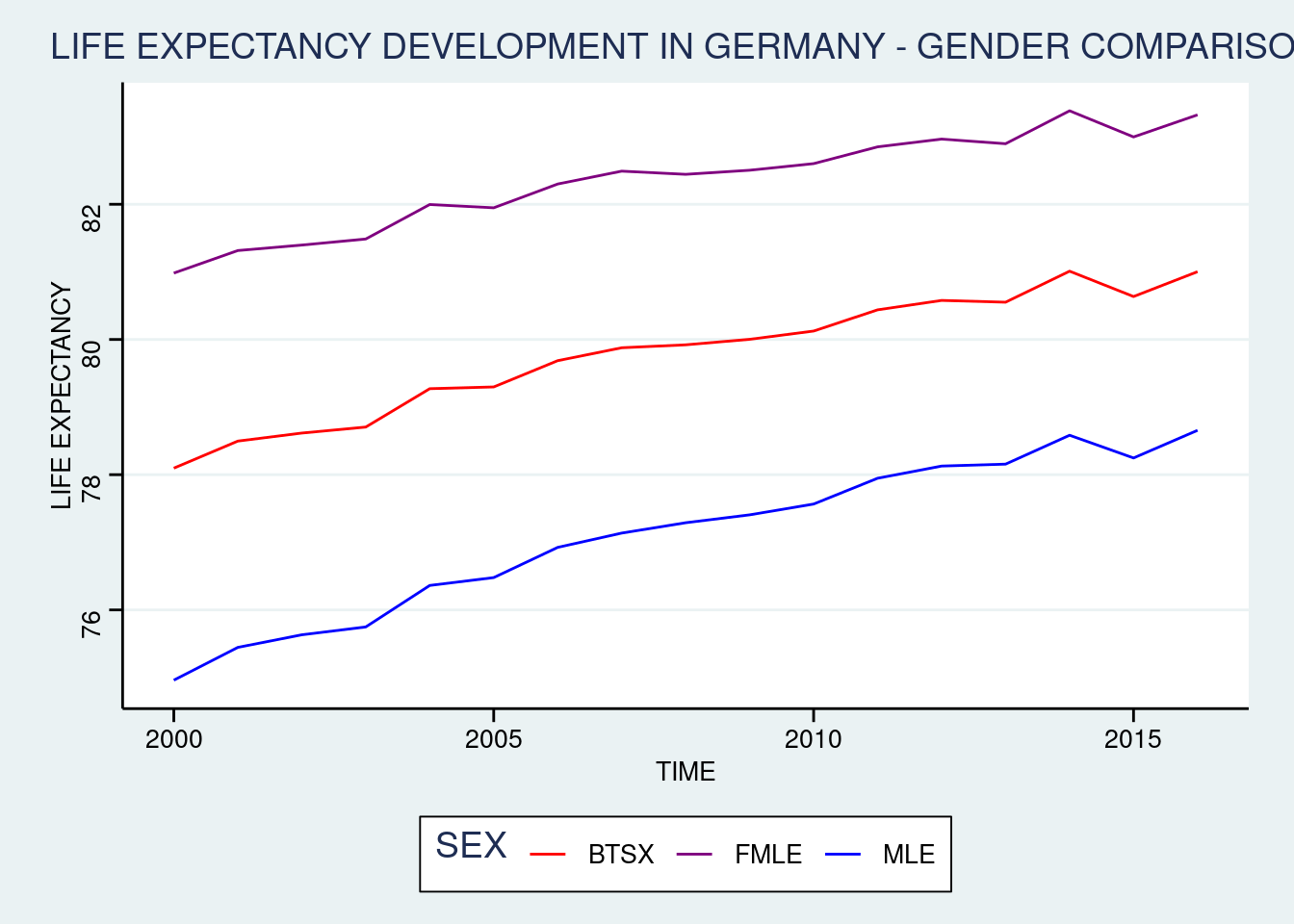
Die Umwandlung in eine plotly-Abbildung geschieht dann einfach folgendermaßen:
ggplotly(ggplot1)## Warning: `group_by_()` is deprecated as of dplyr 0.7.0.
## Please use `group_by()` instead.
## See vignette('programming') for more help
## This warning is displayed once every 8 hours.
## Call `lifecycle::last_warnings()` to see where this warning was generated.Wie man sehen kann, ergibt sich ein sehr ähnlicher Plot - einzig und allein mit dem Unterschied, dass der ggplot bereits an einigen Stellen modifiziert wurde (bspw. xlab('TIME') für die x-Achsenbeschriftung und theme_stata() für das Aussehen von Achsen, Hintergrund und Gitterlinien). Die Frage ist: Geht das auch direkt mit plotly oder macht es sogar mehr Sinn den Weg über ggplot2 zu gehen?
Diese Frage ist nicht pauschal zu beantworten. Für einige simplere Plots sind die Ergebnisse von ggplot2 und plotly kaum unterscheidbar, weshalb in diesem Fall einfach die Empfehlung lautet: Nutze das Paket, das dir besser liegt. Doch an einigen Stellen und Typen von Plots stößt ggplot2 in der Kombination mit den Funktionen von plotly an seine Grenzen (dazu mehr im Abschnitt zum zweiten Plot ).
Nun stellt sich noch die Frage, wie man die angesprochenen Formatierungen mit plotly umsetzt. Hierfür gibt es einen einfachen Befehl: layout, mit dem alle graphischen Anpassungen getätigt werden können. Auch hier bietet es sich an, mit %>% die erstellte Abbildung direkt in die layout-Funktion zu überführen. Im folgenden werden wir dir beispielhaft die Formatierung von theme_stata mit plotly nachempfinden - für eine breite Auswahl an Formatierungsmöglichkeiten kannst du dich auf dieser Website über alle Layout-Anpassungen informieren. Hinter den einzelnen Zeilen findest du jeweils kurz erklärt, was genau diese bewirken.
filter(data2, COUNTRY == "Germany") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "lines+markers",
colors = CRP(3)) %>%
layout(
# Hier bestimmen wir die Hintergrundfarbe des gesamten Papiers - mit `plot_bgcolor` kann man weiterhin auch die Hintergrundfarbe des Plots bestimmen
paper_bgcolor = "#eaf2f3",
# Mit dem Argument `legend` passen wir die Legende an - das geschieht in einer Liste (`list()`)
legend = list(
font = list( # Anpassung der Schrift der Legende
family = "sans-serif", # Schriftfamilie
size = 12, # Schriftgröße
color = "#000"), # Schriftfarbe
bgcolor = "#E2E2E2", # Hintergrundfarbe der Legende
bordercolor = "#0a0a0a", # Rahmenfarbe der Legende
borderwidth = 2, # Dicke des Rahmens der Legende
yanchor = "center", # Anker für y-Wert in nächster Zeile
y = 0.5), # Position der Legende auf der y-Achse (Bedeutung abhängig von Ankersetzung)
# Mit `annotations` kann man Text in die Abbildung einfügen.
annotations = list(
text = "GERMANY", # dieser Text soll eingefügt werden
font = list( # Hier wird wiederum die Schrift angepasst
family = "Arial, Helvetica, sans-serif", # Schriftfamilie
size = 18, # Schriftgröße
color = "black"), # Schriftfarbe
xref = "paper", # Positionierung auf der x-Achse
xanchor = "center",
x = 0.5,
yref = "paper", # Positionierung auf der y-Achse
yanchor = "bottom",
align = "center",
y = 0.95,
showarrow = FALSE # soll der Text mit einem Pfeil abgedruckt werden - default:TRUE
),
# Mit `xaxis` kann man Anpassungen an der x-Achse vornehmen.
xaxis = list(
title = "Time (in Years)", # Titel der x-Achse
gridcolor = "white", # Farbe der Gitterlinien der x-Achse (vertikale Gitterlinien)
gridwidth = 0, # Dicke der Gitterlinien der x-Achse (vertikale Gitterlinien)
tickcolor = "black", # Farbe der Ticks an der x-Achse (Striche zu der Skalierung)
tickwidth = 1, # Dicke der Ticks an der x-Achse (Striche zu der Skalierung)
linecolor = "black", # Farbe der Linie der x-Achse (an dieser Linie ist die Beschriftung)
linewidth = 1 # Dicke der Linie der x-Achse (an dieser Linie ist die Beschriftung)
),
# Mit `yaxis` kann man Anpassungen an der y-Achse vornehmen. (siehe `xaxis`)
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)Grafik 2 - Ein Plot mit veränderter
Dieser zweite Plot unterscheidet sich grundsätzlich kaum vom ersten. Aus diesem Grund können wir das Grundgerüst erst einmal übernehmen:hover-Info
filter(data2, COUNTRY == "Germany") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "lines+markers",
colors = CRP(3))An diesem Grundgerüst müssen wir nun an drei Stellen Veränderungen vornehmen:
- Die zugrundeliegenden Daten beschränken sich nun nicht nur auf ein Land, sondern auf einen Kontinent UND eine Ausprägung auf der Gendervariable. Das heißt, dass die Filter-Bedingung in der ersten Zeile angepasst werden muss.
- Die Farbe der einzelnen Linien hängt außerdem nicht mehr von der Variable
SEXab. Stattdessen soll in diesem Liniendiagramm jedes Land (COUNTRY) durch eine andere Linie repräsentiert werden. - In Folge dessen muss auch die Anzahl der Farben verändert werden, die aus dem Farbverlauf
CRP()gezogen werden. Hier muss stattdessen die Anzahl der in den WHO-Daten erfassten Ländern des ausgewählten Kontinents verwendet werden.
Anmerkung: Wir haben hier eine andere vorgefertigte Farbpalette verwendet, da jene aus rot und blau kaum Differenzierungen ermöglicht.
filter(data2, SEX == "BTSX" & CONTINENT == "Europe") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
colors = viridisLite::magma(nrow(filter(data2, CONTINENT == "Europe"))/51))Jetzt stellt sich noch die Frage: Wie passe ich hier die hover-Info an? Momentan sieht diese folgendermaßen aus:
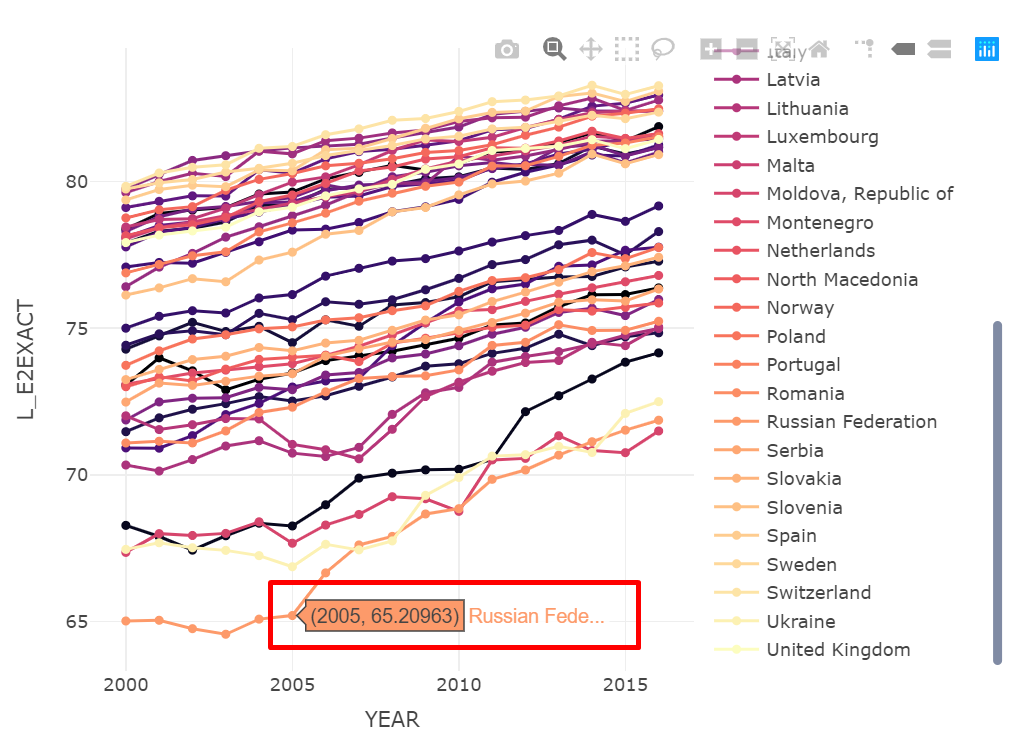
Das ist jedoch noch nicht optimal und Bedarf einiger Bearbeitung, denn zum einen liefert diese Info nicht den maximal möglichen Informationsgehalt und zum anderen sieht die Info auch optisch nicht sehr ansprechend aus. Mit ggplotly stößt man dabei an Grenzen - zumindest haben wir keinen Weg gefunden, die hover-Info hier verlässlich zu modifizieren.
Mit plotly direkt ist dies jedoch relativ leicht und kann auf mehrere Wege bewerkstelligt werden. Man kann beispielsweise die beiden Argumente text und hoverinfo verwenden. Hinter text fügt man dann einfach in paste0() den gewünschten hover-Infotext mit Formatierungen ein und macht dann hoverinfo = "text", um diesen Text als hoverinfo einzufügen. Das kann dann beispielsweise so aussehen:
filter(data2, SEX == "BTSX" & CONTINENT == "Europe") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$YEAR,
"<br><b>Sex:</b> ", filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$SEX,
"<br><b>Continent:</b> ", filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$CONTINENT),
hoverinfo = "text",
colors = viridisLite::magma(nrow(filter(data2, CONTINENT == "Europe"))/51)) %>%
layout(
paper_bgcolor = "#eaf2f3",
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center",
y = 0.5),
annotations = list(
text = "EUROPE",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.95,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)Damit ist auch der zweite Plot erfolgreich erstellt. Es sollte deutlich werden, dass die Möglichkeit, plotly und ggplot2 zu verbinden, sehr nützlich sein kann, jedoch mit Einschränkungen verbunden ist - insbesondere bezogen auf durch plotly gegebene Funktionen wie die hoverinfo. Aus diesem Grund werden alle Abbildungen in unseren Lösungen im Folgenden direkt mit plotly erstellt.
Grafik 3 - Mehrere Plots
Die dritte Abbildung soll dazu dienen, dass du dich mit der Möglichkeit auseinandersetzt, mehrere Plots in einer Abbildung darzustellen. Damit kann man beispielsweise mehrere Gruppen von Daten miteinander vergleichen. Um das umzusetzen, braucht man nur die subplot-Funktion, die einfach mehrere plotly-Abbildungen als Argumente annimmt und dann zusammenfügt. In der Problemstellung wurden dafür zwei mögliche Beispiele genannt:
- ein Gendervergleich mit mehreren Ländern in einem Plot (Bsp.: Europa)
- ein Ländervergleich mit allen drei Genderbezeichnungen in einem Plot
Dafür erstellt man zunächst jeden Plot einzeln und speichert diese jeweils in einem Objekt ab, um diese Objekte in der subplot-Funktion wieder verwenden zu können. Erstellen wir also zunächst einmal einen der Plots für Beispiel 1 - diesen können wir zufälligerweise einfach aus dem Abschnitt ‘Grafik 2’ übernehmen (nur Anpassung der annotation: In diesem Vergleich sind alle Datem aus Europa. Diese Info kann also eine allgemeine Überschrift auf der Ebene der subplot-Funktion liefern. Stattdessen ist hier für den einzelnen Plot die Ausprägung der Gendervariable von Interesse):
p1 <- filter(data2, SEX == "BTSX" & CONTINENT == "Europe") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$YEAR,
"<br><b>Sex:</b> ", filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$SEX,
"<br><b>Continent:</b> ", filter(data2, SEX == "BTSX" & CONTINENT == "Europe")$CONTINENT),
hoverinfo = "text",
colors = viridisLite::magma(nrow(filter(data2, CONTINENT == "Europe"))/51)) %>%
layout(
paper_bgcolor = "#eaf2f3",
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center",
y = 0.5),
annotations = list(
text = "Both Sexes",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.95,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p1Somit haben wir den ersten Plot (p1) zu der Ausprägung BTSX (= Both Sexes) bereits erstellt. Für p2 und p3 müssen wir nun einfach den Filter in der filter-Funktion und der hoverinfo verändern und die annotation auf den Gendervergleich anpassen. Ist das getan, sind die einzelnen Plots fertig und wir können uns mit der subplot-Funktion befassen. Da das Erstellen der einzelnen Plots an dieser Stelle sehr repetitiv wäre, werden wir dir den Code dazu nicht zwangsläufig präsentieren. Falls du Probleme hast oder die genauen Anpassungen anschauen möchtest, kannst du einfach den folgenden Unterabschnitt ausklappen.
Erstellen von
p2 und p3
p2: SEX = “MLE”
p2 <- filter(data2, SEX == "MLE" & CONTINENT == "Europe") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, SEX == "MLE" & CONTINENT == "Europe")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, SEX == "MLE" & CONTINENT == "Europe")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, SEX == "MLE" & CONTINENT == "Europe")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, SEX == "MLE" & CONTINENT == "Europe")$YEAR,
"<br><b>Sex:</b> ", filter(data2, SEX == "MLE" & CONTINENT == "Europe")$SEX,
"<br><b>Continent:</b> ", filter(data2, SEX == "MLE" & CONTINENT == "Europe")$CONTINENT),
hoverinfo = "text",
colors = viridisLite::magma(nrow(filter(data2, CONTINENT == "Europe"))/51)) %>%
layout(
paper_bgcolor = "#eaf2f3",
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center",
y = 0.5),
annotations = list(
text = "Male",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.95,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p2p3: SEX = “FMLE”
p3 <- filter(data2, SEX == "FMLE" & CONTINENT == "Europe") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, SEX == "FMLE" & CONTINENT == "Europe")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, SEX == "FMLE" & CONTINENT == "Europe")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, SEX == "FMLE" & CONTINENT == "Europe")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, SEX == "FMLE" & CONTINENT == "Europe")$YEAR,
"<br><b>Sex:</b> ", filter(data2, SEX == "FMLE" & CONTINENT == "Europe")$SEX,
"<br><b>Continent:</b> ", filter(data2, SEX == "FMLE" & CONTINENT == "Europe")$CONTINENT),
hoverinfo = "text",
colors = viridisLite::magma(nrow(filter(data2, CONTINENT == "Europe"))/51)) %>%
layout(
paper_bgcolor = "#eaf2f3",
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center",
y = 0.5),
annotations = list(
text = "Female",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.95,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p3Nun geht es darum, diese drei Plots mit dem subplot-Befehl in eine Abbildung zusammenzufassen. Schauen wir uns dafür zunächst den subplot-Befehl und dessen Argumente in der R-internen Hilfe an. Gib dafür ?subplot in der Konsole ein. Neben den default-Einstellungen dieser Funktion findest du darin die folgende Auflistung von Argumenten:

Wie man sieht, gibt man in dieser Funktion zunächst “any number of plotly/ggplot2 objects” an - das heißt, dass unser erstes Argument einfach eine mit Kommas getrennte Auflistung der drei Plots sein kann: p1, p2, p3. Das könnte jedoch mit Problemen verbunden sein, da jeder Plot so noch seine eigene Legende mitbringt. Um das zu vermeiden, kann man für jedes Plot-Objekt nach folgendem Schema einzeln angeben, ob man die Legende haben möchte: style(p1, showlegend = FALSE).
Darüber hinaus stellt sich die Frage, ob man möchte, dass die Plots jeweils eigene Achsen haben oder ob sich diese geteilt werden sollen. Dies ist prinzipiell mit den Argumenten shareY und shareX möglich. Für den Geschlechtervergleich gibt es nur drei Plots, die vermutlich ohne Probleme nebeneinander dargestellt werden können. Aus diesem Grund macht es zwar eigentlich nur Sinn, dass die Plots sich ihre y-Achse teilen - zu einem Teilen der x-Achse käme es ja sowieso nicht (die Plots sind ja nebeneinander). Doch ohne shareX = TRUE würde die x-Achsenbeschriftung fehlen, weshalb wir dieses Argument trotzdem einfach mit aufnehmen sollten.
subplot(style(p1, showlegend = FALSE),
style(p2, showlegend = FALSE),
style(p3, showlegend = FALSE),
shareY = TRUE, shareX = TRUE)Über die layout-Funktion kann man an der ganzen Abbildung nun noch Anpassungen vornehmen. Wie bereits erwähnt, können wir hier beispielsweise in einer Überschrift die örtliche Eingrenzung der Daten deutlich machen.
subplot(style(p1, showlegend = FALSE),
style(p2, showlegend = FALSE),
style(p3, showlegend = FALSE),
shareY = TRUE, shareX = TRUE) %>%
layout(
title = list(
text = 'LIFE EXPECTANCY DEVELOPMENT IN EUROPE - GENDER COMPARISON',
font = list(size = 17, color = "darkblue", family = ""),
xref = "paper",
x = 0.5,
yref = "paper",
y = 1
)
)Wie man hier sehen kann, ist die Position des Titels jedoch sehr ungünstig und auch eine Veränderung des y-Wertes (bspw. y = 1.1) hilft dabei nicht wirklich. Stattdessen kann man jedoch innerhalb der subplot-Funktion mit dem heights-Argument die prozentuale Höhe verändern, die die Plots in der Abbildung einnehmen sollen. Mit heights = c(0.92) kann man dadurch ausreichend Platz für die Überschrift sammeln. Normalerweise kommt das heights-Argument dann zur Anwendung, wenn der subplot aus mehreren Spalten besteht, um die Größe der einzelnen Spalten zu bestimmen. Aus diesem Grund wird diese Höhenangabe auch vektorisiert (c()).
subplot(style(p1, showlegend = FALSE),
style(p2, showlegend = FALSE),
style(p3, showlegend = FALSE),
shareY = TRUE, shareX = TRUE, heights = c(0.92)) %>%
layout(
title = list(
text = 'LIFE EXPECTANCY DEVELOPMENT IN EUROPE - GENDER COMPARISON',
font = list(size = 17, color = "darkblue", family = ""),
xref = "paper",
x = 0.5,
yref = "paper",
y = 1
)
)Mit dieser Veränderung hat man nun eine relativ ansehnliche Abbildung und kann die europäischen Länder bezüglich der Entwicklung ihrer genderabhängigen Lebenserwartung vergleichen. Dabei sind Vergleiche zwischen Ländern, aber auch zwischen den verschiedenen Genderausprägungen möglich.
Beispiel 2 soll ein Ländervergleich mit allen drei Genderbezeichnungen in einem Plot sein. Hierfür wählen wir beispielhaft die sechs europäischen Länder Deutschland, Italien, England, Russland, Moldawien und Lettland. Plot 1 mit Deutschland sieht dann folgendermaßen aus (größtenteils übernommen aus ‘Grafik 1’):
p1 <- filter(data2, COUNTRY == "Germany") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, COUNTRY == "Germany")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, COUNTRY == "Germany")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, COUNTRY == "Germany")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, COUNTRY == "Germany")$YEAR,
"<br><b>Sex:</b> ", filter(data2, COUNTRY == "Germany")$SEX,
"<br><b>Continent:</b> ", filter(data2, COUNTRY == "Germany")$CONTINENT),
hoverinfo = "text",
colors = viridisLite::magma(3)) %>%
layout(
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "#e6e6e6",
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center",
y = 0.5),
annotations = list(
text = "Germany",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 16,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.93,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "#e6e6e6",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p1p2 bis p6 werden jetzt nach dem selben Schema erstellt. Diese Anpassungen sind sehr repetetiv, weshalb du diese auch einfach überspringen kannst. Falls du doch Probleme hast, kannst du dir die einzelnen Plots im folgenden Unterabschnitt anschauen.
Erstellen von
p2 bis p6p2: COUNTRY = “Italy”
p2 <- filter(data2, COUNTRY == "Italy") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, COUNTRY == "Italy")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, COUNTRY == "Italy")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, COUNTRY == "Italy")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, COUNTRY == "Italy")$YEAR,
"<br><b>Sex:</b> ", filter(data2, COUNTRY == "Italy")$SEX,
"<br><b>Continent:</b> ", filter(data2, COUNTRY == "Italy")$CONTINENT),
hoverinfo = "text",
colors = viridisLite::magma(3)) %>%
layout(
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "#e6e6e6",
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center",
y = 0.5),
annotations = list(
text = "Italy",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 16,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.93,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "#e6e6e6",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p2p3: COUNTRY = “United Kingdom”
p3 <- filter(data2, COUNTRY == "United Kingdom") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, COUNTRY == "United Kingdom")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, COUNTRY == "United Kingdom")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, COUNTRY == "United Kingdom")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, COUNTRY == "United Kingdom")$YEAR,
"<br><b>Sex:</b> ", filter(data2, COUNTRY == "United Kingdom")$SEX,
"<br><b>Continent:</b> ", filter(data2, COUNTRY == "United Kingdom")$CONTINENT),
hoverinfo = "text",
colors = viridisLite::magma(3)) %>%
layout(
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "#e6e6e6",
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center",
y = 0.5),
annotations = list(
text = "United Kingdom",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 16,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.93,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "#e6e6e6",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p3p4: COUNTRY = “Russian Federation”
p4 <- filter(data2, COUNTRY == "Russian Federation") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, COUNTRY == "Russian Federation")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, COUNTRY == "Russian Federation")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, COUNTRY == "Russian Federation")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, COUNTRY == "Russian Federation")$YEAR,
"<br><b>Sex:</b> ", filter(data2, COUNTRY == "Russian Federation")$SEX,
"<br><b>Continent:</b> ", filter(data2, COUNTRY == "Russian Federation")$CONTINENT),
hoverinfo = "text",
colors = viridisLite::magma(3)) %>%
layout(
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "#e6e6e6",
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center",
y = 0.5),
annotations = list(
text = "Russian Federation",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 16,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.93,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "#e6e6e6",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p4p5: COUNTRY = “Moldova, Republic of”
p5 <- filter(data2, COUNTRY == "Moldova, Republic of") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, COUNTRY == "Moldova, Republic of")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, COUNTRY == "Moldova, Republic of")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, COUNTRY == "Moldova, Republic of")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, COUNTRY == "Moldova, Republic of")$YEAR,
"<br><b>Sex:</b> ", filter(data2, COUNTRY == "Moldova, Republic of")$SEX,
"<br><b>Continent:</b> ", filter(data2, COUNTRY == "Moldova, Republic of")$CONTINENT),
hoverinfo = "text",
colors = viridisLite::magma(3)) %>%
layout(
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "#e6e6e6",
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center",
y = 0.5),
annotations = list(
text = "Moldova, Republic of",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 16,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.93,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "#e6e6e6",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p5p6: COUNTRY = “Latvia”
p6 <- filter(data2, COUNTRY == "Latvia") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, COUNTRY == "Latvia")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, COUNTRY == "Latvia")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, COUNTRY == "Latvia")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, COUNTRY == "Latvia")$YEAR,
"<br><b>Sex:</b> ", filter(data2, COUNTRY == "Latvia")$SEX,
"<br><b>Continent:</b> ", filter(data2, COUNTRY == "Latvia")$CONTINENT),
hoverinfo = "text",
colors = viridisLite::magma(3)) %>%
layout(
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "#e6e6e6",
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center",
y = 0.5),
annotations = list(
text = "Latvia",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 16,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.93,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "#e6e6e6",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p6Zu diesem Zeitpunkt solltest du alle 6 Plots erstellt haben, sodass wir nun zu der Erstellung des subplots übergehen können. Prinzipiell ändert sich wenig an den Argumenten innerhalb der subplot-Funktion. Übernehmen wir deshalb erst einmal den Befehl aus Beispiel 1:
subplot(style(p1, showlegend = FALSE),
style(p2, showlegend = FALSE),
style(p3, showlegend = FALSE),
shareY = TRUE, shareX = TRUE, heights = c(0.92)) %>%
layout(
title = list(
text = 'LIFE EXPECTANCY DEVELOPMENT IN EUROPE - GENDER COMPARISON',
font = list(size = 17, color = "darkblue", family = ""),
xref = "paper",
x = 0.5,
yref = "paper",
y = 1
)
)Hier müssen jetzt noch die drei fehlenden Plots hinzugefügt werden. Außerdem werden sechs Plots vermutlich nicht in eine Zeile passen - dafür brauchen wir also das richtige Argument. Versuche das richtige Argument hier zu finden:

Das benötigte Argument in diesem Fall ist nrows, denn wir wollen die Plots zwei-reihig anordnen. In Folge dessen muss auch das heights-Argument angepasst werden - dort müssen nun zwei Werte stehen (zwei Zeilen - jeweils muss eine prozentuale Höhe angegeben werden).
Zuletzt kann man noch den Titel ein wenig anpassen und fertig ist die Abbildung:
subplot(style(p1, showlegend = FALSE), style(p2, showlegend = FALSE),
style(p3, showlegend = FALSE), style(p4, showlegend = FALSE),
style(p5, showlegend = FALSE), style(p6, showlegend = FALSE),
shareY = TRUE, shareX = TRUE, nrows = 2, heights = c(0.46, 0.46)) %>%
layout(
title = list(
text = 'LIFE EXPECTANCY DEVELOPMENT IN EUROPE - COUNTRY COMPARISON',
font = list(size = 17, color = "darkblue", family = ""),
xref = "paper",
x = 0.5,
yref = "paper",
y = 1
)
)Grafik 4 - Statische Kartendarstellung
In der vierten vorgeschlagen Abbildung soll eine statische Kartendarstellung erstellt werden. Diese soll als Vorstufe für die animierte Kartendarstellung gelten und bildet demnach das Grundgerüst für Grafik 5. Wichtig ist dafür zunächst einmal, dass wir die Datengrundlage eingrenzen. Auf jeden Fall benötigen wir hier nur Daten von Ländern - damit fallen die WHO-Regionen raus (also SpatialDimType == "COUNTRY". Da die einzelnen Länder je nach Lebenserwartung eingefärbt werden sollen, brauchen wir außerdem pro Land nur EINEN Datenpunkt. Das heißt, wir beschränken uns in diesem Beispiel auf Daten beider Geschlechter (also SEX == "BTSX") aus dem Jahr 2014 (also YEAR == 2014). Das bedeutet, dass unsere Karte auf folgendem Datensatz basieren wird:
filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX" & YEAR == 2014)## # A tibble: 183 x 11
## DatID VarID SpatialDimType COUNTRYCODE YEAR SEX LIFE_EXPECTANCY L_E2EXACT
## <dbl> <chr> <chr> <chr> <dbl> <chr> <chr> <dbl>
## 1 1.82e7 WHOS… COUNTRY AFG 2014 BTSX 63.0 63.0
## 2 1.82e7 WHOS… COUNTRY AGO 2014 BTSX 61.7 61.7
## 3 1.82e7 WHOS… COUNTRY ALB 2014 BTSX 76.1 76.1
## 4 1.82e7 WHOS… COUNTRY ARE 2014 BTSX 76.9 76.9
## 5 1.82e7 WHOS… COUNTRY ARG 2014 BTSX 76.7 76.7
## 6 1.82e7 WHOS… COUNTRY ARM 2014 BTSX 74.5 74.5
## 7 1.82e7 WHOS… COUNTRY ATG 2014 BTSX 74.6 74.6
## 8 1.82e7 WHOS… COUNTRY AUS 2014 BTSX 82.7 82.7
## 9 1.82e7 WHOS… COUNTRY AUT 2014 BTSX 81.7 81.7
## 10 1.82e7 WHOS… COUNTRY AZE 2014 BTSX 72.6 72.6
## # … with 173 more rows, and 3 more variables: COMMENT <chr>, COUNTRY <chr>,
## # CONTINENT <chr>Aufschluss darüber, wie du eine Kartendarstellung mit plotly erstellst, gibt folgendes Unterkapitel aus dem Buch zu plotly, das bereits in der Übersicht zu Projekt 8 zur Vorbereitung vorgestellt wurde. Unter dem Punkt 4.1.2 “Chloropleths” findest du dann direkt als erstes Beispiel eine Karte, nach deren Prinzip auch wir unsere Karte erstellen können:
#Zuerst erstellen wir ein neues Farbschema (von rot bis grün), um damit positiv und negativ zu symbolisieren.
CRP <- colorRampPalette(c('darkred', 'red', 'orange', 'yellow', 'green', 'darkgreen'))
#Dann erstellen wir die Karte
filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX" & YEAR == 2014) %>% #Auswahl der Daten
plot_geo() %>% # Erstellung einer Karte
add_trace(
z = ~L_E2EXACT, # Einfärbungsvariable - Lebenserwartung
locations = ~COUNTRYCODE, # Wie werden die Daten den Ländern auf der Karte zugeordnet? Welche Variable soll dafür verwendet werden?
locationmode = 'ISO-3', # Wie liegen diese Ortsangaben vor? -> Im `ISO-3`-Format
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX" & YEAR == 2014)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX" & YEAR == 2014)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX" & YEAR == 2014)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX" & YEAR == 2014)$YEAR,
"<br><b>Sex:</b> ", filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX" & YEAR == 2014)$SEX,
"<br><b>Continent:</b> ", filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX" & YEAR == 2014)$CONTINENT),
hoverinfo = "text",
colors = CRP(20), #sorgt für 20 farbliche Abstufungen - Genauigkeit der Map
zmin = 35, zmax = 85 #vorbestimmtes Minimum und Maximum der Legende
) %>%
layout( #optische Anpassung...
geo = list( #...der Karte
scope = "world", #Weltkarte oder nur ein bestimmter Kontinent/nur die USA?
showocean = TRUE, #sollen Ozeane angezeigt werden?
oceancolor = '#80bfff' #Welche Farbe sollen Ozeane haben?
),
title = list( #...des Titels (bereits bekannt)
text = 'WHO Measurement of the Life Expectancy around the World in 2014',
font = list(size = 18, color = "darkblue", family = ""), #Schrift
xref = "paper", x = 0.5, #Positionierung auf der x-Achse
yref = "paper", yanchor = "bottom", y = 0.85 #Positionierung auf der y-Achse
)
) %>%
colorbar( #Anpassen der `colorbar` (Leiste, die am Rand als Legende fungiert - Was bedeuten die abgebildeten Farben?)
title = list(
text = '<b>Life Expectancy</b>', #Titel der Colorbar
side = "right"), #Position/Richtung des Titels: `right` = rechts von der Colorbar + senkrecht/von unten nach oben
y = 0.8 #Position auf der y-Achse
)Erklärungen und weitere Anpassungsmöglichkeiten findest du beispielsweise hier. Dort findest du alle Argumente, die unter layout in der Liste zu geo (kartenbezogene Anpassungen) möglich sind. So wird beispielsweise aufgeführt, welche scopes der Karte zur Verfügung stehen.
Grafik 5 - Animierte Kartendarstellung
Um die im Abschnitt ‘Grafik 4’ erstellte Karte nun zu animieren, müssen lediglich einige wenige Anpassungen vorgenommen werden. Welche das genau sind, findest du hier. Dabei empfehlen wir dir, den Abschnitt 14.1 “Animation API” vollständig zu lesen und dir die insbesondere die ersten beiden Beispiele genau anzuschauen - denn diese beinhalten alles, was du für eine animierte Kartendarstellung benötigst. Aber machen wir eines nach dem anderen:
Initiierung der Animation
1. Zunächst müssen wir den Filter der Daten verändern. In ‘Grafik 4’ haben wir uns hier noch auf ein bestimmtes Jahr beschränken müssen. In einer Animation können wir jede beliebige Zeitspanne verwenden, werden in diesem Beispiel der Einfachheit halber aber alle Daten verwenden. Der filter-Befehl muss demnach um den & YEAR == 2014 Zusatz bereinigt werden.
- Innerhalb der
add_trace-Funktion muss außerdem deutlich werden, welche Variable als “Animationsvariable” verwendet werden soll (Welche Variable ist Treiber der Animation und ordnet diese?). In diesem Fall ist das demnach dieYEAR-Variable, die in dieser animierten Abbildung neu hinzu kommt. Wie der oben verlinkte Buchausschnitt erwähnt, verdeutlich man das mit demframe-Argument.
Modifikation der Animation
3. Damit die Animation jetzt auch noch richtig funktioniert, müssen ein paar Anpassungen vorgenommen werden. Öffnet man die Animation ohne diese Anpassungen, so ändern sich lediglich die in der hoverinfo angezeigten Daten der Länder und nicht deren entsprechende Färbung. Dafür müssen wir außerhalb der add_trace-Funktion direkt die Animation formatieren. Das funktioniert über die animation_opts-Funktion, in die wir mithilfe von %>% die bisherige Abbildung überführen. Darin können wir nun bspw. anpassen, wie lange die einzelnen Frames angezeigt werden sollen (in ms) und wie die Übergänge aussehen sollen (bspw. easing = "linear"). Am wichtigsten ist jedoch das redraw-Argument. Hiermit bestimmt man, ob die Karte für jedes Frame neu gezeichnet werden soll (TRUE) oder nicht (FALSE-default). Da sich bei uns die Färbung der Länder auf Basis der Lebenserwartung verändern soll, müssen wir hier also redraw = TRUE angeben.
- Zuletzt kannst du den
animation_buttonund denanimation_sliderin weiteren Funktionen anpassen. Hier kannst du beispielsweise die Position der beiden Elemente manuell anpassen. Beimanimation_sliderhaben wir außerdem eine Anmerkung beigefügt und formatiert, die den aktuellen Wert der Animation anzeigt (in unserem Fall also, welches Jahr die Karte momentan darstellt):currentvalue = list(prefix = "YEAR ", font = list(color="darkblue")).
filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX") %>%
plot_geo() %>%
add_trace(
z = ~L_E2EXACT,
locations = ~COUNTRYCODE,
locationmode = 'ISO-3',
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX")$YEAR,
"<br><b>Sex:</b> ", filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX")$SEX,
"<br><b>Continent:</b> ", filter(data2, SpatialDimType == "COUNTRY" & SEX == "BTSX")$CONTINENT),
hoverinfo = "text",
colors = CRP(20),
zmin = 35, zmax = 85,
frame = ~YEAR
) %>%
animation_opts(1000, easing = "linear", redraw = TRUE) %>% #redraw muss auf TRUE, damit die Karte immer wieder neu bemalt wird
animation_button(
x = 1, xanchor = "right", y = 0.075, yanchor = "bottom" #Position des Play-Buttons
) %>%
animation_slider(
currentvalue = list(prefix = "YEAR ", font = list(color="darkblue")), #prefix - string der vor Variable über der Leiste abgedruckt werden soll, dahinter die Farbe
y = -0.05, yanchor = "bottom" #Position der Slider-Leiste
) %>%
layout(
geo = list(
scope = "world",
showocean = TRUE,
oceancolor = '#80bfff'
),
title = list(
text = 'WHO Measurement of the Development of Life Expectancy around the World',
font = list(size = 18, color = "darkblue", family = ""),
xref = "paper", x = 0.5,
yref = "paper", yanchor = "bottom", y = 0.85
)
) %>%
colorbar(
title = list(
text = '<b>Life Expectancy</b>',
side = "right"),
y = 0.8
)Umsetzung einer shiny-App
Zu diesem Zeitpunkt solltest du einen guten Überblick über das Erstellen von Abbildungen mit plotly erhalten haben. In diesem Abschnitt geht es nun darum, die erstellten Abbildungen in eine shiny-App zu überführen, um damit die Vorteile von shiny und plotly zu kombinieren. An diesem Punkt ist es empfehlenswert, zunächst in Projekt 7 - ShinyR reinzuschauen, da sich dieses ausschließlich mit der Erstellung von shiny-Apps auseinandersetzt. Aus diesem Grund werden die folgenden Abhandlungen nicht bis ins Detail alle shiny-Grundlagen erklären. Stattdessen werden wir nur auf einige neue Elemente genauer eingehen.
conditionalPanel-Befehl und zum anderen auf der generellen App-Struktur.
Das User Interface
Beginnen wir das Ganze, indem wir das shiny-Grundgerüst öffnen. Gib dafür einfach shiny in das “Source”-Fenster (oben links - da wo du deinen Code eingibst und ausführst) ein und wähle dann in den dazugehörigen Vorschlägen “shinyapp {snippet}” aus. Folgendes Grundgerüst öffnet sich dann automatisch (Anmerkung: Sofern du das shiny-Paket bisher noch nicht installiert hast, dann solltest du das an diesem Punkt mit install.packages("shiny") tun):
#install.packages("shiny")
library(shiny)
ui <- fluidPage(
)
server <- function(input, output, session) {
}
shinyApp(ui, server)Jetzt müssen wir uns überlegen, was die App alles beinhalten soll. Zuvor haben wir fünf bzw. sechs verschiedene Abbildungen mit plotly erstellt. Gehen wir an diesem Punkt einfach davon aus, dass wir alle Abbildungen in die shiny-App übernehmen wollen (ausgenommen ‘Grafik 4’, da statische Karten auch durch ‘Grafik 5’ abgebildet werden können). Alle diese Darstellungen haben unterschiedliche Parameter, die reaktiv gestaltet werden können. Deshalb sollte man ganz am Anfang der App auswählen können, welche Abbildung man erstellen möchte. Anhand dieser Auswahl sollte dann eine darauf abgestimmte Kombination an input-Fenstern erscheinen.
Bauen wir also erst einmal das Grundgerüst der App und die Auswahl der Abbildung:
ui <- fluidPage(
theme = "bootstrap.css", style = "background: #337ab7; color: white",
titlePanel(
h1(strong("Life Expectancy all over the world (by GHO)"), align = "center"),
windowTitle = "Life Expectancy all over the world (by GHO)"),
wellPanel(
style = "background: #87CEFA; border-color: #2e6da4",
h1(strong("Illustration")),
selectInput(
inputId = "type",
label = "Type of Illustration",
choices = c("Select a type of illustration!" = "", "Map",
"Diagram (Country Comparison)" = "Diagram1",
"Diagram (Gender Comparison)" = "Diagram2",
"Diagram (Country x Gender Comparison)" = "Diagram3",
"Diagram (Country x Country Comparison)" = "Diagram4")
)
)
)
shinyApp(ui, server)Im titlePanel haben wir der Seite erst einmal einen Titel gegeben und diesen zentriert. Außerdem haben wir dem Browser-Fenster über windowTitle einen Namen gegeben - diesen Namen trägt dann der Tab in deinem Browser, in dem die App geöffnet wurde. Danach haben wir ein wellPanel verwendet, um die beiden Abschnitte voneinander abzugrenzen. Hier kann man auch andere Seitenstrukturen wählen, wir wollen es jedoch einfach halten und werden den Input- und Output-Block einfach auf der vollen Seitenbreite direkt unter die Überschrift packen. Für dieses Panel geben wir wiederum style-Vorschriften, geben eine Überschrift und erstellen den selectInput, mit dem später die Art der Abbildung ausgewählt werden soll.
Besonderheit: Im choices-Argument des selectInput-Befehls haben wir die Möglichkeit genutzt, den Wortlaut der Auswahlmöglichkeiten im User Interface und die Information, die im Server ankommt, zu modifizieren:
- Die erste Auswahlmöglichkeit stellt einen Platzhalter dar (“Select a type of illustration”). Das erkennt die Funktion deshalb, da dieser Möglichkeit im Server ein fehlender Wert ("") zugewiesen wurde.
- Bei allen anderen Möglichkeiten wird der erste Text im User Interface angezeigt, während man diesen Wert nur über den zweiten “Server-Wert” abrufen kann. Das ermöglicht nähere Erläuterungen für den Betrachter, ohne dabei die Schreibarbeit im Server(/User Interface) unnötig zu verkomplizieren. Wählt also jemand die Möglichkeit “Diagram (Country Comparison)” so ergibt sich im Server der Wert “Diagram1” (=
input$type == "Diagram1").
Im nächsten Schritt können wir uns den einzelnen Abbildungen widmen und was diese jeweils für Inputs benötigen.
- Die Kartendarstellung (
input$type == "Map") - Hier brauchen wir Inputs, um die Ausprägung auf der Gendervariable, eine Zeitspanne der angezeigten Daten und den Titel der Grafik zu bestimmen. Darüber hinaus benötigen wir einenactionButton, um alle Angaben gebündelt auf die Abbildung anzuwenden. Zuletzt müssen wir uns um die örtliche Eingrenzung der Daten kümmern. Hier sollte es zum einen möglich sein, entweder alle Länder einzufärben oder nur die Länder eines bestimmten Kontinents. Zum anderen sollte man in der Lage sein, diescopeder Karte zu verändern. Das soll aber nur dann möglich sein, wenn in dem anderenselectInputnicht nichts (das ist in der Ausgangsstellung mit dem Platzhalter der Fall) oder alle Daten ausgewählt wurden.
conditionalPanel(
#Wenn im Input mit der Id 'type' die Möglichkeit 'Map' ausgewählt wurde, dann soll das User Interface folgende Inputs enthalten:
condition = "input.type == 'Map'",
#Die Inputs werden mit `fluidRow` und `column` strukturiert.
fluidRow(
column(3, selectInput( #Ein Input mit vorgegebenen Möglichkeiten zur Bestimmung der Gendervariable.
inputId = "sex",
label = "Sex",
choices = c("Select a sex!" = "", "Both Sexes" = "BTSX", "Male" = "MLE", "Female" = "FMLE"), #Für UI "übersetzt" in ganze Wörter
selectize = TRUE
)),
column(2, selectInput( #Ein Input mit vorgegebenen Möglichkeiten zur Bestimmung der Zeitspanne - Beginn.
inputId = "time1", label = "Time Interval",
choices = c("Start" = "", 2000:2016)
)),
column(2, selectInput( #Ein Input mit vorgegebenen Möglichkeiten zur Bestimmung der Zeitspanne - Ende.
inputId = "time2", label = "Time Interval",
choices = c("End" = "", 2000:2016)
)),
column(3, textInput( #Ein freier Input für Text, um den Titel der Abbildung zu bestimmen.
inputId = "title", label = "Title of the Illustration", placeholder = "Give your illustration a title!"
)),
column(2, br(), actionButton( #Ein Break(`br()`), um den `actionButton` tiefer zu platzieren.
inputId = "generate", label = strong("Generate Map")
))
),
fluidRow(
column(6, selectInput( #Ein Input mit vorgegebenen Möglichkeiten zur Bestimmung des örtlichen Umfangs der verwendeten Daten.
inputId = "scope",
label = "Scope of the Data",
choices = c("Select a scope!" = "", "World", "Africa", "Americas", "Asia", "Europe", "Oceania"),
selectize = TRUE
)),
column(6, conditionalPanel(
#Wurde bei input$scope nichts oder "World" ausgewählt, dann wird dieses Input-Fenster gar nicht angezeigt.
condition = "input.scope != 'World' & input.scope != ''",
#Wenn ein Kontinent gewählt wurde, kann man nun bestimmen, ob auch die Karte auf einen bestimmten Kontinent begrenzt werden soll.
selectInput(
inputId = "map", label = "Scope of the Map",
choices = c("Should the map be reduced to your chosen continent?" = "",
"No" = "world", "Africa" = "africa",
"North America" = "north america",
"South America" = "south america",
"Asia" = "asia", "Europe" = "europe")
)
))
)
)Anmerkung: Bei den choices-Argumenten wurde deshalb mit einer UI- und einer Server-Variante gearbeitet, da die angepassten Server-Varianten zwar perfekt in die Manipulation der Abbildungen eingesetzt werden können, jene Schreibweisen aber nicht einheitlich sind und teilweise unverständlich für den Anwender (Bsp.: SEX == “BTSX” - Was bedeutet “BTSX”?).
- Liniendiagramm 1 (
input$type == "Diagram1") - Hier brauchen wir Inputs für Geschlecht, Anfang und Ende der zu betrachtenden Zeitspanne, den Titel der Abbildung und zur örtlichen Eingrenzung der betrachteten Daten. Letzterer Input wird dabei auf sieben Möglichkeiten eingeschränkt: Länder, Regionen und die fünf Kontinente (Afrika, Amerika, Asien, Europa und Ozeanien). Zuletzt benötigen wir noch einen Action Button und fertig ist das User Interface für das erste Liniendiagramm.
conditionalPanel(
condition = "input.type == 'Diagram1'",
fluidRow(
column(2, selectInput(
inputId = "scopeB",
label = "Scope of the Data",
choices = c("Select a scope!" = "", "Regions" = "REGION", "Countries" = "COUNTRY",
"Africa", "Americas", "Asia", "Europe", "Oceania"),
selectize = TRUE
)),
column(2, selectInput(
inputId = "sexB",
label = "Sex",
choices = c("Select a sex!" = "", "Both Sexes" = "BTSX", "Male" = "MLE", "Female" = "FMLE"),
selectize = TRUE
)),
column(2, selectInput(
inputId = "time1B", label = "Time Interval",
choices = c("Start" = "", 2000:2016)
)),
column(2, selectInput(
inputId = "time2B", label = "Time Interval",
choices = c("End" = "", 2000:2016)
)),
column(2, textInput(
inputId = "titleB", label = "Title of the Illustration",
placeholder = "Give your illustration a title!"
)),
column(2, br(), actionButton(
inputId = "generateB", label = strong("Generate Diagram")
))
)
)- Liniendiagramm 2 (
input$type == "Diagram2") - Für die dritte Abbildung kommt nun eine Besonderheit hinzu. Gehen wir aber zunächst die repetitiven Inputs durch: Zeitintervall (2 Inputs für Anfang und Ende), Titeleingabe und Action Button. In einer weiteren Reihe (fluidRow) setzen wir nun die Auswahl eines Landes oder einer Region. Dafür soll man zunächst übergeordnet auswählen können, auf welchem Kontinent das gesuchte Land liegt, um dann in einem weiteren Input aus einer kleineren Liste auswählen zu können. Hinzu kommt noch die Möglichkeit, eine WHO-Region auszuwählen. Das heißt, dass wir einen Input haben wollen, dessen Auswahl beeinflusst, welche Antwortmöglichkeiten ein anderer Input bereitstellt. Hierbei handelt es sich dementsprechend um eine reaktive Operation, die innerhalb des Servers stattfinden muss - dies fordert also die Verwendung deruiOutput-Funktion. In diesem Fall nennen wir den Output “Choices”, sodass wir diesen im Server überoutput$Choicesanwählen bzw. erstellen können (für die Umsetzung davon siehe “Der Server - Teil 1”).
conditionalPanel(
condition = "input.type == 'Diagram2'",
fluidRow(
column(3, selectInput(
inputId = "time1C", label = "Time Interval",
choices = c("Start" = "", 2000:2016)
)),
column(3, selectInput(
inputId = "time2C", label = "Time Interval",
choices = c("End" = "", 2000:2016)
)),
column(3, textInput(
inputId = "titleC", label = "Title of the Illustration",
placeholder = "Give your illustration a title!"
)),
column(3, br(), actionButton(
inputId = "generateC", label = strong("Generate Diagram")
))
),
fluidRow(
column(6, selectInput(
inputId = "scope1C",
label = "Scope of the Data",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(6, uiOutput("Choices"))
)
)- Mehrere Liniendiagramme 1 (
input$type == "Diagram3") - In dieser Abbildung sollen mehrere Plots nach dem Typ der vorherigen Abbildung (3.) in einemsubplotzusammengefasst werden. Der generelle Aufbau des User Interfaces ist demnach identisch bis auf den Fakt, dass für alle sechs möglichen Plots (Maximum) die örtliche Eingrenzung der Daten einzeln geschehen muss. Deshalb wird dieser Teil des User Interfaces aus 3. einfach sechs Mal kopiert. Dabei ist es jedoch von besonderer Relevanz, die Id’s der einzelnen Input schlüssig zu wählen, damit es später im Server nicht zu kompliziert wird. (Anmerkung: Dieser Teil der App ist sehr repetitiv. Möglicherweise gibt es hier deshalb eine klügere und sparsamere Vorgehensweise.)
conditionalPanel(
condition = "input.type == 'Diagram3'",
fluidRow(
column(3, selectInput(
inputId = "time1D", label = "Time Interval",
choices = c("Start" = "", 2000:2016)
)),
column(3, selectInput(
inputId = "time2D", label = "Time Interval",
choices = c("End" = "", 2000:2016)
)),
column(3, textInput(
inputId = "titleD", label = "Title of the Illustration",
placeholder = "Give your illustration a title!"
)),
column(3, br(), actionButton(
inputId = "generateD", label = strong("Generate Diagram")
))
),
h2("Which countries(/regions) do you want to compare? (max. 6)"),
fluidRow(
column(3, selectInput(
inputId = "scope1D1",
label = "Scope of the Data 1",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices1")),
column(3, selectInput(
inputId = "scope1D2",
label = "Scope of the Data 2",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices2"))
),
fluidRow(
column(3, selectInput(
inputId = "scope1D3",
label = "Scope of the Data 3",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices3")),
column(3, selectInput(
inputId = "scope1D4",
label = "Scope of the Data 4",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices4"))
),
fluidRow(
column(3, selectInput(
inputId = "scope1D5",
label = "Scope of the Data 5",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices5")),
column(3, selectInput(
inputId = "scope1D6",
label = "Scope of the Data 6",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices6"))
)
)- Mehrere Liniendiagramme 2 (
input$type == "Diagram4") - Die letzte Abbildung soll auch einsubplotsein. Dieser soll sich aus Plots zu den fünf Kontinenten und einem Plot zu den WHO-Regionen zusammensetzen. Dabei sollen ledigleich Zeitspanne, Geschlecht und Titel manipuliert werden können. Demnach ergibt sich folgendes User Interface:
conditionalPanel(
condition = "input.type == 'Diagram4'",
fluidRow(
column(2, selectInput(
inputId = "time1E", label = "Time Interval",
choices = c("Start" = "", 2000:2016)
)),
column(2, selectInput(
inputId = "time2E", label = "Time Interval",
choices = c("End" = "", 2000:2016)
)),
column(2, selectInput(
inputId = "sexE",
label = "Sex",
choices = c("Select a sex!" = "", "Both Sexes" = "BTSX", "Male" = "MLE", "Female" = "FMLE"),
selectize = TRUE
)),
column(3, textInput(
inputId = "titleE", label = "Title of the Illustration",
placeholder = "Give your illustration a title!"
)),
column(3, br(), actionButton(
inputId = "generateE", label = strong("Generate Diagram")
))
)
)- Das Endergebnis: Zusammensetzung aller Bestandteile des User Interfaces der einzelnen Abbildungen - Diese fünf Abschnitte des User Interfaces müssen wir jetzt nur noch zusammensetzen und am Ende um einen
plotlyOutputergänzen. Diese Funktion schafft einen Raum für die im Server erstellten Abbildungen, dem diese Abbildungen dann über dieoutputIdzugewiesen werden können. Dieses Fenster soll unabhängig von der Auswahl des Abbildungstyps bestehen und wird deshalb außerhalb derconditionalPanels erstellt.
ui <- fluidPage(
theme = "bootstrap.css", style = "background: #337ab7; color: white",
titlePanel(
h1(strong("Life Expectancy all over the world (by GHO)"), align = "center"),
windowTitle = "Life Expectancy all over the world (by GHO)"),
wellPanel(
style = "background: #87CEFA; border-color: #2e6da4",
h1(strong("Illustration")),
selectInput(
inputId = "type",
label = "Type of Illustration",
choices = c("Select a type of illustration!" = "", "Map",
"Diagram (Country Comparison)" = "Diagram1",
"Diagram (Gender Comparison)" = "Diagram2",
"Diagram (Country x Gender Comparison)" = "Diagram3",
"Diagram (Country x Country Comparison)" = "Diagram4")
),
conditionalPanel(
condition = "input.type == 'Map'",
fluidRow(
column(3, selectInput(
inputId = "sex",
label = "Sex",
choices = c("Select a sex!" = "", "Both Sexes" = "BTSX", "Male" = "MLE", "Female" = "FMLE"),
selectize = TRUE
)),
column(2, selectInput(
inputId = "time1", label = "Time Interval",
choices = c("Start" = "", 2000:2016)
)),
column(2, selectInput(
inputId = "time2", label = "Time Interval",
choices = c("End" = "", 2000:2016)
)),
column(3, textInput(
inputId = "title", label = "Title of the Illustration",
placeholder = "Give your illustration a title!"
)),
column(2, br(), actionButton(
inputId = "generate", label = strong("Generate Map")
))
),
fluidRow(
column(6, selectInput(
inputId = "scope",
label = "Scope of the Data",
choices = c("Select a scope!" = "", "World", "Africa", "Americas", "Asia", "Europe", "Oceania"),
selectize = TRUE
)),
column(6, conditionalPanel(
condition = "input.scope != 'World' & input.scope != ''",
selectInput(
inputId = "map", label = "Scope of the Map",
choices = c("Should the map be reduced to your chosen continent?" = "",
"No" = "world", "Africa" = "africa",
"North America" = "north america",
"South America" = "south america", "Asia" = "asia",
"Europe" = "europe"))
))
)
),
conditionalPanel(
condition = "input.type == 'Diagram1'",
fluidRow(
column(2, selectInput(
inputId = "scopeB",
label = "Scope of the Data",
choices = c("Select a scope!" = "","Regions" = "REGION", "Countries" = "COUNTRY",
"Africa", "Americas", "Asia", "Europe", "Oceania"),
selectize = TRUE
)), #Erst "World", "Continent", "Country" - dann je nachdem nen selectInput
column(2, selectInput(
inputId = "sexB",
label = "Sex",
choices = c("Select a sex!" = "", "Both Sexes" = "BTSX", "Male" = "MLE", "Female" = "FMLE"),
selectize = TRUE
)),
column(2, selectInput(
inputId = "time1B", label = "Time Interval",
choices = c("Start" = "", 2000:2016)
)),
column(2, selectInput(
inputId = "time2B", label = "Time Interval",
choices = c("End" = "", 2000:2016)
)),
column(2, textInput(
inputId = "titleB", label = "Title of the Illustration",
placeholder = "Give your illustration a title!"
)),
column(2, br(), actionButton(
inputId = "generateB", label = strong("Generate Diagram")
))
)
),
conditionalPanel(
condition = "input.type == 'Diagram2'",
fluidRow(
column(3, selectInput(
inputId = "time1C", label = "Time Interval",
choices = c("Start" = "", 2000:2016)
)),
column(3, selectInput(
inputId = "time2C", label = "Time Interval",
choices = c("End" = "", 2000:2016)
)),
column(3, textInput(
inputId = "titleC", label = "Title of the Illustration",
placeholder = "Give your illustration a title!"
)),
column(3, br(), actionButton(
inputId = "generateC", label = strong("Generate Diagram")
))
),
fluidRow(
column(6, selectInput(
inputId = "scope1C",
label = "Scope of the Data",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(6, uiOutput("Choices"))
)
),
conditionalPanel(
condition = "input.type == 'Diagram3'",
fluidRow(
column(3, selectInput(
inputId = "time1D", label = "Time Interval",
choices = c("Start" = "", 2000:2016)
)),
column(3, selectInput(
inputId = "time2D", label = "Time Interval",
choices = c("End" = "", 2000:2016)
)),
column(3, textInput(
inputId = "titleD", label = "Title of the Illustration",
placeholder = "Give your illustration a title!"
)),
column(3, br(), actionButton(
inputId = "generateD", label = strong("Generate Diagram")
))
),
h2("Which countries(/regions) do you want to compare? (max. 6)"),
fluidRow(
column(3, selectInput(
inputId = "scope1D1",
label = "Scope of the Data 1",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices1")),
column(3, selectInput(
inputId = "scope1D2",
label = "Scope of the Data 2",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices2"))
),
fluidRow(
column(3, selectInput(
inputId = "scope1D3",
label = "Scope of the Data 3",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices3")),
column(3, selectInput(
inputId = "scope1D4",
label = "Scope of the Data 4",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices4"))
),
fluidRow(
column(3, selectInput(
inputId = "scope1D5",
label = "Scope of the Data 5",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices5")),
column(3, selectInput(
inputId = "scope1D6",
label = "Scope of the Data 6",
choices = c("Choose a continent or 'WHO-Region'!" = "", "Africa", "Americas", "Asia", "Europe", "Oceania", "WHO-Region"),
selectize = TRUE
)),
column(3, uiOutput("Choices6"))
)
),
conditionalPanel(
condition = "input.type == 'Diagram4'",
fluidRow(
column(2, selectInput(
inputId = "time1E", label = "Time Interval",
choices = c("Start" = "", 2000:2016)
)),
column(2, selectInput(
inputId = "time2E", label = "Time Interval",
choices = c("End" = "", 2000:2016)
)),
column(2, selectInput(
inputId = "sexE",
label = "Sex",
choices = c("Select a sex!" = "", "Both Sexes" = "BTSX", "Male" = "MLE", "Female" = "FMLE"),
selectize = TRUE
)),
column(3, textInput(
inputId = "titleE", label = "Title of the Illustration",
placeholder = "Give your illustration a title!"
)),
column(3, br(), actionButton(
inputId = "generateE", label = strong("Generate Diagram")
))
)
),
fluidRow(
plotlyOutput(outputId = "diagram", height = "700px")
)
)
)Um nun die einzelnen Inputs und deren Reaktivität (auf die Auswahl des Abbildungstyps) auf deren Existenz, Funktionalität und Anordnung zu testen, kannst du die App mit folgenden Befehlen testweise erstellen. Alle uiOutputs müssen noch im Server erstellt werden - dafür siehe “Der Server - Teil 1 (uiOutput/renderUI)”
server <- function(input, output, session) {
}
shinyApp(ui, server)Mögliche offene Fragen:
Warum haben wir für alle Abbildungstypen ein komplett eigenes User Interface erstellt und nicht einige Inputs standardmäßig übernommen (bspw. Zeitspanne und Titel)?
- Zum einen kann man hier den optischen Aspekt erwähnen - durch die Reaktivität des gesamten User Interfaces auf den Darstellungstyp kann man die Inputs individuell anordnen. Zum anderen kann man so den Ursprung von Fehlern leichter diagnostizieren - läuft bei der Erstellung eines bestimmten Plots etwas schief, so sind mögliche Fehlerquellen im User Interface beschränkt auf den bestimmten Bereich dieser Abbildung.
Warum hat jede Abbildung ihren eigenen Action Button mit unterschiedlicher inputId?
- Zunächst einmal wurde hier jeweils ein Action Button verwendet, damit die Funktion zur Erstellung einer Abbildung nicht ständig versucht auf neue Informationen (Inputs) zu reagieren, ohne dass alle nötigen Informationen zur Verfügung stehen. Stattdessen soll die Funktion auf Knopfdruck neue Inputs gesammelt in die Abbildung aufnehmen - das verhindert Fehlermeldungen und verbessert die Perfomance. Darüber hinaus haben alle Action Buttons unterschiedliche
inputIds damit nur der dazugehörige Server-Teil “aktiviert” wird und nicht alles auf einmal - das würde mitunter zu einer Menge Fehlermeldungen führen.
Der Server - Teil 1 (
uiOutput/renderUI)
Fangen wir an mit den Vorbereitungen im Server. Damit im Server später Abbildungen erstellt werden können, müssen bestimmte R-Pakete geladen und der Datensatz eingelesen, angemessen vorbereitet und um neue Variablen (COUNTRY & CONTINENT) ergänzt werden. Das alles können wir einfach aus einem früheren Abschnitt der Lösungen übernehmen:
server <- function(input, output, ...) {
# Pakete laden
library(readxl)
library(tidyverse)
library(ISOcodes)
library(countrycode)
library(plotly)
# Daten einlesen
data1 <- read_excel("GHO-Daten.xlsx", na = "NA", sheet = "Tabelle4")
#Daten formatieren
data1 <- as_tibble(data1)
data2 <- data1 %>%
select_if(function(col) !is.logical(col)) %>%
select(-c(TimeDimType, Dim1Type, Date, TimeDimensionValue, TimeDimensionBegin,
TimeDimensionEnd))
names(data2) <- c('DatID', 'VarID', 'SpatialDimType', 'COUNTRYCODE', 'YEAR', 'SEX',
'LIFE_EXPECTANCY','L_E2EXACT', 'COMMENT')
data2 <- data2[-c(1:48), ] #Ruanda ist doppelt
# Neue Variablen erstellen: 1. Ländername, 2. Kontinent
for (i in 1:9438){
if (data2$COUNTRYCODE[i] == "AFR"){data2$COUNTRY[i] <- "African Region"}
else if (data2$COUNTRYCODE[i] == "AMR"){data2$COUNTRY[i] <- "Region of the Americas"}
else if (data2$COUNTRYCODE[i] == "EMR"){data2$COUNTRY[i] <- "Eastern Mediterranean Region"}
else if (data2$COUNTRYCODE[i] == "EUR"){data2$COUNTRY[i] <- "European Region"}
else if (data2$COUNTRYCODE[i] == "GLOBAL"){data2$COUNTRY[i] <- "GLOBAL"}
else if (data2$COUNTRYCODE[i] == "SEAR"){data2$COUNTRY[i] <- "South-East Asian Region"}
else if (data2$COUNTRYCODE[i] == "WPR"){data2$COUNTRY[i] <- "Western Pacific Region"}
else {data2$COUNTRY[i] <- ISO_3166_1$Name[ISO_3166_1$Alpha_3 == data2$COUNTRYCODE[i]]}
}
data2 <- data2 %>% mutate(
CONTINENT = countrycode(
sourcevar = data2$COUNTRY,
origin = "country.name",
destination = "continent",
nomatch = NA))
# Erstellen der Farbfunktion für die Karte
CRP <- colorRampPalette(c('darkred', 'red', 'orange', 'yellow', 'green', 'darkgreen'))
}Nun können wir uns dem Erstellen der reaktiven UI-Inhalte widmen. Da es sich bei allen sieben uiOutputs um die gleiche Art von Output handelt, brauchen wir uns nur mit einem im Detail beschäftigen. Es geht jeweils darum, die Auswahlmöglichkeiten in einem selectInput durch die Auswahl in einem anderen selectInput zu begrenzen. Wird beispielsweise im ersten Input mit der Id scope1C “Afrika” ausgewählt, dann sollen im zweiten Input mit der Id scope2C alle afrikanischen Länder des Datensatzes zur Auswahl stehen. Außerdem soll die Auswahl des zweiten selectInputs immer dann aktualisiert werden, wenn sich die Auswahl im ersten selectInput verändert.
Das bedeutet also, dass wir die renderUI-Funktion zur Erstellung des reaktiven UI-Inhaltes in eine observeEvent-Funktion einbetten sollten, die den ersten selectInput (nach dem Beispiel also input$scope1C) beobachtet.
Als nächtes müssen wir ein reaktives Objekt erstellen, das anhand des Inputs von scope1C die Auswahlmöglichkeiten für scope2C ermittelt und abspeichert. Dieses Objekt nennen wir beispielsweise choices. Mithilfe einer Wenn-Dann-Funktion weisen wir dann diesem Element die richtigen Auswahlmöglichkeiten zu. Dafür reduzieren wir jeweils zuerst die Daten auf den gewünschten Kontinent bzw. die WHO-Regionen und nutzen dann die unique-Funktion, um alle möglichen Ausprägungen der COUNTRY-Variable des gefilterten Datensatzes zu erhalten. Durch die return-Funktion werden diese Ländernamen dann aus der reactive-Funktion “zurückgemeldet” und als choices abgespeichert.
Im Folgenden können wir dann den uiOutput erstellen, indem wir der richtigen Output ID (-> output$Choices) mit renderUI ein reaktives selectInput zuweisen. Hierbei ist es wichtig, dass das zuvor erstellte reaktive Objekt choices nur als Funktion aufgerufen werden kann (choices()).
Die fertige Funktion für den reaktiven Input im User Interface sieht dann folgendermaßen aus:
observeEvent(input$scope1C, {
choices <- reactive({
if(input$scope1C == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices <- renderUI({
selectInput(
inputId = "scope2C",
label = "Scope of the Data",
choices = c("Choose a country/a region!" = "", choices()),
selectize = TRUE
)
})
})Die anderen 6 uiOutput werden fast identisch erstellt und hier deshalb nicht direkt gezeigt. Falls du damit trotzdem Probleme hast, kannst du einfach den folgenden Unterabschnitt aufklappen. Dort werden diese ohne weitere Erläuterungen aufgelistet.
Die fehlenden sechs
uiOutputs
observeEvent(input$scope1D1, {
choices1 <- reactive({
if(input$scope1D1 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices1 <- renderUI({
selectInput(
inputId = "scope2D1",
label = "Scope of the Data 1",
choices = c("Choose a country/a region!" = "", choices1()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D2, {
choices2 <- reactive({
if(input$scope1D2 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices2 <- renderUI({
selectInput(
inputId = "scope2D2",
label = "Scope of the Data 2",
choices = c("Choose a country/a region!" = "", choices2()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D3, {
choices3 <- reactive({
if(input$scope1D3 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices3 <- renderUI({
selectInput(
inputId = "scope2D3",
label = "Scope of the Data 3",
choices = c("Choose a country/a region!" = "", choices3()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D4, {
choices4 <- reactive({
if(input$scope1D4 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices4 <- renderUI({
selectInput(
inputId = "scope2D4",
label = "Scope of the Data 4",
choices = c("Choose a country/a region!" = "", choices4()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D5, {
choices5 <- reactive({
if(input$scope1D5 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices5 <- renderUI({
selectInput(
inputId = "scope2D5",
label = "Scope of the Data 5",
choices = c("Choose a country/a region!" = "", choices5()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D6, {
choices6 <- reactive({
if(input$scope1D6 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices6 <- renderUI({
selectInput(
inputId = "scope2D6",
label = "Scope of the Data 6",
choices = c("Choose a country/a region!" = "", choices6()),
selectize = TRUE
)
})
})Das fertiggestellte User Interface unserer App mit vorbereitetem Datensatz und reaktiven Inputs im Server sieht dann folgendermaßen aus:
server <- function(input, output, ...) {
# Pakete laden
library(readxl)
library(tidyverse)
library(ISOcodes)
library(countrycode)
library(plotly)
# Daten einlesen
data1 <- read_excel("GHO-Daten.xlsx", na = "NA", sheet = "Tabelle4")
#Daten formatieren
data1 <- as_tibble(data1)
data2 <- data1 %>%
select_if(function(col) !is.logical(col)) %>%
select(-c(TimeDimType, Dim1Type, Date, TimeDimensionValue, TimeDimensionBegin,
TimeDimensionEnd))
names(data2) <- c('DatID', 'VarID', 'SpatialDimType', 'COUNTRYCODE', 'YEAR', 'SEX',
'LIFE_EXPECTANCY','L_E2EXACT', 'COMMENT')
data2 <- data2[-c(1:48), ] #Ruanda ist doppelt
# Neue Variablen erstellen: 1. Ländername, 2. Kontinent
for (i in 1:9438){
if (data2$COUNTRYCODE[i] == "AFR"){data2$COUNTRY[i] <- "African Region"}
else if (data2$COUNTRYCODE[i] == "AMR"){data2$COUNTRY[i] <- "Region of the Americas"}
else if (data2$COUNTRYCODE[i] == "EMR"){data2$COUNTRY[i] <- "Eastern Mediterranean Region"}
else if (data2$COUNTRYCODE[i] == "EUR"){data2$COUNTRY[i] <- "European Region"}
else if (data2$COUNTRYCODE[i] == "GLOBAL"){data2$COUNTRY[i] <- "GLOBAL"}
else if (data2$COUNTRYCODE[i] == "SEAR"){data2$COUNTRY[i] <- "South-East Asian Region"}
else if (data2$COUNTRYCODE[i] == "WPR"){data2$COUNTRY[i] <- "Western Pacific Region"}
else {data2$COUNTRY[i] <- ISO_3166_1$Name[ISO_3166_1$Alpha_3 == data2$COUNTRYCODE[i]]}
}
data2 <- data2 %>% mutate(
CONTINENT = countrycode(
sourcevar = data2$COUNTRY,
origin = "country.name",
destination = "continent",
nomatch = NA))
# Erstellen der Farbfunktion für die Karte
CRP <- colorRampPalette(c('darkred', 'red', 'orange', 'yellow', 'green', 'darkgreen'))
# Reaktive UI-Inhalte
observeEvent(input$scope1C, {
output$Choices <- renderUI({
choices <- reactive({
if(input$scope1C == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
selectInput(
inputId = "scope2C",
label = "Scope of the Data",
choices = c("Choose a country/a region!" = "", choices()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D1, {
choices1 <- reactive({
if(input$scope1D1 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices1 <- renderUI({
selectInput(
inputId = "scope2D1",
label = "Scope of the Data 1",
choices = c("Choose a country/a region!" = "", choices1()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D2, {
choices2 <- reactive({
if(input$scope1D2 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices2 <- renderUI({
selectInput(
inputId = "scope2D2",
label = "Scope of the Data 2",
choices = c("Choose a country/a region!" = "", choices2()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D3, {
choices3 <- reactive({
if(input$scope1D3 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices3 <- renderUI({
selectInput(
inputId = "scope2D3",
label = "Scope of the Data 3",
choices = c("Choose a country/a region!" = "", choices3()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D4, {
choices4 <- reactive({
if(input$scope1D4 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices4 <- renderUI({
selectInput(
inputId = "scope2D4",
label = "Scope of the Data 4",
choices = c("Choose a country/a region!" = "", choices4()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D5, {
choices5 <- reactive({
if(input$scope1D5 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices5 <- renderUI({
selectInput(
inputId = "scope2D5",
label = "Scope of the Data 5",
choices = c("Choose a country/a region!" = "", choices5()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D6, {
choices6 <- reactive({
if(input$scope1D6 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices6 <- renderUI({
selectInput(
inputId = "scope2D6",
label = "Scope of the Data 6",
choices = c("Choose a country/a region!" = "", choices6()),
selectize = TRUE
)
})
})
}Als Testlauf kannst du wieder einfach shinyApp(ui, server) ausführen. Dein User Interface sollte nun abgeschlossen sein. Jetzt fehlt nur noch die Erstellung der reaktiven Abbildungen.
plotly-Abbildungen mit den Inputs zu verbinden, sodass die Abbildungen auf die Inputs reagieren.
Der Server - Teil 2 (Erstellung der Abbildungen)
Allgemein sollen die Abbildungen dann erstellt werden, wenn der Action Button betätigt wurde. Das bedeutet, dass alle renderPlotly-Befehle in observeEvent-Befehle eingebettet werden sollten, die den jeweiligen Action Button beobachten. Da immer nur ein Action Button sichtbar ist, weiß der Server zu jedem Zeitpunkt, welche Abbildung im Output-Fenster angezeigt werden soll (siehe inputId-Frage am Ende des Abschnitts zum User Interface).
Beginnen wir mit der ersten Abbildung: Der Kartendarstellung. Die Daten müssen hier mithilfe der 4 Inputs sex, time1, time2 und scope korrekt gefiltert werden und in einem reaktiven Datensatz abgespeichert werden (reactiveValues als Ausgangswert, rv$data um die gefilterten Daten abzuspeichern). Darüber hinaus muss eine weitere Variable erstellt werden, die die gewünschte scope der Karte beinhaltet (auf Basis des map-Inputs) - geoscope. Danach kann dem output$diagram die reaktive Kartendarstellung (renderPlotly) zugewiesen werden. Hierbei muss man darauf achten, den Datensatz in rv$data umzuändern (auch in der hoverinfo), den title isoliert an der richtigen Stelle einzufügen (isolate()) und unter layout - geo - scope das auf dem map-Input basierende geoscope-Objekt einzusetzen. Das funktionale Endergebnis sieht dann folgendermaßen aus:
# Karte
observeEvent(input$generate, {
##Datenauswahl
rv <- reactiveValues(data = data2)
rv$data <- data2 %>%
filter(SEX == input$sex
& YEAR >= input$time1
& YEAR <= input$time2
& if(input$scope != "World"){CONTINENT == input$scope} else{SpatialDimType == "COUNTRY"})
if(!is.na(input$map)){geoscope <- input$map} else{geoscope <- 'world'}
##Karte erstellen
output$diagram <- renderPlotly({
rv$data %>%
plot_geo() %>%
add_trace(
z = ~L_E2EXACT,
locations = ~COUNTRYCODE,
locationmode = 'ISO-3',
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", rv$data$COUNTRY,
"<br><b>Country-Code:</b> ", rv$data$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(rv$data$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", rv$data$YEAR,
"<br><b>Sex:</b> ", rv$data$SEX,
"<br><b>Continent:</b> ", rv$data$CONTINENT),
hoverinfo = "text",
colors = CRP(20), #sorgt für 20 farbliche Abstufungen - Genauigkeit der Map
zmin = 35, zmax = 85, #sorgt dafür, dass die Legende gleich bleibt
frame = ~YEAR #Animationsvariable
) %>%
animation_opts(1000, easing = "linear", redraw = TRUE) %>% #redraw muss auf TRUE, damit die Karte immer wieder neu bemalt wird
animation_button(
x = 1, xanchor = "right", y = 0.075, yanchor = "bottom" #Position des Play-Buttons
) %>%
animation_slider(
currentvalue = list(prefix = "YEAR: ", font = list(color="darkblue")), #prefix - string der vor Variable über der Leiste abgedruckt werden soll, dahinter die Farbe
y = -0.05, yanchor = "bottom" #Position der Slider-Leiste
) %>%
layout(
geo = list(
scope = geoscope,
showocean = TRUE,
oceancolor = '#80bfff'
),
title = list(
text = isolate(input$title),
font = list(size = 30, color = "darkblue", family = ""),
xref = "paper",
x = 0.5,
yref = "sheet",
yanchor = "bottom", y = 0.9
)
) %>%
colorbar(
title = list(
text = '<b>Life Expectancy</b>',
side = "right"),
y = 0.8
)
})
})Die Erstellung der anderen Abbildungen ist sehr repetitiv, da alle Veränderungen nach dem selben Prinzip wie bei der Kartendarstellung ablaufen. Deshalb werden wir hier darauf verzichten, alle Abbildungen einzeln durchzugehen. Sofern ihr euch den Code für die übrigen Abbildungen trotzdem anschauen wollt, dann könnt ihr einfach den folgenden Unterabschnitt aufklappen.
Reaktive Abbildung 2-5
Abbildung 2 - Diagram (Country Comparison)
observeEvent(input$generateB, {
#Daten auswählen
rv <- reactiveValues(data = data2)
rv$data <- data2 %>%
filter(SEX == input$sexB
& YEAR >= input$time1B
& YEAR <= input$time2B
& if(input$scopeB != "REGION" & input$scopeB != "COUNTRY"){CONTINENT == input$scopeB} else if(input$scopeB == "REGION"){SpatialDimType == input$scopeB} else if(input$scopeB == "COUNTRY"){SpatialDimType == input$scopeB})
#Diagramm erstellen
output$diagram <- renderPlotly({
plot_ly(
rv$data,
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", rv$data$COUNTRY,
"<br><b>Country-Code:</b> ", rv$data$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(rv$data$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", rv$data$YEAR,
"<br><b>Sex:</b> ", rv$data$SEX,
"<br><b>Continent:</b> ", rv$data$CONTINENT),
hoverinfo = "text",
colors = CRP(190)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$titleB),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.95,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
paper_bgcolor = "#eaf2f3"
)
})
})Abbildung 3 - Diagram (Gender Comparison)
observeEvent(input$generateC, {
#Daten auswählen
rv <- reactiveValues(data = data2)
rv$data <- data2 %>%
filter(YEAR >= input$time1C
& YEAR <= input$time2C
& COUNTRY == input$scope2C)
#Diagramm erstellen
output$diagram <- renderPlotly({
plot_ly(
rv$data,
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", rv$data$COUNTRY,
"<br><b>Country-Code:</b> ", rv$data$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(rv$data$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", rv$data$YEAR,
"<br><b>Sex:</b> ", rv$data$SEX,
"<br><b>Continent:</b> ", rv$data$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$titleC),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.95,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
})
})Abbildung 4 - Diagram (Country x Gender Comparison)
observeEvent(input$generateD, {
output$diagram <- renderPlotly({
#Daten auswählen
rv <- reactiveValues(data = data2)
rv$data <- data2 %>%
filter(YEAR >= input$time1D
& YEAR <= input$time2D)
#Diagramm erstellen
p11 <- filter(rv$data, COUNTRY == input$scope2D1) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D1)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D1)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D1)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D1)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D1)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D1)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D1),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p12 <- filter(rv$data, COUNTRY == input$scope2D2) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D2)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D2)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D2)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D2)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D2)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D2)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D2),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p13 <- filter(rv$data, COUNTRY == input$scope2D3) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D3)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D3)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D3)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D3)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D3)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D3)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D3),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p14 <- filter(rv$data, COUNTRY == input$scope2D4) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D4)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D4)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D4)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D4)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D4)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D4)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D4),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p15 <- filter(rv$data, COUNTRY == input$scope2D5) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D5)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D5)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D5)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D5)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D5)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D5)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D5),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p16 <- filter(rv$data, COUNTRY == input$scope2D6) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D6)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D6)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D6)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D6)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D6)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D6)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D6),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
subplot(style(p11, showlegend = FALSE), style(p12, showlegend = FALSE),
style(p13, showlegend = FALSE), style(p14, showlegend = FALSE),
style(p15, showlegend = FALSE), style(p16, showlegend = FALSE),
shareY = TRUE, shareX = TRUE, nrows = 2, heights = c(0.46, 0.46)) %>%
layout(
title = list(
text = isolate(input$titleD),
font = list(size = 30, color = "darkblue", family = ""),
xref = "paper",
x = 0.5,
yref = "paper",
y = 1
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white"
)
})
})Abbildung 5 - Diagram (Country x Country Comparison)
observeEvent(input$generateE, {
#Daten auswählen
rv <- reactiveValues(data = data2)
rv$data <- data2 %>%
filter(YEAR >= input$time1E
& YEAR <= input$time2E
& SEX == input$sexE)
#Diagramm erstellen
output$diagram <- renderPlotly({
p1 <- filter(rv$data, CONTINENT == "Africa") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, CONTINENT == "Africa")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, CONTINENT == "Africa")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, CONTINENT == "Africa")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, CONTINENT == "Africa")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, CONTINENT == "Africa")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, CONTINENT == "Africa")$CONTINENT),
hoverinfo = "text",
colors = CRP(54)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "AFRICA",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p2 <- filter(rv$data, CONTINENT == "Americas") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, CONTINENT == "Americas")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, CONTINENT == "Americas")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, CONTINENT == "Americas")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, CONTINENT == "Americas")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, CONTINENT == "Americas")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, CONTINENT == "Americas")$CONTINENT),
hoverinfo = "text",
colors = CRP(33)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "AMERICAS",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p3 <- filter(rv$data, CONTINENT == "Asia") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, CONTINENT == "Asia")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, CONTINENT == "Asia")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, CONTINENT == "Asia")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, CONTINENT == "Asia")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, CONTINENT == "Asia")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, CONTINENT == "Asia")$CONTINENT),
hoverinfo = "text",
colors = CRP(47)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "ASIA",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p4 <- filter(rv$data, CONTINENT == "Europe") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, CONTINENT == "Europe")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, CONTINENT == "Europe")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, CONTINENT == "Europe")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, CONTINENT == "Europe")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, CONTINENT == "Europe")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, CONTINENT == "Europe")$CONTINENT),
hoverinfo = "text",
colors = CRP(39)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "EUROPE",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p5 <- filter(rv$data, CONTINENT == "Oceania") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, CONTINENT == "Oceania")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, CONTINENT == "Oceania")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, CONTINENT == "Oceania")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, CONTINENT == "Oceania")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, CONTINENT == "Oceania")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, CONTINENT == "Oceania")$CONTINENT),
hoverinfo = "text",
colors = CRP(10)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "OCEANIA",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p6 <- filter(rv$data, SpatialDimType == "REGION") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, SpatialDimType == "REGION")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, SpatialDimType == "REGION")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, SpatialDimType == "REGION")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, SpatialDimType == "REGION")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, SpatialDimType == "REGION")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, SpatialDimType == "REGION")$CONTINENT),
hoverinfo = "text",
colors = CRP(7)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "WHO-Regions",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
subplot(style(p1, showlegend = FALSE), style(p2, showlegend = FALSE),
style(p3, showlegend = FALSE), style(p4, showlegend = FALSE),
style(p5, showlegend = FALSE), style(p6, showlegend = FALSE),
shareY = TRUE, shareX = TRUE, nrows = 2, heights = c(0.46, 0.46)) %>%
layout(
title = list(
text = isolate(input$titleE),
font = list(size = 30, color = "darkblue", family = ""),
xref = "paper",
x = 0.5,
yref = "paper",
y = 1
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white"
)
})
})Jetzt können wir alles im Server zusammenfügen, sodass der fertige Server-Befehl folgendermaßen aussieht:
server <- function(input, output, ...) {
# Pakete laden
library(tidyverse)
library(plotly)
library(readxl)
library(ISOcodes)
library(countrycode)
# Daten einlesen
data1 <- read_excel("GHO-Daten.xlsx", na = "NA", sheet = "Tabelle4")
#Daten formatieren
data1 <- as_tibble(data1)
data2 <- data1 %>%
select_if(function(col) !is.logical(col)) %>%
select(-c(TimeDimType, Dim1Type, Date, TimeDimensionValue, TimeDimensionBegin,
TimeDimensionEnd))
names(data2) <- c('DatID', 'VarID', 'SpatialDimType', 'COUNTRYCODE', 'YEAR', 'SEX',
'LIFE_EXPECTANCY','L_E2EXACT', 'COMMENT')
data2 <- data2[-c(1:48), ] #Ruanda ist doppelt
# Neue Variablen erstellen: 1. Ländername, 2. Kontinent
for (i in 1:9438){
if (data2$COUNTRYCODE[i] == "AFR"){data2$COUNTRY[i] <- "African Region"}
else if (data2$COUNTRYCODE[i] == "AMR"){data2$COUNTRY[i] <- "Region of the Americas"}
else if (data2$COUNTRYCODE[i] == "EMR"){data2$COUNTRY[i] <- "Eastern Mediterranean Region"}
else if (data2$COUNTRYCODE[i] == "EUR"){data2$COUNTRY[i] <- "European Region"}
else if (data2$COUNTRYCODE[i] == "GLOBAL"){data2$COUNTRY[i] <- "GLOBAL"}
else if (data2$COUNTRYCODE[i] == "SEAR"){data2$COUNTRY[i] <- "South-East Asian Region"}
else if (data2$COUNTRYCODE[i] == "WPR"){data2$COUNTRY[i] <- "Western Pacific Region"}
else {data2$COUNTRY[i] <- ISO_3166_1$Name[ISO_3166_1$Alpha_3 == data2$COUNTRYCODE[i]]}
}
data2 <- data2 %>% mutate(
CONTINENT = countrycode(
sourcevar = data2$COUNTRY,
origin = "country.name",
destination = "continent",
nomatch = NA))
CRP <- colorRampPalette(c('darkred', 'red', 'orange', 'yellow', 'green', 'darkgreen'))
# Karte
observeEvent(input$generate, {
##Datenauswahl
rv <- reactiveValues(data = data2)
rv$data <- data2 %>%
filter(SEX == input$sex
& YEAR >= input$time1
& YEAR <= input$time2
& if(input$scope != "World"){CONTINENT == input$scope} else{SpatialDimType == "COUNTRY"})
if(!is.na(input$map)){geoscope <- input$map} else{geoscope <- 'world'}
##Karte erstellen
output$diagram <- renderPlotly({
rv$data %>%
plot_geo() %>%
add_trace(
z = ~L_E2EXACT,
locations = ~COUNTRYCODE,
locationmode = 'ISO-3',
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", rv$data$COUNTRY,
"<br><b>Country-Code:</b> ", rv$data$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(rv$data$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", rv$data$YEAR,
"<br><b>Sex:</b> ", rv$data$SEX,
"<br><b>Continent:</b> ", rv$data$CONTINENT),
hoverinfo = "text",
colors = CRP(20), #sorgt für 20 farbliche Abstufungen - Genauigkeit der Map
zmin = 35, zmax = 85, #sorgt dafür, dass die Legende gleich bleibt
frame = ~YEAR #Animationsvariable
) %>%
animation_opts(1000, easing = "linear", redraw = TRUE) %>% #redraw muss auf TRUE, damit die Karte immer wieder neu bemalt wird
animation_button(
x = 1, xanchor = "right", y = 0.075, yanchor = "bottom" #Position des Play-Buttons
) %>%
animation_slider(
currentvalue = list(prefix = "YEAR: ", font = list(color="darkblue")), #prefix - string der vor Variable über der Leiste abgedruckt werden soll, dahinter die Farbe
y = -0.05, yanchor = "bottom" #Position der Slider-Leiste
) %>%
layout(
geo = list(
scope = geoscope,
showocean = TRUE,
oceancolor = '#80bfff'
),
title = list(
text = isolate(input$title),
font = list(size = 30, color = "darkblue", family = ""),
xref = "paper",
x = 0.5,
yref = "sheet",
yanchor = "bottom", y = 0.9
)
) %>%
colorbar(
title = list(
text = '<b>Life Expectancy</b>',
side = "right"),
y = 0.8
)
})
})
#Diagramm 1 (örtlicher Vergleich)
observeEvent(input$generateB, {
#Daten auswählen
rv <- reactiveValues(data = data2)
rv$data <- data2 %>%
filter(SEX == input$sexB
& YEAR >= input$time1B
& YEAR <= input$time2B
& if(input$scopeB != "REGION" & input$scopeB != "COUNTRY"){CONTINENT == input$scopeB} else if(input$scopeB == "REGION"){SpatialDimType == input$scopeB} else if(input$scopeB == "COUNTRY"){SpatialDimType == input$scopeB})
#Diagramm erstellen
output$diagram <- renderPlotly({
plot_ly(
rv$data,
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", rv$data$COUNTRY,
"<br><b>Country-Code:</b> ", rv$data$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(rv$data$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", rv$data$YEAR,
"<br><b>Sex:</b> ", rv$data$SEX,
"<br><b>Continent:</b> ", rv$data$CONTINENT),
hoverinfo = "text",
colors = CRP(190)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$titleB),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.95,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
paper_bgcolor = "#eaf2f3"
)
})
})
#Diagramm 2 (Geschlechtervergleich)
observeEvent(input$scope1C, {
choices <- reactive({
if(input$scope1C == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1C == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
output$Choices <- renderUI({
selectInput(
inputId = "scope2C",
label = "Scope of the Data",
choices = c("Choose a country/a region!" = "", choices()),
selectize = TRUE
)
})
})
observeEvent(input$generateC, {
#Daten auswählen
rv <- reactiveValues(data = data2)
rv$data <- data2 %>%
filter(YEAR >= input$time1C
& YEAR <= input$time2C
& COUNTRY == input$scope2C)
#Diagramm erstellen
output$diagram <- renderPlotly({
plot_ly(
rv$data,
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", rv$data$COUNTRY,
"<br><b>Country-Code:</b> ", rv$data$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(rv$data$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", rv$data$YEAR,
"<br><b>Sex:</b> ", rv$data$SEX,
"<br><b>Continent:</b> ", rv$data$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$titleC),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.95,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
})
})
#Diagramm 3 (multipler Geschlechtsvergleich) -> subplot
observeEvent(input$scope1D1, {
output$Choices1 <- renderUI({
choices1 <- reactive({
if(input$scope1D1 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D1 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
selectInput(
inputId = "scope2D1",
label = "Scope of the Data 1",
choices = c("Choose a country/a region!" = "", choices1()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D2, {
output$Choices2 <- renderUI({
choices2 <- reactive({
if(input$scope1D2 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D2 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
selectInput(
inputId = "scope2D2",
label = "Scope of the Data 2",
choices = c("Choose a country/a region!" = "", choices2()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D3, {
output$Choices3 <- renderUI({
choices3 <- reactive({
if(input$scope1D3 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D3 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
selectInput(
inputId = "scope2D3",
label = "Scope of the Data 3",
choices = c("Choose a country/a region!" = "", choices3()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D4, {
output$Choices4 <- renderUI({
choices4 <- reactive({
if(input$scope1D4 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D4 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
selectInput(
inputId = "scope2D4",
label = "Scope of the Data 4",
choices = c("Choose a country/a region!" = "", choices4()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D5, {
output$Choices5 <- renderUI({
choices5 <- reactive({
if(input$scope1D5 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D5 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
selectInput(
inputId = "scope2D5",
label = "Scope of the Data 5",
choices = c("Choose a country/a region!" = "", choices5()),
selectize = TRUE
)
})
})
observeEvent(input$scope1D6, {
output$Choices6 <- renderUI({
choices6 <- reactive({
if(input$scope1D6 == "Africa"){
xy <- data2 %>% filter(CONTINENT == "Africa")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Americas"){
xy <- data2 %>% filter(CONTINENT == "Americas")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Asia"){
xy <- data2 %>% filter(CONTINENT == "Asia")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Europe"){
xy <- data2 %>% filter(CONTINENT == "Europe")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "Oceania"){
xy <- data2 %>% filter(CONTINENT == "Oceania")
return(unique(xy$COUNTRY))}
else if(input$scope1D6 == "WHO-Region"){
xy <- data2 %>% filter(SpatialDimType == "REGION")
return(unique(xy$COUNTRY))}
})
selectInput(
inputId = "scope2D6",
label = "Scope of the Data 6",
choices = c("Choose a country/a region!" = "", choices6()),
selectize = TRUE
)
})
})
observeEvent(input$generateD, {
output$diagram <- renderPlotly({
#Daten auswählen
rv <- reactiveValues(data = data2)
rv$data <- data2 %>%
filter(YEAR >= input$time1D
& YEAR <= input$time2D)
#Diagramm erstellen
p11 <- filter(rv$data, COUNTRY == input$scope2D1) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D1)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D1)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D1)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D1)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D1)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D1)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D1),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p12 <- filter(rv$data, COUNTRY == input$scope2D2) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D2)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D2)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D2)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D2)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D2)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D2)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D2),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p13 <- filter(rv$data, COUNTRY == input$scope2D3) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D3)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D3)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D3)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D3)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D3)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D3)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D3),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p14 <- filter(rv$data, COUNTRY == input$scope2D4) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D4)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D4)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D4)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D4)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D4)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D4)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D4),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p15 <- filter(rv$data, COUNTRY == input$scope2D5) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D5)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D5)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D5)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D5)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D5)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D5)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D5),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p16 <- filter(rv$data, COUNTRY == input$scope2D6) %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~SEX,
type = "scatter",
mode = "line",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, COUNTRY == input$scope2D6)$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, COUNTRY == input$scope2D6)$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, COUNTRY == input$scope2D6)$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, COUNTRY == input$scope2D6)$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, COUNTRY == input$scope2D6)$SEX,
"<br><b>Continent:</b> ", filter(rv$data, COUNTRY == input$scope2D6)$CONTINENT),
hoverinfo = "text",
colors = CRP(3)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = isolate(input$scope2D6),
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white",
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
subplot(style(p11, showlegend = FALSE), style(p12, showlegend = FALSE),
style(p13, showlegend = FALSE), style(p14, showlegend = FALSE),
style(p15, showlegend = FALSE), style(p16, showlegend = FALSE),
shareY = TRUE, shareX = TRUE, nrows = 2, heights = c(0.46, 0.46)) %>%
layout(
title = list(
text = isolate(input$titleD),
font = list(size = 30, color = "darkblue", family = ""),
xref = "paper",
x = 0.5,
yref = "paper",
y = 1
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white"
)
})
})
#Diagramm 4 (ötlicher Vergleich - 5 Kontinente + WHO-Regionen) -> subplot
observeEvent(input$generateE, {
#Daten auswählen
rv <- reactiveValues(data = data2)
rv$data <- data2 %>%
filter(YEAR >= input$time1E
& YEAR <= input$time2E
& SEX == input$sexE)
#Diagramm erstellen
output$diagram <- renderPlotly({
p1 <- filter(rv$data, CONTINENT == "Africa") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, CONTINENT == "Africa")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, CONTINENT == "Africa")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, CONTINENT == "Africa")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, CONTINENT == "Africa")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, CONTINENT == "Africa")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, CONTINENT == "Africa")$CONTINENT),
hoverinfo = "text",
colors = CRP(54)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "AFRICA",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p2 <- filter(rv$data, CONTINENT == "Americas") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, CONTINENT == "Americas")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, CONTINENT == "Americas")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, CONTINENT == "Americas")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, CONTINENT == "Americas")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, CONTINENT == "Americas")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, CONTINENT == "Americas")$CONTINENT),
hoverinfo = "text",
colors = CRP(33)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "AMERICAS",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p3 <- filter(rv$data, CONTINENT == "Asia") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, CONTINENT == "Asia")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, CONTINENT == "Asia")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, CONTINENT == "Asia")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, CONTINENT == "Asia")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, CONTINENT == "Asia")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, CONTINENT == "Asia")$CONTINENT),
hoverinfo = "text",
colors = CRP(47)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "ASIA",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p4 <- filter(rv$data, CONTINENT == "Europe") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, CONTINENT == "Europe")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, CONTINENT == "Europe")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, CONTINENT == "Europe")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, CONTINENT == "Europe")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, CONTINENT == "Europe")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, CONTINENT == "Europe")$CONTINENT),
hoverinfo = "text",
colors = CRP(39)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "EUROPE",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p5 <- filter(rv$data, CONTINENT == "Oceania") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, CONTINENT == "Oceania")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, CONTINENT == "Oceania")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, CONTINENT == "Oceania")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, CONTINENT == "Oceania")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, CONTINENT == "Oceania")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, CONTINENT == "Oceania")$CONTINENT),
hoverinfo = "text",
colors = CRP(10)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "OCEANIA",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
p6 <- filter(rv$data, SpatialDimType == "REGION") %>%
plot_ly(
x = ~YEAR,
y = ~L_E2EXACT,
color = ~COUNTRY,
type = "scatter",
mode = "lines+markers",
text = paste0("<b><i>Data from the Global Health Observatory (GHO)</i></b>",
"<br><br><b>Country:</b> ", filter(rv$data, SpatialDimType == "REGION")$COUNTRY,
"<br><b>Country-Code:</b> ", filter(rv$data, SpatialDimType == "REGION")$COUNTRYCODE,
"<br><b>Life Expectancy:</b> ", round(filter(rv$data, SpatialDimType == "REGION")$L_E2EXACT, digits = 3),
"<br><b>Year:</b> ", filter(rv$data, SpatialDimType == "REGION")$YEAR,
"<br><b>Sex:</b> ", filter(rv$data, SpatialDimType == "REGION")$SEX,
"<br><b>Continent:</b> ", filter(rv$data, SpatialDimType == "REGION")$CONTINENT),
hoverinfo = "text",
colors = CRP(7)) %>%
layout(
legend = list(
font = list(
family = "sans-serif",
size = 12,
color = "#000"),
bgcolor = "#E2E2E2",
bordercolor = "#0a0a0a",
borderwidth = 2,
yanchor = "center", # use center of legend as anchor
y = 0.5), # put legend in center of y-axis,
annotations = list(
text = "WHO-Regions",
font = list(
family = "Arial, Helvetica, sans-serif",
size = 18,
color = "black"),
xref = "paper",
yref = "paper",
yanchor = "bottom",
xanchor = "center",
align = "center",
x = 0.5,
y = 0.92,
showarrow = FALSE
),
xaxis = list(
title = "Time (in Years)",
gridcolor = "white",
gridwidth = 0,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
),
yaxis = list(
title = "Life Expectancy",
gridcolor = "#eaf2f3",
gridwidth = 1,
tickcolor = "black",
tickwidth = 1,
linecolor = "black",
linewidth = 1
)
)
subplot(style(p1, showlegend = FALSE), style(p2, showlegend = FALSE),
style(p3, showlegend = FALSE), style(p4, showlegend = FALSE),
style(p5, showlegend = FALSE), style(p6, showlegend = FALSE),
shareY = TRUE, shareX = TRUE, nrows = 2, heights = c(0.46, 0.46)) %>%
layout(
title = list(
text = isolate(input$titleE),
font = list(size = 30, color = "darkblue", family = ""),
xref = "paper",
x = 0.5,
yref = "paper",
y = 1
),
paper_bgcolor = "#eaf2f3",
plot_bgcolor = "white"
)
})
})
}Wenn du jetzt shinyApp(ui, server) ausführst, solltest du eine voll funktionsfähige App erhalten, die anhand deiner eigenen Inputs verschiedene Abbildungen anzeigen kann.
Anmerkung: Wie weit du bei deiner eigenen App gehst, ist dir völlig selbst überlassen. Die von uns vorgestellte Lösung soll dir nur zeigen, dass alle mit plotly erstellten Abbildung durch shiny unterstützt werden und durch ein User Interface direkt manipulierbar sein können. Probiere dich gerne auch an anderen Abbildungen aus. Falls du Interesse hast, mehr mit shiny zu üben, schau doch gerne auch mal in Projekt 7 rein. Dieses beschäftigt sich ausschließlich mit shiny-Apps.
