Regression und Ausreißerdiagnostik
Einleitung
In der Einführungssitzung hatten wir etwas über das Einlesen von Datensätzen, einfache Deskriptivstatistiken und den \(t\)-Test gelernt und in diesem Rahmen einige Grundlagen der Statistik wiederholt. Nun wollen wir mit etwas komplexeren, aber bereits bekannten, Methoden weitermachen und eine multiple Regression in R durchführen. Hierbei werden wir auch die zu diesem Verfahren notwendigen Voraussetzungen prüfen sowie das Vorliegen von Ausreißern untersuchen.
Bevor wir dazu die Daten einlesen, sollten wir als erstes die nötigen R-Pakete laden. Die R-Pakete, die wir im Folgenden brauchen, sind: das car-Paket, das MASS-Paket sowie das Paket mit dem Namen lm.beta. Diese Pakete müssen zunächst installiert werden. Dies können Sie via install.packages machen:
install.packages("car") # Die Installation ist nur einmalig von Nöten!
install.packages("lm.beta") # Sie müssen nur zu Update-Zwecken erneut installiert werden.
install.packages("MASS")Anschließend werden Pakete mit der library-Funktion geladen:
library(lm.beta) # Standardisierte beta-Koeffizienten für die Regression
library(car) # Zusätzliche Funktion für Diagnostik von Datensätzen
library(MASS) # Zusätzliche Funktion für Diagnostik von Datensätzen Daten einladen
Der Datensatz ist der selbe, wie in der Einführungssitzung: Eine Stichprobe von 100 Schülerinnen und Schülern hat einen Lese- und einen Mathematiktest, sowie zusätzlich einen allgemeinen Intelligenztest, bearbeitet. Im Datensatz enthalten ist zudem das Geschlecht (female, 0=m, 1=w). Sie können den Datensatz “Schulleistungen.rda” hier herunterladen.
Nun müssen wir mit load die Daten laden. Liegt der Datensatz bspw. auf dem Desktop, so müssen wir den Dateipfad dorthin legen und können dann den Datensatz laden (wir gehen hier davon aus, dass Ihr PC “Musterfrau” heißt) Tipp: Verwenden Sie unbedingt die automatische Vervollständigung von R-Studio, wie in der letzten Sitzung beschrieben.
load("C:/Users/Musterfrau/Desktop/Schulleistungen.rda")Genauso sind Sie in der Lage, den Datensatz direkt aus dem Internet zu laden. Hierzu brauchen Sie nur die URL und müssen R sagen, dass es sich bei dieser um eine URL handelt, indem Sie die Funktion url auf den Link anwenden. Der funktionierende Befehl sieht so aus (wobei die URL in Anführungszeichen geschrieben werden muss):
load(url("https://pandar.netlify.app/post/Schulleistungen.rda"))Nun sollte in R-Studio oben rechts in dem Fenster unter der Rubrik “Data” unser Datensatz mit dem Namen “Schulleistungen” erscheinen.
Ein Überblick über die Daten
Wir können uns die ersten (6) Zeilen des Datensatzes mit der Funktion head ansehen. Dazu müssen wir diese Funktion auf den Datensatz (das Objekt) Schulleistungen anwenden:
head(Schulleistungen)## female IQ reading math
## 1 1 81.77950 449.5884 451.9832
## 2 1 106.75898 544.8495 589.6540
## 3 0 99.14033 331.3466 509.3267
## 4 1 111.91499 531.5384 560.4300
## 5 1 116.12682 604.3759 659.4524
## 6 0 106.14127 308.7457 602.8577Wir erkennen die 4 Spalten mit dem Geschlecht, dem IQ, der Lese- und der Mathematikleistung.
Das Folgende fundiert zum Teil auf Sitzungen zur Korrelation und Regression aus Veranstaltungen aus dem Bachelorstudium zur Statistik Vertiefung.
(Multiple-) Lineare Regression
Eine Wiederholung der Regressionsanalyse (und der Korrelation) finden Sie bspw. in Eid, Gollwitzer und Schmitt (2017) Kapitel 16 bis 19, Agresti und Finlay (2013), Kapitel 9 bis 11 und Pituch und Stevens (2016) in Kapitel 3.
Das Ziel einer Regression besteht darin, die Variation einer Variable durch eine oder mehrere andere Variablen vorherzusagen (Prognose und Erklärung). Die vorhergesagte Variable wird als Kriterium, Regressand oder auch abhängige Variable (AV) bezeichnet und üblicherweise mit \(y\) symbolisiert. Die Variablen zur Vorhersage der abhängigen Variablen werden als Prädiktoren, Regressoren oder unabhängige Variablen (UV) bezeichnet und üblicherweise mit \(x\) symbolisiert. Die häufigste Form der Regressionsanalyse ist die lineare Regression, bei der der Zusammenhang über eine Gerade bzw. eine (Hyper-)Ebene beschrieben wird. Demzufolge kann die lineare Beziehung zwischen den vorhergesagten Werten und den Werten der unabhängigen Variablen mathematisch folgendermaßen beschrieben werden:
\[y_i = b_0 +b_{1}x_{i1} + ... +b_{m}x_{im} + e_i\]
- Ordinatenabschnitt/ \(y\)-Achsenabschnitt/ Konstante/ Interzept \(b_0\):
- Schnittpunkt der Regressionsgeraden mit der \(y\)-Achse
- Erwartung von y, wenn alle UVs den Wert 0 annehmen
- Regressionsgewichte \(b_{1},\dots, b_m\):
- beziffern die Steigung der Regressionsgeraden
- Interpretation: die Steigung der Geraden lässt erkennen, um wie viele Einheiten \(y\) zunimmt, wenn (das jeweilige) x um eine Einheit zunimmt (unter Kontrolle aller weiteren Variablen im Modell)
- Regressionsresiduum (kurz: Residuum), Residualwert oder Fehlerwert \(e_i\):
- die Differenz zwischen einem beobachteten und einem vorhergesagten y-Wert (\(e_i=y_i-\hat{y}_i\))
- je größer die Fehlerwerte (betraglich), umso größer ist die Abweichung (betraglich) eines beobachteten vom vorhergesagten Wert
In Matrixschreibweise sieht die Gleichung folgendermaßen aus
\[Y = X\mathbf{b} + \mathbf{e},\]
was wiederum eine Kurzschreibweise für
\[\underbrace{\begin{pmatrix}y_1 \\ \vdots \\ y_n \end{pmatrix}}_Y = \underbrace{\begin{pmatrix}1 & x_{i1} & \dots & x_{m1}\\ \vdots & \vdots & \ddots & \vdots \\ 1 & x_{n1} & \dots & x_{nm} \end{pmatrix}}_X\underbrace{\begin{pmatrix}b_1\\ \vdots\\ b_m\end{pmatrix}}_\mathbf{b} + \underbrace{\begin{pmatrix}e_1 \\ \vdots \\ e_n \end{pmatrix}}_\mathbf{e}\]
ist. Hier steht \(n\) für die Stichprobengröße und \(m\) für die Anzahl an Prädiktoren. Wenn Sie also Ihre Daten in folgender Form vorliegen haben, so können Sie R nutzen und mit Hilfe von Matrixoperationen die Regressionskoeffizienten mit Hilfe des Kleinste-Quadrate-Schätzer berechnen:
\[\hat{\mathbf{b}}=(X'X)^{-1}X'Y\]
Wie dies genau gemacht wird und wie weitere Kennwerte in der Regression (bspw. \(R^2\)) “zu Fuß” bestimmt werden, können Sie im Appendix A nachlesen - hierbei ist zu beachten, dass Appendix A als “weiterführende Literatur” anzusehen und somit nicht verpflichtend ist. Oft hilft es aber beim Verständnis, sich solche Rechenoperationen anzusehen. Außerdem kann es etwas die Komplexität aus der Sache nehmen, wenn erkannt wird, dass wir bspw. die Regressionskoeffizienten durch einfache Matrixprodukte schätzen können.
Unser Modell und das Lesen von R-Outputs
Wir wollen mit Hilfe eines Regressionsmodells die Leseleistung durch das Geschlecht und durch die Intelligenz vorhersagen. Dies funktioniert in R ganz leicht mit der lm ("linear modeling) Funktion. Dieser müssen wir zwei Argumente übergeben: 1) unsere angenommene Beziehung zwischen den Variablen; 2) den Datensatz, in welchem die Variablen zu finden sind:
lm(reading ~ 1 + female + IQ, data = Schulleistungen)##
## Call:
## lm(formula = reading ~ 1 + female + IQ, data = Schulleistungen)
##
## Coefficients:
## (Intercept) female IQ
## 88.209 38.470 3.944Hierbei zeigt die Tilde (~) auf, welche Variable die AV ist (sie steht links der Tilde), welche die UVs sind (sie stehen rechts der Tilde und werden durch + getrennt) und ob das Interzept mitgeschätzt werden soll (per Default wird dieses mitgeschätzt, was bedeutet, dass “1 +” redundant ist und daher hier weggelassen werden könnte - nicht mit einbezogen wird das Interzept via “0 +”). Diese Notation wird in sehr vielen Modell verwendet, in welchen es um die Beziehung zwischen unabhängigen und abhängigen Variablen geht! Im Appendix B können Sie nachlesen, welche weiteren Befehle zum gleichen Ergebnis führen und wie bspw. explizit das Interzept in die Gleichung mit aufgenommen werden kann (oder darauf verzichtet wird!).
Im Output sehen wir die Parameterschätzungen unseres Regressionsmodells:
\[\text{Reading}_i=b_0+b_1\text{Female}_i+b_2\text{IQ}_i+\varepsilon_i,\]
für \(i=1,\dots,100=:n\). Wir wollen uns die Ergebnisse unserer Regressionsanalyse noch detaillierter anschauen. Dazu können wir wieder die summary Funktion anwenden. Um auch die standardisierten Ergebnisse zu erhalten, verwenden wir die Funktion lm.beta (lm steht hier für lineares Modell und beta für die standardisierten Koeffizienten. Achtung: Häufig werden allerdings auch unstandardisierten Regressionskoeffizienten als \(\beta\)s bezeichnet, sodass darauf stets zu achten ist, bspw. in Appendix D dieser Sitzung).
Der lm Befehl erzeugt ein Objekt, welches wir weiterverwenden können, um darauf zum Beispiel die Funktion summary auszuführen. Wir speichern dieses Objekt ab, indem wir eine Zuordnung durchführen via <- und einen Namen vergeben. Sind wir doch mal besonders phantasievoll und nennen unser Modell: “our_model”.
our_model <- lm(reading ~ 1 + female + IQ, data = Schulleistungen)
summary(our_model)##
## Call:
## lm(formula = reading ~ 1 + female + IQ, data = Schulleistungen)
##
## Residuals:
## Min 1Q Median 3Q Max
## -208.779 -64.215 -0.211 58.652 174.254
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) 88.2093 56.5061 1.561 0.1218
## female 38.4705 17.3863 2.213 0.0293 *
## IQ 3.9444 0.5529 7.134 1.77e-10 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 86.34 on 97 degrees of freedom
## Multiple R-squared: 0.3555, Adjusted R-squared: 0.3422
## F-statistic: 26.75 on 2 and 97 DF, p-value: 5.594e-10Funktionen werden immer “von innen nach außen” angewandt, was ganz einfach bedeutet, dass die innerste Funktion zuerst angewandt wird und ein Objekt erzeugt, auf welches anschließend die nächst äußere Funktion angewandt werden kann. Bspw. können wir, bevor wir die Summary anfordern, lm.beta aus dem gleichnamigen Paket auf das Objekt anwenden, um zusätzlich standardisierte Koeffizienten zu erhalten:
summary(lm.beta(our_model))##
## Call:
## lm(formula = reading ~ 1 + female + IQ, data = Schulleistungen)
##
## Residuals:
## Min 1Q Median 3Q Max
## -208.779 -64.215 -0.211 58.652 174.254
##
## Coefficients:
## Estimate Standardized Std. Error t value Pr(>|t|)
## (Intercept) 88.2093 0.0000 56.5061 1.561 0.1218
## female 38.4705 0.1810 17.3863 2.213 0.0293 *
## IQ 3.9444 0.5836 0.5529 7.134 1.77e-10 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 86.34 on 97 degrees of freedom
## Multiple R-squared: 0.3555, Adjusted R-squared: 0.3422
## F-statistic: 26.75 on 2 and 97 DF, p-value: 5.594e-10Seit der R Version 4.1.0 gibt es den sogenannten Pipe-Operator |> mit welchem das Schachteln von Funktionen vereinfacht dargestellt werden kann. Die soll bei vielen nacheinander Anwendnungen von Funktionen helfen, den Überblick zu bewahren. So können wir mit der Pipe obigen Output auch wie folgt erhalten:
our_model |> lm.beta() |> summary()##
## Call:
## lm(formula = reading ~ 1 + female + IQ, data = Schulleistungen)
##
## Residuals:
## Min 1Q Median 3Q Max
## -208.779 -64.215 -0.211 58.652 174.254
##
## Coefficients:
## Estimate Standardized Std. Error t value Pr(>|t|)
## (Intercept) 88.2093 0.0000 56.5061 1.561 0.1218
## female 38.4705 0.1810 17.3863 2.213 0.0293 *
## IQ 3.9444 0.5836 0.5529 7.134 1.77e-10 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 86.34 on 97 degrees of freedom
## Multiple R-squared: 0.3555, Adjusted R-squared: 0.3422
## F-statistic: 26.75 on 2 and 97 DF, p-value: 5.594e-10Der Pipe-Operator übergibt immer das resultiernde Objekt des vorherigen Befehls an die erste Stelle der folgenden Funktion. Somit können wir die Pipe wie folgt lesen: "Nehme our_model und wende darauf lm.beta() and, nehme anschließend das resultierende Objekt und wende darauf summary() an. Je nach dem was Ihnen besser gefällt, können Sie beide Arten verwenden, um in R Funktionen anzuwenden. Wir behalten uns vor beide Arten zu nutzen.
Die Summary ist eine weiterverbreitete Funktion, die Objekte zusammenfasst und interessante Informationen für uns auf einmal bereitstellt. R Outputs sehen fast immer so aus, weswegen es von unabdingbarer Wichtigkeit ist, dass wir uns mit diesem Output vertraut machen. Im Grunde werden uns alle nötigen Informationen, was in dieser Zusammenfassung steht, durch die Überschrift und Spaltenüberschriften gegeben. Was können wir diesem nun Schritt für Schritt entnehmen?
##
## Call:
## lm(formula = reading ~ 1 + female + IQ, data = Schulleistungen)Fasst noch einmal zusammen, welches Objekt “zusammengefasst” wird. Hier steht sozusagen das zuvor untersuchte lm-Objekt (our_model, bzw. lm.beta(our_model)).
##
## Residuals:
## Min 1Q Median 3Q Max
## -208.779 -64.215 -0.211 58.652 174.254Diese Deskriptivstatistiken (gerundet auf 3 Nachkommastellen) geben uns ein Gefühl für die Datengrundlage: die Überschrift sagt uns, dass es hierbei um die Residuen im Regressionsmodell geht. Min steht für das Minimum (-208.779), 1Q beschreibt das erste Quartil (-64.215); also den Prozentrang von 25% - es liegen 25% darunter und 75% der Werte darüber; Median beschreibt den 50. Prozentrang (-0.211), 3Q beschreibt das 3. Quartil, also Prozentrang 75% (58.652) und Max ist der maximale Wert der Residuen (174.254). Der Mittelwert trägt hier keine Information, da die Residuen immer so bestimmt werden, dass sie im Mittel verschwinden, also ihr Mittelwert bei 0 liegt. Da der Median auch sehr nah an der 0 liegt, zeigt dies, dass die Residuen wahrscheinlich recht symmetrisch verteilt sind. Auch das 1. und 3. Quartil verteilen sich ähnlich (also entgegengesetzte Vorzeichen aber betraglich ähnliche Werte), was ebenfalls für die Symmetrie spricht. Wir können die Residuen unserem our_model Objekt ganz leicht entlocken, indem wir den Befehl resid auf dieses Objekt anwenden oder our_model$residuals tippen. Bspw. ergibt sich dann das 1. Quartil oder der Mittelwert als:
quantile(x = resid(our_model), probs = .25) # .25 = 25% = 25. PR mit der Funktion resid()## 25%
## -64.21544mean(x = our_model$residuals) # Mittelwert mit Referenzierung aus dem lm Objekt "our_model"## [1] 1.048103e-15Mit Hilfe der quantile Funktion können wir beliebige Prozentränge eines Vektors anfordern. Hierbei gibt resid(our_model) (genauso wie our_model$residuals) einen Vektor mit Residuen aus. Indem wir probs z.B. auf .01 setzen, würden wir den 1. Prozentrang (1%) erhalten.
Die Zahl, die beim Mittelwert ausgegeben wird, ist folgendermaßen zu lesen: e-15 steht für \(*10^{-15}=0.000000000000001\) (die Dezimalstelle wird um 15 Stellen nach links verschoben), somit ist 1.048103e-15\(=1.048103*10^{-15}=0.000000000000001048103\approx 0\). Hier kommt in diesem Beispiel nicht exakt 0 heraus, da innerhalb der Berechnungen immer auf eine gewisse Genauigkeit gerundet wird. Diese hängt von der sogennanten Maschinengenauigkeit von R ab. Eine noch höhere Genauigkeit der Darstellung von Zahlen würde einfach zu viel Speicherplatz verbrauchen!
Nun kommen wir zum eigentlich Spannenden, nämlich der Übersicht über die Parameterschätzung (Coefficients). Diese sieht in sehr vielen Analysen sehr ähnlich aus (z.B. logistische Regression oder Multi-Level-Analysen/hierarchische Regression aus diesem Kurs oder konfirmatorische Faktorenanalyse und Strukturgleichungsmodelle aus dem Folgekurs im Sommersemester).
##
## Coefficients:
## Estimate Standardized Std. Error t value Pr(>|t|)
## (Intercept) 88.2093 0.0000 56.5061 1.561 0.1218
## female 38.4705 0.1810 17.3863 2.213 0.0293 *
## IQ 3.9444 0.5836 0.5529 7.134 1.77e-10 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Insgesamt gibt es 6 Spalten, wobei die Standardized-Spalte extra durch das Paket lm.beta angefordert wurde (sie ist also nicht immer in der Summary enthalten). In der ersten Spalte stehen die Variablennamen, die selbsterklärend sein sollten. Die Spalte Estimate zeigt den unstandardisierten Parameter (hier Regressionsgewicht). Z.B. liegt das Interzept \(b_0\) bei 88.2093. Das Partialregressionsgewicht vom Geschlecht \(b_\text{female}\) liegt bei 38.4705. Da Mädchen mit 1 kodiert sind, bedeutet dies: Wenn Mädchen im Vergleich zu Jungen betrachtet werden, so steigt die Leseleistung durchschnittlich um 38.4705 Punkte (“durchschnittlich” in der Interpretation ist enorm wichtig, da es ja noch den Vorhersagefehler gibt). Folglich können wir das Interzept ebenfalls interpretieren: Ein Junge mit einem Intelligenzquotienten von 0 (dieser Wert ist leider unrealistisch, da der IQ hier nicht zentriert wurde - die Effekte von Zentrierung schauen wir uns in der Sitzung zu Multi-Level-Modeling genauer an!) hat eine durchschnittliche Leseleistung von 88.2093.
In der Spalte Standardized stehen die standardisierten Regressionsgewichte. Hier werden die Daten so transformiert, dass sowohl die AV als auch die UVs jeweils Mittelwerte von 0 und Varianzen (bzw. Standardabweichungen) von 1 aufweisen. Das Interzept ist nun nicht länger interessant, da, wenn \(y\) einen Mittelwert von 0 hat und auch die unabhängigen Variablen zentriert sind (also Mittelwerte von 0 haben), dann ist das Interzept gerade jener vorhergesagte Wert für \(y\), der anfällt, wenn alle Prädiktoren den Wert 0 -also ihren Mittelwert - annehmen. Bei der Regression ist es aber so, dass an der Stelle, an der die Prädiktoren ihren Mittelwert annehmen, auch der Mittelwert von \(y\) liegt; hier also der Wert 0. Folglich ist das Interzept im standardisierten Fall immer 0.
Das standardisierte Regressionsgewicht der Intelligenz \(\beta_\text{IQ}\) liegt bei 0.5836, was bedeutet, dass bei einer Erhöhung der Intelligenz um eine Standardabweichung auch die Leseleistung im Mittel (im Durchschnitt) um 0.5836 Standardabweichungen ansteigt. Für die Interpretation des Geschlechts als Prädiktor bringt die Standardisierung eine Erschwerung mit sich. Die beiden Ausprägungen sind nun nicht mehr eine Einheit bzw. Standardabweichung voneinander entfernt. Daher kann man den Vergleich nicht mehr mit einbeziehen. Es lässt sich nur sagen: Steigt die Variable Geschlecht um eine Standardabweichung auf der Dimension zwischen Jungen und Mädchen, so steigt die Leseleistung um durchschnittlich 0.181 Standardabweichungen. Die Standardabweichung einer solchen dichotomen Variablen hängt von der Verteilung der beiden Ausprägungen ab. Sie ist am größten, wenn gleich viele Beobachtungen in den beiden Gruppen vorhanden sind.
Die Spalte Std.Error enthält die Standardfehler. Diese werden in \(t\)-Werte umgerechnet via \(\frac{Est}{SE}\): es wird also die Parameterschätzung durch seinen Standardfehler geteilt und in der nächsten Spalte t value dargestellt. In einigen Summaries wird auch anstelle des \(t\)-Wertes der \(z\)-Wert verwendet. Hierbei ändert sich nichts, nur wird zur Herleitung der \(p\)-Werte eben die \(z\)- anstatt der \(t\)-Verteilung verwendet. Wenn wir uns allerdings an Statistik aus dem Bachelor erinnern, so bemerken wir, dass für große Stichproben die \(t\) und die \(z\)-Verteilung identisch (bzw. sehr nahe beieinander liegend) sind. Als groß gilt hierbei bereits eine Stichprobengröße von 50!
In der Spalte Pr(>|t|) stehen die zugehörigen \(p\)-Werte. “Pr” steht für die Wahrscheinlichkeit (Probability), dass die Teststatistik (hier der \(t\)-Wert) im Betrag ein extremeres Ergebnis aufzeigt, als das Beobachtete. Außerdem bekommen wir noch mit Sternchen angezeigt, auf welchem Signifikanzniveau die einzelnen Parameter statistisch bedeutsam sind.
Zusammenfassend entnehmen wir dem Output, dass das Interzept nicht bedeutsam von 0 verschieden ist - die Effekte der Prädiktoren sind allerdings auf dem 5%-Niveau statistisch signifikant. Dies bedeutet bspw. für die Intelligenz, dass mit einer Irrtumswahrscheinlichkeit von 5% der Regressionsparameter der Intelligenz in der Population nicht 0 ist und somit auch in der Population mit dieser Irrtumswahrscheinlichkeit von einem Effekt zu sprechen ist. Hierbei ist es essentiell, dass sich die statistiche Interpretation immer auf die Population bezieht. Dass ein Koeffizient nicht 0 ist (in der Stichprobe), erkennen wir einfach daran, dass er von 0 abweicht, allerdings kann dieses Ergebnis eben durch Zufall aufgetreten sein - so wie es der Fall beim Interzept ist. Dieses weicht offensichtlich in unserer Stichprobe von 0 ab, allerdings nicht stark genug, als dass wir dies auch für die Population schlussfolgern (auch: schließen/inferieren, desewegen auch Inferenzstatistik/schließende Statistik) würden (mit einer Irrtumswahrscheinlichkeit von 5%).
Regressionskoeffizienten können einzeln signifikant sein, ohne, dass signifikante Anteile der Variation der abhängigen Variable erklärt werden.
##
## Residual standard error: 86.34 on 97 degrees of freedom
## Multiple R-squared: 0.3555, Adjusted R-squared: 0.3422
## F-statistic: 26.75 on 2 and 97 DF, p-value: 5.594e-10Dazu entnehmen wir dem letzten Block den Standardfehler der Residuen (Residual standard error), der im Grunde die Fehlervariation von \(y\) beschreibt, sowie das multiple \(R^2\) (Multiple R-squared), welches anzeigt, dass ca. 35.5% der Variation der Leseleistung auf die Prädiktoren Geschlecht und Intelligenz zurückgeführt werden kann. Dieses Varianzinkrement ist statistisch signifikant, was wir am \(F\)-Test in der letzten Zeile ablesen können. Der \(F\)-Wert (F-statistic) liegt bei 26.75, wobei die Hypothesenfreiheitsgrade hier gerade 2 sind (\(df_h\)) und die Residualfreiheitsgrade bei 97 (\(df_e\)) liegen. Der zugehörige \(p\)-Wert ist deutlich kleiner als 5% und liegt bei \(5.594*10^{-10}\). Dies bedeutet, dass mit einer Irrtumswahrscheinlichkeit von 5% auch in der Population eine Vorhersage der Leseleistung durch Geschlecht und Intelligenz gemeinsam angenommen werden kann (\(R^2\neq0\)). In einem Artikel (oder einer Abschlussarbeit) würden wir zur Untermauerung F(2,97)=26.75, p<.001 in den Fließtext schreiben.
Außerdem könnten wir das Summary Objekt auch unter einem Namen abspeichern und ihm dann weitere Informationen entlocken. Bspw. erhalten wir mit $coefficients die Tabelle der Koeffizienten.
summary_our_model <- summary(lm.beta(our_model))
summary_our_model$coefficients # Koeffiziententabelle## Estimate Standardized Std. Error t value Pr(>|t|)
## (Intercept) 88.20929 0.0000000 56.5060958 1.561058 1.217669e-01
## female 38.47046 0.1810138 17.3863424 2.212683 2.926502e-02
## IQ 3.94442 0.5836188 0.5528999 7.134059 1.765181e-10names(summary_our_model) # weitere mögliche Argumente, die wir erhalten können## [1] "call" "terms" "residuals" "coefficients"
## [5] "aliased" "sigma" "df" "r.squared"
## [9] "adj.r.squared" "fstatistic" "cov.unscaled"summary_our_model$r.squared # R^2## [1] 0.3554865Gleiches können wir mit allen Summary-Objekten auch in späteren Sitzungen machen!
Wenn wir diese Siginfikanzentscheidungen nutzen wollen, um die Effekte in der Population auf diese Weise zu interpretieren, so müssen einige Voraussetzungen erfüllt sein, die zunächst noch geprüft werden müssten. Bspw. nehmen wir für ein Regressionsmodell an, dass die Regressoren lineare Beziehungen mit dem Kriterium aufweisen. Die Personen/Erhebungen sollten unabhängig und identisch verteilt sein (sie sollten aus einer i.i.d. Population stammen, also keinerlei Beziehung untereinander aufweisen und dem gleichen Populationsmodell folgen). Die Residuen innerhalb der Regression werden als normalverteilt und homoskedastisch (also mit gleicher Varianz über alle Ausprägungen der Prädiktoren) angenommen. Nur unter diesen Voraussetzungen lassen sich die Signifikanzentscheidungen überhaupt interpretieren. Außerdem beeinflussen Ausreißer die Schätzungen der Regressionskoeffizienten drastisch.
Prüfen der Voraussetzungen
Dieser Abschnitt fundiert auf der Bachelorsitzung zu Regression III. Die Voraussetzung der Unabhängigkeit und der Gleichverteiltheit ist und bleibt eine Annahme, die wir nicht prüfen können. Wir können jedoch durch das Studiendesign (Randomisierung, Repräsentativität) diese Annahme plausibilisieren. Wir schauen uns als nächstes die Linearitätsannahme an und machen danach mit der Verteilung der Residuen weiter. Zum Schluss schauen wir uns neben der Multikollinearität noch mögliche Ausreißer an:
- Linearität
- Homoskedastizität
- Normalverteilung der Residuen
- Multikollinearität
- Identifikation von Ausreißern und einflussreichen Datenpunkten
Im Folgenden werden wir mit dem unstandardisierten Modell weiterarbeiten, welches wir im Objekt our_model gespeichert hatten.
Vertiefende Literatur zum folgenden Stoff finden wir bspw. in Eid, et al. (2017) in Kapitel 19.13 und Pituch und Stevens (2016) in Kapitel 3.10 - 3.14.
Linearität
Eine grafische Prüfung der partiellen Linearität zwischen den einzelnen Prädiktorvariablen und dem Kriterium kann durch partielle Regressionsplots (engl. Partialplots) erfolgen. Dafür sagen wir in einem Zwischenschritt einen einzelnen Prädiktor durch alle übrigen Prädiktoren im Modell vorher und speichern die Residuen, die sich aus dieser Regression ergeben. Diese kennzeichnen den eigenständigen Anteil, den ein Prädiktor nicht mit den übrigen Prädiktoren gemein hat (er wird also von den übrigen Prädiktoren bereinigt). Dann werden die Residuen aus dieser Vorhersage gegen die Residuen des Kriteriums bei Vorhersage durch den jeweiligen Prädiktor dargestellt. Diese Grafiken können Hinweise auf systematische nicht-lineare Zusammenhänge geben, die in der Modellspezifikation nicht berücksichtigt wurden. Die zugehörige R-Funktion des car Pakets heißt avPlots und braucht als Argument lediglich das spezifizierte Regressionsmodell our_model.
avPlots(model = our_model, pch = 16, lwd = 4) 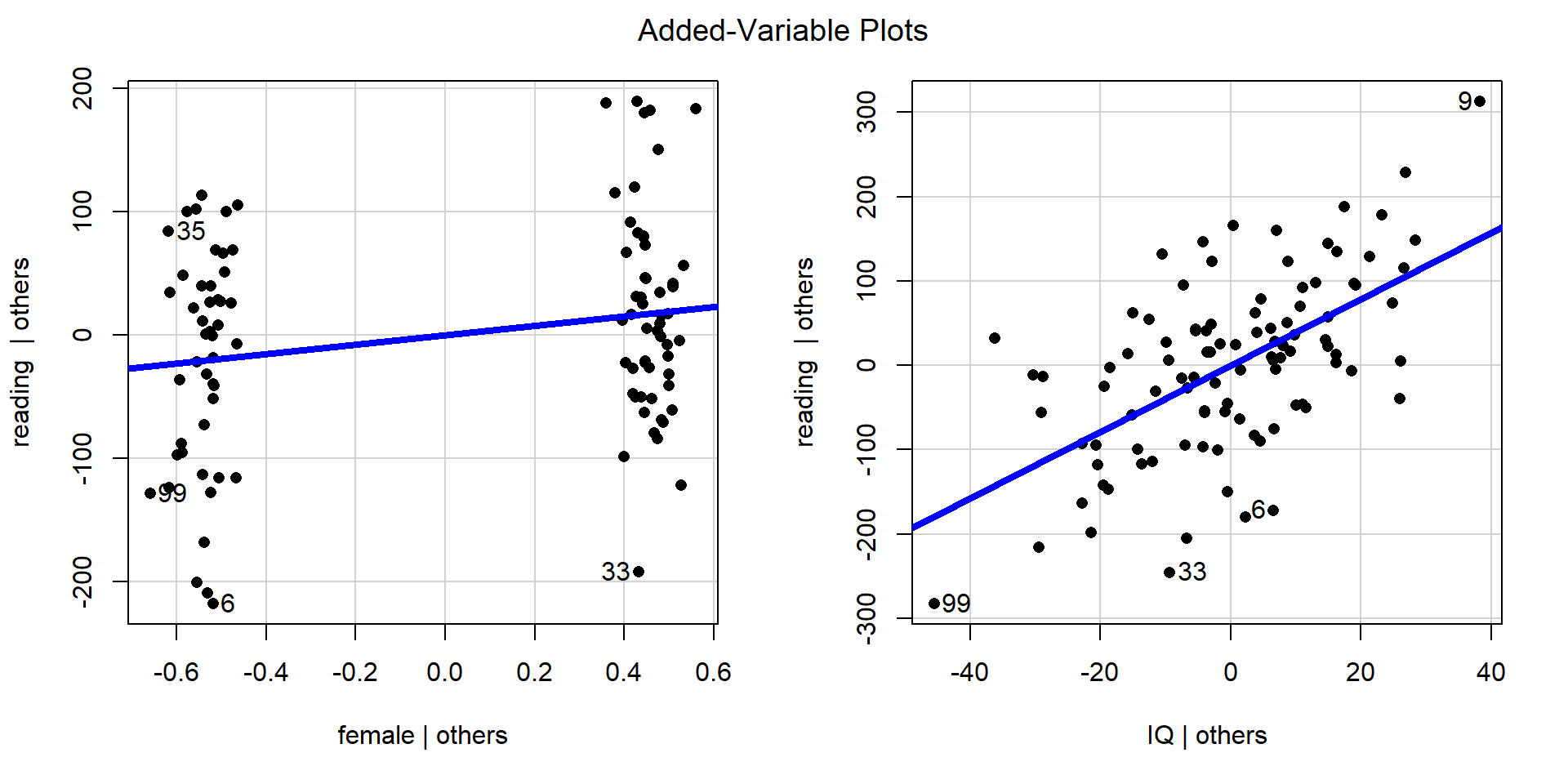
Mit Hilfe der Argumente pch=16 und lwd=4 werden die Darstellung der Punkte (ausgefüllt anstatt leer) sowie die Dicke der Linie (vierfache Dicke) manipuliert (für mehr zu Grafikparametern in R siehe hier). Den Achsenbeschriftungen ist zu entnehmen, dass auf der Y-Achse jeweils reading | others dargestellt ist. Die vertikale Linie | steht hierbei für den mathematischen Ausdruck gegeben. Others steht hierbei für alle weiteren (anderen) Prädiktoren im Modell. Dies bedeutet, dass es sich hierbei um die Residuen aus der Regression von reading auf alle anderen Prädiktoren handelt. Bei den unabhängigen Variablen (UV, female, IQ) steht UV | others also jeweils für die jeweilige UV gegeben der anderen UVs im Modell. Somit beschreiben die beiden Plots jeweils die Beziehungen, die die UVs über die anderen UVs im Modell hinaus mit dem Kriterium (AV, abhängige Variable) haben. Es ist zu beachten, dass die Variable Geschlecht hier nur zwei Ausprägungen hat.
Die hier dargestellten Plots sehen sehr linear aus, weswegen wir nicht an der Liniearitätsannahme zweifeln.
Verteilung der Residuen
Regressionsresiduen sollten homoskedastisch und normalverteilt sein.
Homoskedastizität
Die Varianz der Residuen sollte unabhängig von den Ausprägungen der Prädiktoren sein. Dies wird i.d.R. grafisch geprüft, indem die Residuen \(e_i\) gegen die vorhergesagten Werte \(\hat{y}_i\) geplottet werden: der sogenannte Residuenplot (engl.: residual plot). In diesem Streudiagramm sollten die Residuen gleichmäßig über \(\hat{y}_i\) streuen und keine systematischen Trends (linear, quadratisch, auffächernd, o.ä.) erkennbar sein. Prüfung nicht-linearer Zusammenhänge: Die Funktion residualPlots des Pakets car erzeugt separate Streudiagramme für die Residuen in Abhängigkeit von jedem einzelnen Prädiktor \(x_j\) und von den vorhergesagten Werten \(\hat{y}_i\) (“Fitted Values”); als Input braucht die Funktion das Modell our_model. Zusätzlich wird für jeden Plot ein quadratischer Trend eingezeichnet und auf Signifikanz getestet, wodurch eine zusätzliche Prüfung auf nicht-lineare Effekte erfolgt. Sind diese Tests nicht signifikant, ist davon auszugehen, dass diese Effekte nicht vorliegen und die Voraussetzung der Homoskedastizität damit nicht verletzt ist.
residualPlots(model = our_model, pch = 16)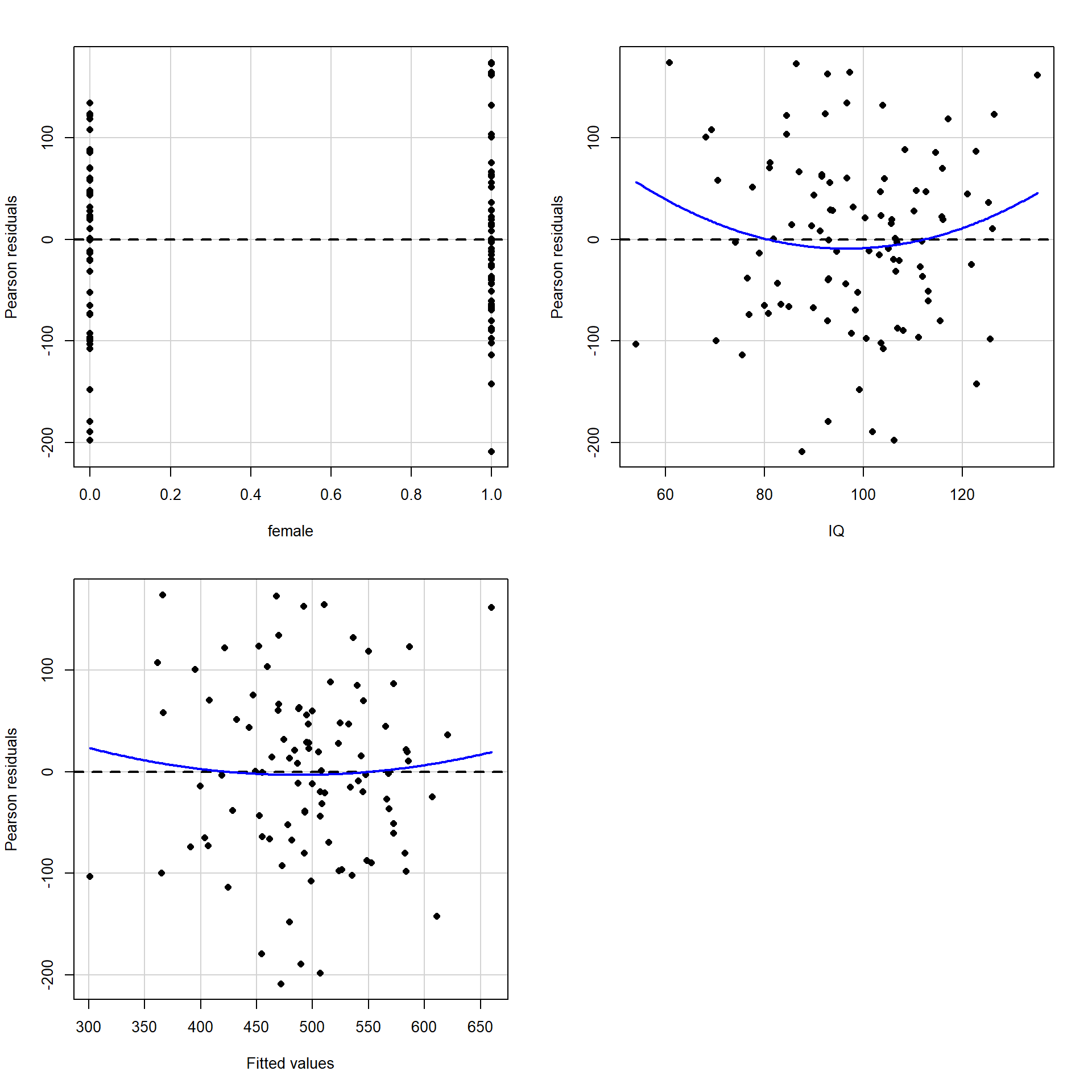
## Test stat Pr(>|Test stat|)
## female 0.0207 0.9835
## IQ 1.4015 0.1643
## Tukey test 0.5234 0.6007Da die Plots alle recht unsystematisch aussehen und die nichtlinearen Effekte nicht signifikant sind, spricht all dies für die Homoskedastizitätsannahme.
Normalverteilung
Voraussetzung für die Signifikanztests im Kontext der linearen Regression ist die Normalverteilung der Residuen. Auch diese Annahme wird i.d.R. grafisch geprüft. Hierfür bietet sich zum einen ein Histogramm der Residuen an, zum anderen ein Q-Q-Diagramm (oder “Quantile-Quantile-Plot”). Zusätzlich zur grafischen Darstellung erlaubt z.B. der Kolmogorov-Smirnov (ks.test) Test eine inferenzstatistische Prüfung, welcher eine deskriptive Verteilung mit einer theoretischen Verteilung vergleicht.
Wir beginnen mit der Vorbereitung der Daten. Wir wollen die studentisierten Residuen grafisch darstellen. Der Befehl studres aus dem MASS Paket speichert die Residuen aus einem lm Objekt, also aus dem definierten Modell our_model, und studentisiert diese. Das Studentisieren der Residuen bezeichnet eine Art der Standardisierung, sodass anschließend der Mittelwert 0 und die Varianz 1 ist (somit lassen sich solche Plots immer gleich interpretieren und besser vergleichen). Mit der Funktion hist erstellen wir ein Histogramm (Zusatzeinstellung freq = F bewirkt, dass wir relative anstatt absoluter Häufigkeiten betrachten). Anschließend müssen wir die Normalverteilung einzeichnen. Hierzu erzeugen wir zunächst eine Sequenz von Zahlen (mit seq), die vom Minimum von res (from = min(res)) bis zum Maximum von res (to = max(res)) in 0.01er-Schritten (by = 0.01) laufen soll. dnorm berechnet die Dichte einer Normalverteilung zu gegebenem x und vorgegebenem Mittelwert und Standardabweichung. Wir wollen die empirische Varianz und den empirischen Mittelwert verwenden und wählen daher dnorm(x = x, mean = mean(res), sd = sd(res)), um die Dichte der Normalverteilung für alle Punkte in x (also vom Minimum bis zum Maximum der studentisierten Residuen res) zu bestimmen (für weitere Informationen siehe bspw. R-Verteilungen auf Wiki
oder
R: Verteilungen auf Data Camp). Mit dem lines Befehl plotten wir anschließend diese Dichte als y-Werte gegen unsere vorgegebenen x-Werte. lwd = 3 bewirkt erneut, dass diese Linie 3 mal so dick wie der Default ausfällt. Außerdem gibt es noch weitere Grafikparameter, wie etwa main, mit dem sich die Grafiküberschrift ändern lässt (probieren Sie es doch selbst aus!). Wer sich mit R schon länger auskennt, und mit dem Paket ggplot2 vertraut ist, kann im Appendix C den Code für die ggplot Funktion dort nachlesen.
res <- studres(our_model) # Studentisierte Residuen als Objekt speichern
hist(res, freq = F)
xWerte <- seq(from = min(res), to = max(res), by = 0.01)
lines(x = xWerte, y = dnorm(x = xWerte, mean = mean(res), sd = sd(res)), lwd = 3)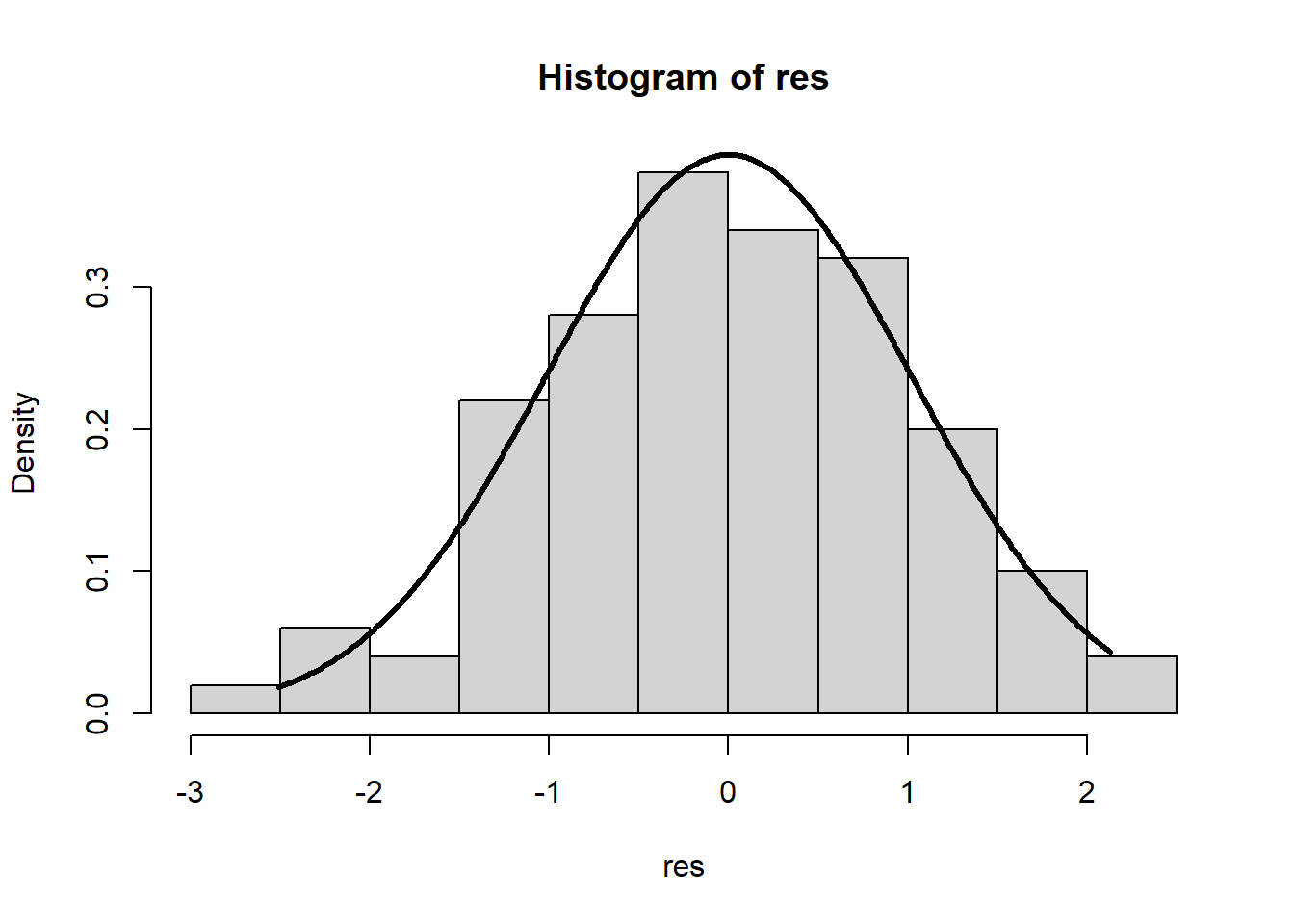
Das Histogramm zeigt keine großen Verstöße gegen die Normalverteilungsannahme.
Ähnliche Informationen sind aus dem Q-Q-Plot zu entnehmen. Dafür kann der Befehl qqPlot aus dem car Paket genutzt werden. Hier kann die Verteilung, gegen die geprüft werden soll, direkt mit distribution festgelegt werden.
qqPlot(our_model, pch = 16, distribution = "norm")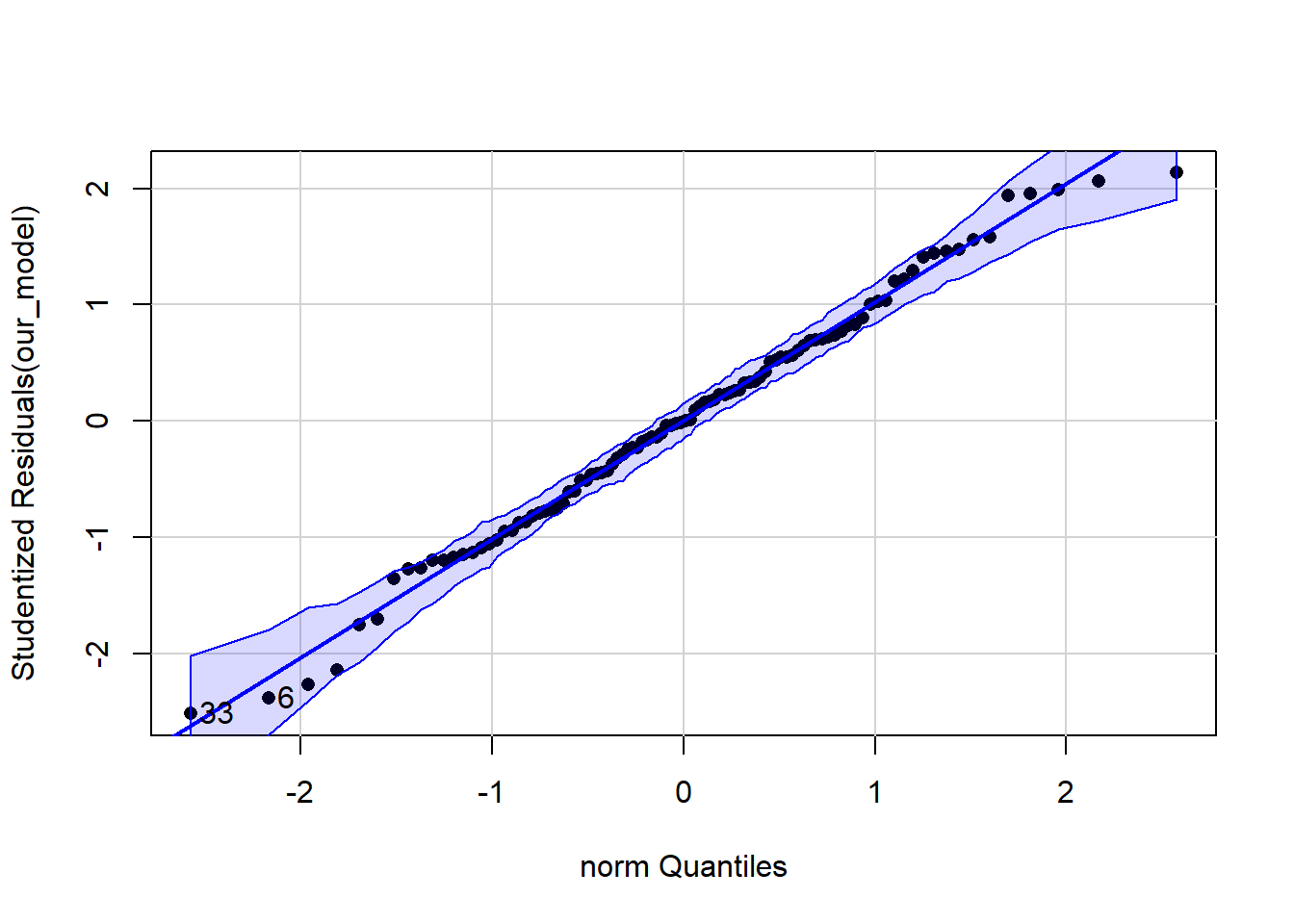
## [1] 6 33Durch die Angabe von distribution = "norm" als Argument für die Funktion qqPlot des car-Pakets wird ein Vergleich der vorliegenden Variable gegen eine Normalverteilung erzeugt (auch der Vergleich zu anderen Verteilungen wäre möglich - die Anführungszeichen sind dabei essentiell. Welche weiteren Verteilungen es gibt, kann bspw. in der oben angegebenen Quelle gefunden werden). Die blaue Fläche zeigt ein “gebootstraptes” (vgl. Eid, et al., 2017, S. 283) 95% Konfidenzintervall an. In diesem Plot sind studentisierte Residuen dargestellt, welche gegen die zugehörigen Qunatile der angenommenen Verteilung abgetragen werden (vgl. Eid, 2017, S. 717 und folgend). Die Punkte sollten möglichst nah an der durchgezogenen Linie (Winkelhalbierende oder Gerade “\(y=x\)”) liegen. Zahlen im Plot zeigen extreme Werte auf (zur Erinnerung, bei einer Standardnormalverteilung mit Mittelwert 0 und Varianz 1 sind Werte ab 1.96 auf dem 5% Niveau extrem - deshalb werden hier die Residuen der Proband*innen 6 und 33 als extrem dargestellt). Diese Zahlen werden auch nochmal in der Console ausgegeben. Insgesamt scheint auch hier keinen Grund zum Zweifeln an der Normalverteilungsannahme zu geben.
Nun wollen wir die Voraussetzung zusätzlich mittels des Kolmogorov Smirnov Tests überprüfen (siehe bspw. Eid, et al., 2017, Kapitel 10.16.1). Dieser testet, wenn wir gegen die Normalverteilung prüfen, im Grunde genommen die Null-Hypothese: \(H_0:\) “Normalverteilung liegt vor”. (Wir wissen allerdings, dass wir Hypothesen nur verwerfen können und nicht wirklich annehmen, weshalb diese Tests immer nur unter Vorbehalt interpretiert werden sollten - Aussagen wie etwa: “\(H_0\) nicht verworfen; daraus folgt, dass in der Population Normalverteilung herrscht”, sind nicht zulässig. Wird die \(H_0\) nicht verworfen, nehmen wir nur weiterhin an, dass eine Normalverteilung vorliegt, ein Beweis ist dies jedoch nicht). Die Funktion ks.test benötigt als Argument die Variable, die auf Normalverteilung geprüft werden soll (hier also x = res); und als weiteres Argument die Vergleichsverteilung - entweder ebenfalls als Vektor (Vergleich von empirischen Verteilungen) oder die theoretisch angenommene Verteilungsfunktion in Anführungszeichen (es sind alle in R implementierten Verteilungen möglich); hier y = "pnorm". In diesem spezifischen Beispiel brauchen wir nun keine weiteren Einstellungen, da die Residuen studentisiert wurden. Normalerweise müssten wir noch die verteilungsspezifischen Parameter vorgeben: hier der Mittelwert und die Standardabweichung. Der vollständige Befehl für alle Normalverteilungen mit dem beobachten Mittelwert und beobachteter Standardabweichung erhalten wir so: ks.test(x = res, y = "pnorm", mean(res), sd(res)), wir könnten dort auch Zahlen vorgeben. Hier geht es aber noch einfacher:
ks.test(x = res, y = "pnorm")##
## One-sample Kolmogorov-Smirnov test
##
## data: res
## D = 0.032672, p-value = 0.9999
## alternative hypothesis: two-sidedDer \(p\)-Wert von 0.9999 zeigt an, dass keine signifikante Diskrepanz zwischen der Normalverteilung und unseren Residuen vorliegt. Für unser Modell zeigen somit alle Ergebnisse übereinstimmend, dass die Residuen normalverteilt sind (bzw. lehnen diese Hypothese nicht ab: Kolmogorov Smirnov Test). Auch die Mahalanobisdistanz kann verwendet werden, um Daten auf (multivariate) Normalverteilung zu prüfen. Dafür muss die Mahalanobisdistanz \(\chi^2\)-verteilt sein, mit der Anzahl an Variablen als \(df\). Dies könnte dann wieder via Q-Q-Plot oder Histogramm erfolgen, bzw. via dem Kolmogorov-Smirnov-Test gegen die \(\chi^2(df)\)-Verteilung. Die Mahalanobisdistanz wird später noch im Zusammenhang mit Ausreißern vorgestellt (dort wird dann auch nochmals die multivariate Normalverteilung aufgegriffen).
Wäre der Kolmogorov Smirnov Test gegen Normalverteilung auf dem 5%-Niveau signifikant, würde dies dafür sprechen, dass “mit einer Irrtumswahrscheinlichkeit von 5% die Null-Hypothese auf Gleichheit der Normalverteilung mit unserer empirischen Verteilung verworfen werden muss, da in der Population die Abweichung der empirischen zur theoretischen Normalverteilung mit der Irrtumswahrscheinlichkeit von 5% ungleich 0 ist”.
Schiefe Verteilungen, welche sich im Histogramm oder im Q-Q-Plot widerspiegeln, sowie zu einem signifikanten Kolmogorov Smirnov Test gegen Normalverteilung führen können, könnten Indizien für nichtlineare (z.B. quadratische) Beziehungen im Datensatz sein. Weitere Analysen wären hierfür nötig.
Multikollinearität
Multikollinearität ist ein potenzielles Problem der multiplen Regressionsanalyse und liegt vor, wenn zwei oder mehrere Prädiktoren hoch miteinander korrelieren. Hohe Multikollinearität
- schränkt die mögliche multiple Korrelation ein, da die Prädiktoren redundant sind und überlappende Varianzanteile in \(y\) erklären.
- erschwert die Identifikation von bedeutsamen Prädiktoren, da deren Effekte untereinander konfundiert sind (die Effekte können schwer voneinander getrennt werden).
- bewirkt eine Erhöhung der Standardfehler der Regressionskoeffizienten (der Standardfehler ist die Standardabweichung zu der Varianz der Regressionskoeffizienten bei wiederholter Stichprobenziehung und Schätzung). Dies bedeutet, dass die Schätzungen der Regressionsparameter instabil, und damit weniger verlässlich, werden. Weitere Informationen zur Instabilität und zu Standardfehlern kann der/die interessierte Leser*in in Appendix D nachlesen.
Multikollinearität kann durch Inspektion der bivariaten Zusammenhänge (Korrelationsmatrix) der Prädiktoren \(x_j\) untersucht werden. Leider kann dies aber nicht alle Formen von Multikollinearität aufdecken. Darüber hinaus ist die Berechnung der sogenannten Toleranz (T) und des Varianzinflationsfaktors (VIF) für jeden Prädiktor möglich. Hierfür wird nacheinander für jeden Prädiktor \(x_j\) der Varianzanteil \(R_j^2\) berechnet, der durch Vorhersage von \(x_j\) durch alle anderen Prädiktoren in der Regression erklärt wird. Toleranz und VIF sind wie folgt definiert:
- \(T_j = 1-R^2_j = \frac{1}{VIF_j}\)
- \(VIF = \frac{1}{1-R^2_j} = \frac{1}{T_j}\)
Offensichtlich genügt eine der beiden Statistiken, da sie vollständig ineinander überführbar und damit redundant sind. Empfehlungen als Grenzwert für Kollinearitätsprobleme sind z. B. \(VIF_j>10\) (\(T_j<0.1\); siehe Eid, et al., 2017, S. 712 und folgend). Die Varianzinflationsfaktoren der Prädiktoren im Modell können mit der Funktion vif des car-Paktes bestimmt werden, der Toleranzwert als Kehrwert des VIFs.
# Korrelation der Prädiktoren
cor(Schulleistungen$female, Schulleistungen$IQ)## [1] -0.08467395Die Prädiktoren sind nur schwach negativ korreliert. Wir schauen uns trotzdem den VIF und die Toleranz an. Dazu übergeben wir wieder das definierte Regressionsmodell an vif.
vif(our_model) # VIF## female IQ
## 1.007221 1.0072211/vif(our_model) # Toleranz## female IQ
## 0.9928303 0.9928303In diesem Beispiel mit nur 2 Prädiktoren ist \(R_j^2=cor(\text{female},\text{IQ})^2\) und die Formeln sind daher sehr einfach auch mit Hand zu bestimmen:
1/(1-cor(Schulleistungen$female, Schulleistungen$IQ)^2) # 1/(1-R^2) = VIF## [1] 1.0072211-cor(Schulleistungen$female, Schulleistungen$IQ)^2 # 1-R^2 = Toleranz## [1] 0.9928303Für unser Modell wird ersichtlich, dass die Prädiktoren praktisch unkorreliert sind und dementsprechend kein Multikollinearitätsproblem vorliegt. Unabhängigkeit folgt hieraus allerdings nicht, da nicht-lineare Beziehungen zwischen den Variablen bestehen könnten, die durch diese Indizes nicht abgebildet werden.
Identifikation von Ausreißern und einflussreichen Datenpunkten
Die Plausibilität unserer Daten ist enorm wichtig. Aus diesem Grund sollten Ausreißer oder einflussreiche Datenpunkte analysiert werden. Diese können bspw. durch Eingabefehler entstehen (Alter von 211 Jahren anstatt 21) oder es sind seltene Fälle (hochintelligentes Kind in einer Normstichprobe), welche so in natürlicher Weise (aber mit sehr geringer Häufigkeit) auftreten können. Es muss dann entschieden werden, ob Ausreißer die Repräsentativität der Stichprobe gefährden und ob diese daher besser ausgeschlossen werden sollten.
Hebelwerte
Hebelwerte \(h_j\) (engl.: leverage values) erlauben die Identifikation von Ausreißern aus der gemeinsamen Verteilung der unabhängigen Variablen, d.h. sie geben an, wie weit ein Wert vom Mittelwert einer Prädiktorvariable entfernt ist. Je höher der Hebelwert, desto weiter liegt der einzelne Fall vom Mittelwert der gemeinsamen Verteilung der unabhängigen Variablen entfernt und desto stärker kann somit der Einfluss auf die Regressionsgewichte sein. Diese werden mit der Funktion hatvalues ermittelt. In R werden die Hebelwerte nicht zentriert ausgegeben. Kriterien zur Beurteilung der nicht zentrierten Hebelwerte variieren, so werden von Eid et al. (2017, S. 707) in Kombination mit Pituch et al. (2016, S. 108). Grenzen von \(2\cdot (k+1) / n\) für große und \(3\cdot (k+1) / n\) für kleine Stichproben vorgeschlagen (mit \(k\) als Anzahl der Variablen). Alternativ zu einem festen Cut-Off-Kriterium kann die Verteilung der Hebelwerte inspiziert werden, wobei diejenigen Werte als kritisch bewertet werden, die aus der Verteilung ausreißen. Dies wird empfohlen, da die Grenzen sehr sensibel sind und viele verdächtige Werte anzeigen. Die Funktion hatvalues erzeugt die Hebelwerte aus einem Regression-Objekt. Wir wollen diese als Histogramm darstellen.
n <- length(residuals(our_model)) # Anzahl an Personen bestimmen
h <- hatvalues(our_model) # Hebelwerte
hist(h, breaks = 20)
abline(v = 2*(2+1)/n, col = "red") # Cut-off als große Stichprobe
abline(v = 3*(2+1)/n, col = "blue") # Cut-off als kleine Stichprobe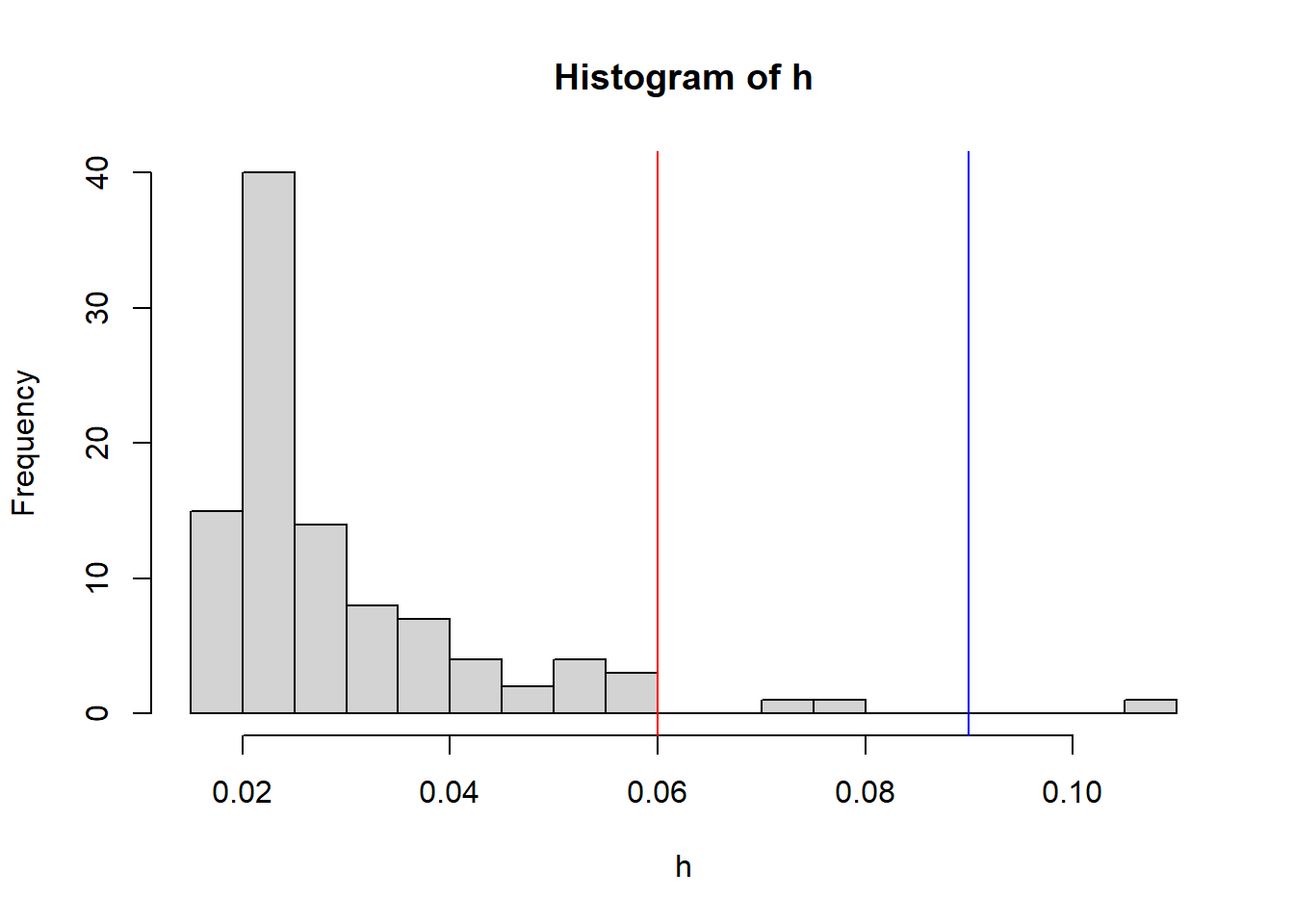
Hier eine kurze Beschreibung aller Argumente in der Grafik: Das Zusatzargument breaks = 20 in hist gibt an, dass 20 Balken gezeichnet werden sollen. abline ist eine Funktion, die eine Gerade einem Plot hinzufügt. Dem Argument v wird hierbei der Punkt übergeben, an welchem eine vertikale Linie eingezeichnet werden soll. col = "red" bzw. col = "blue" gibt an, dass diese Linie rot bzw. blau sein soll.
Cook’s Distanz
Cook’s Distanz \(CD_i\) bezieht sich auf Ausreißer auf der abhängigen Variablen und gibt eine Schätzung an, wie stark sich die Regressionsgewichte verändern, wenn eine Person \(i\) aus dem Datensatz entfernt wird. Fälle, deren Elimination zu einer deutlichen Veränderung der Ergebnisse führen würden, sollten kritisch geprüft werden. Als einfache Daumenregel gilt, dass \(CD_i>1\) auf einen einflussreichen Datenpunkt hinweist. Cook’s Distanz kann mit der Funktion cooks.distance ermittelt werden.
# Cooks Distanz
CD <- cooks.distance(our_model) # Cooks Distanz
hist(CD, breaks = 20)
abline(v = 1, col = "red") # Cut-off bei 1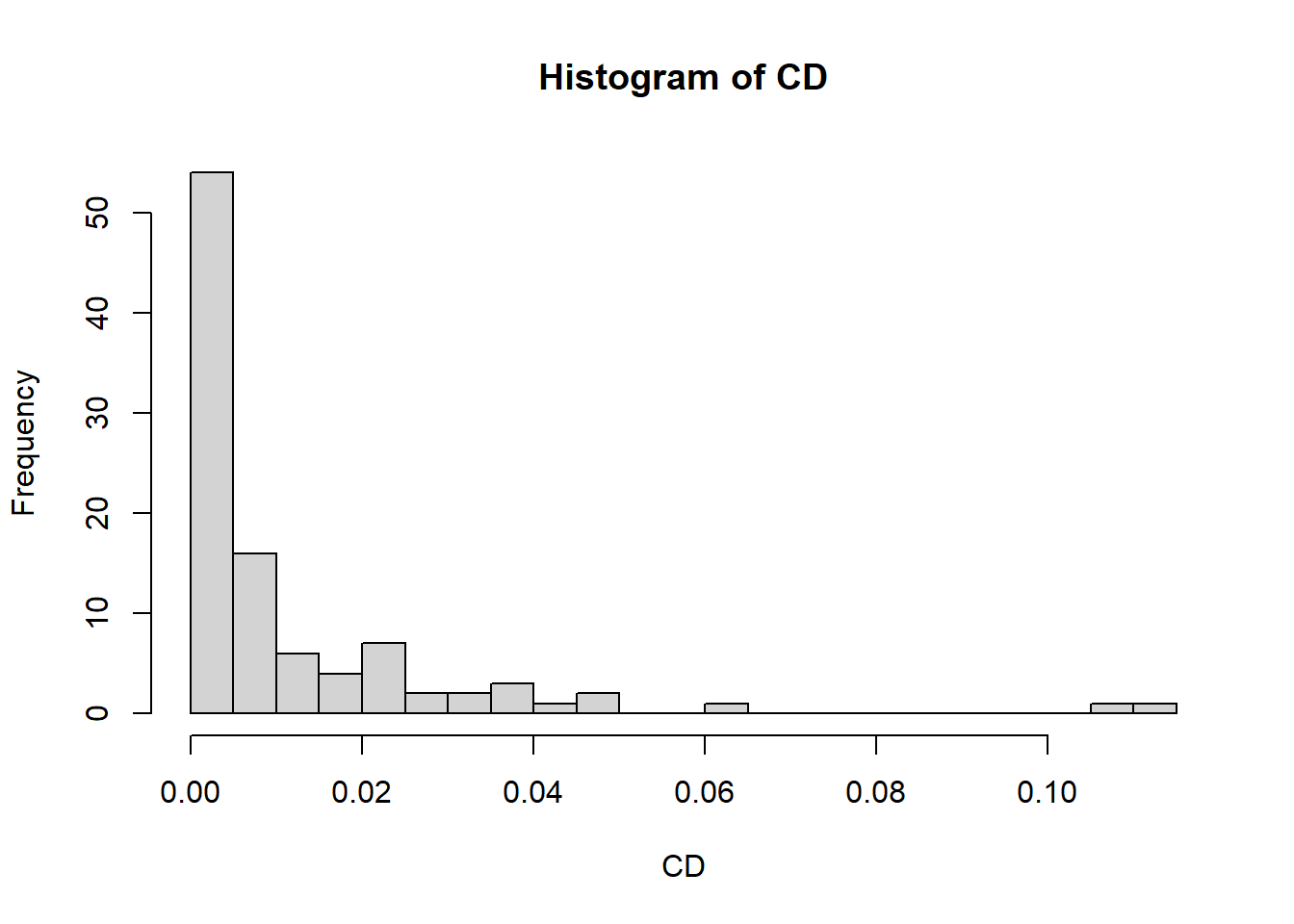 In diesem Plot ist die vertikale Linie nicht enthalten, da der Plot schon zu früh entlang der x-Achse aufhört. Wir können die Grenzen mit
In diesem Plot ist die vertikale Linie nicht enthalten, da der Plot schon zu früh entlang der x-Achse aufhört. Wir können die Grenzen mit xlim = c(0,1) explizit von 0 bis 1 vorgeben:
# Cooks Distanz nochmal
hist(CD, breaks = 20, xlim = c(0, 1))
abline(v = 1, col = "red") # Cut-off bei 1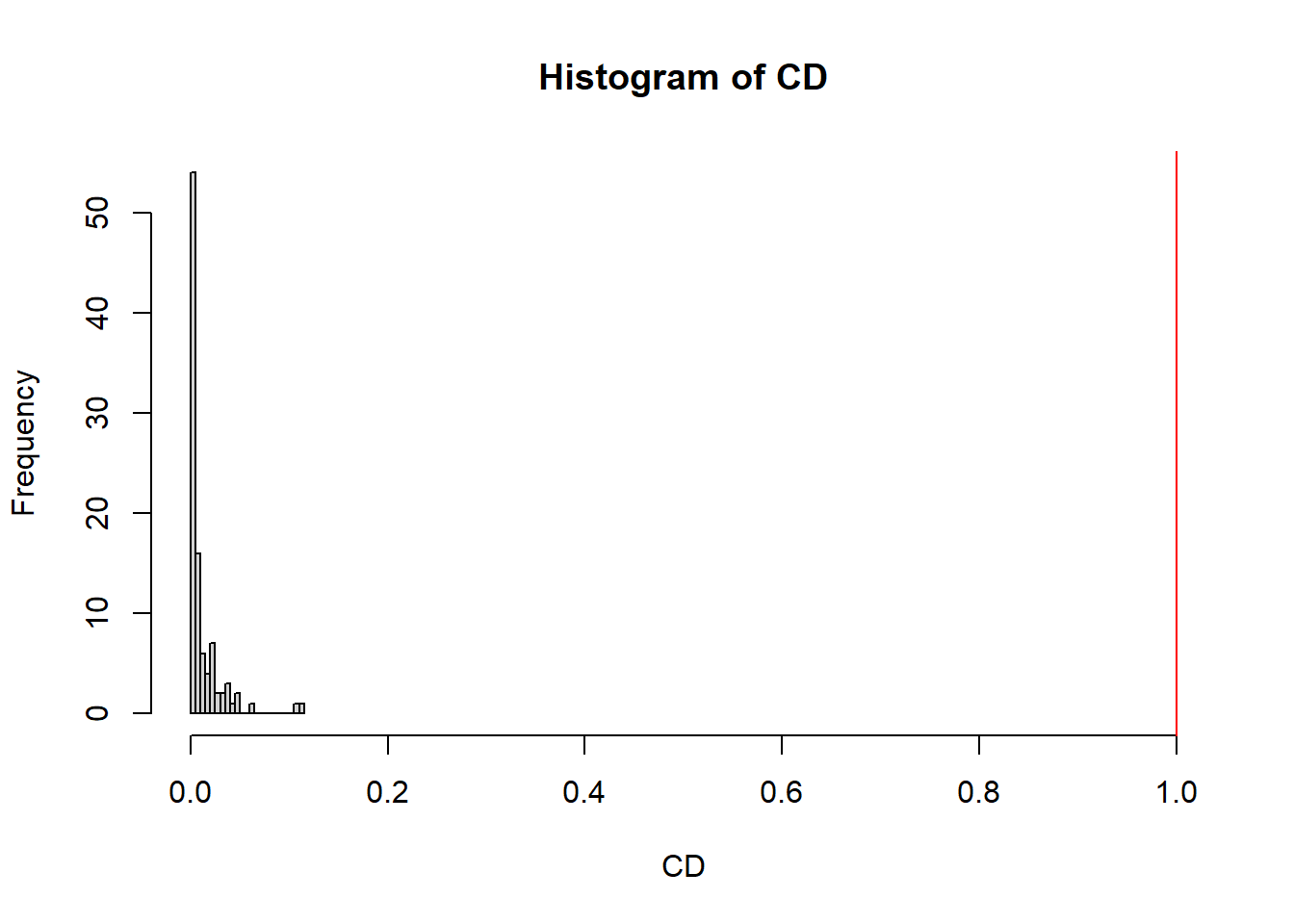
Blasendiagramm
Die Funktion influencePlot des car-Paketes erzeugt ein “Blasendiagramm” zur simultanen grafischen Darstellung von Hebelwerten (auf der x-Achse), studentisierten Residuen (auf der y-Achse) und Cook’s Distanz (als Größe der Blasen). Vertikale Bezugslinien markieren das Doppelte und Dreifache des durchschnittlichen Hebelwertes, horizontale Bezugslinien die Werte -2, 0 und 2 auf der Skala der studentisierten Residuen. Fälle, die nach einem der drei Kriterien als Ausreißer identifiziert werden, werden im Streudiagramm durch ihre Zeilennummer gekennzeichnet. Diese Zeilennummern können verwendet werden, um sich die Daten der auffälligen Fälle anzeigen zu lassen. Sie werden durch InfPlot ausgegeben. Auf diese kann durch as.numeric(row.names(InfPlot)) zugegriffen werden.
# Blasendiagramm mit Hebelwerten, studentisierten Residuen und Cooks Distanz
# In "IDs" werden die Zeilennummern der auffälligen Fälle gespeichert,
# welche gleichzeitig als Zahlen im Blasendiagramm ausgegeben werden
InfPlot <- influencePlot(our_model)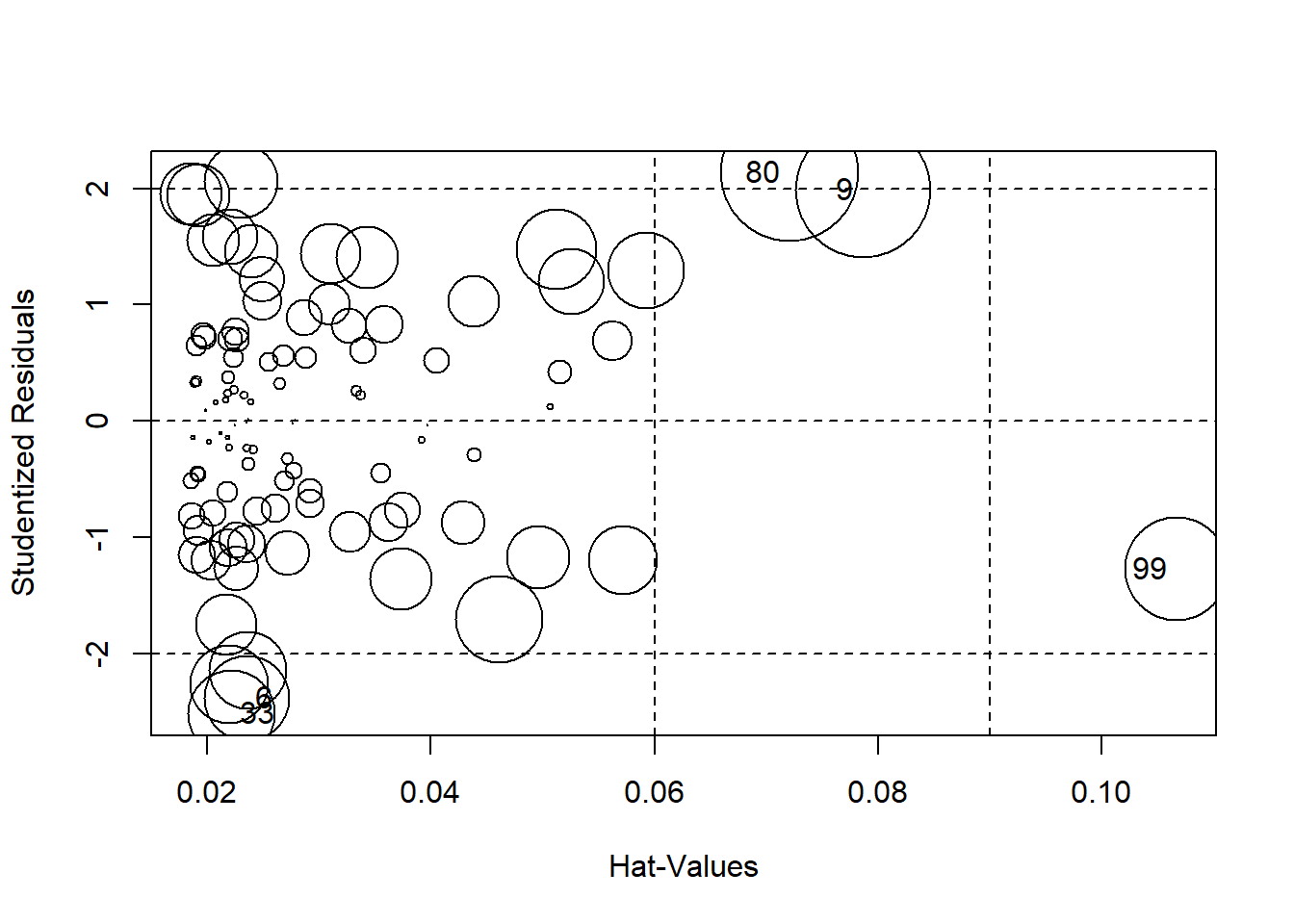
IDs <- as.numeric(row.names(InfPlot))
# Werte der identifizierten Fälle
InfPlot## StudRes Hat CookD
## 6 -2.377185 0.02350872 0.04327384
## 9 1.984692 0.07863226 0.10876003
## 33 -2.511223 0.02211252 0.04506800
## 80 2.133104 0.07208157 0.11365968
## 99 -1.270057 0.10669659 0.06381762Schauen wir uns die möglichen Ausreißer an und standardisieren die Ergebnisse für eine bessere Interpretierbarkeit.
# Rohdaten der auffälligen Fälle (gerundet für bessere Übersichtlichkeit)
round(Schulleistungen[IDs,],2)## female IQ reading math
## 6 0 106.14 308.75 602.86
## 9 1 135.20 822.01 749.56
## 33 1 87.55 263.23 494.10
## 80 1 60.77 540.63 366.73
## 99 0 54.05 198.11 367.98# z-standardisierte Werte der auffälligen Fälle
round(scale(Schulleistungen)[IDs,],2) ## female IQ reading math
## [1,] -1.08 0.51 -1.76 0.35
## [2,] 0.92 2.35 3.06 1.61
## [3,] 0.92 -0.67 -2.19 -0.58
## [4,] 0.92 -2.37 0.42 -1.67
## [5,] -1.08 -2.80 -2.80 -1.66Die Funktion scale z-standardisiert den Datensatz, mit Hilfe von [IDs,], werden die entsprechenden Zeilen der Ausreißer aus dem Datensatz ausgegeben und anschließend auf 2 Nachkommastellen gerundet. Hierbei ist es extrem wichtig, dass wir scale(Schulleistungen)[IDs,] und nicht scale(Schulleistungen[IDs,]) schreiben (das ist mir nämlich schon mal passiert…), da bei der zweiten Schreibweise die Daten reskaliert (z-standardisiert) werden, allerdings auf Basis der ausgewählten Fälle (n=5) und nicht auf Basis der gesamten Stichprobe (n=100). Vielleicht hilft dem einen oder der anderen ja folgende Schreibweise: (Schulleistungen |> scale())[IDs,] mit Hilfe der Pipe. Mit Hilfe der z-standardisierten Ergebnisse lassen sich Ausreißer hinsichtlich ihrer Ausprägungen einordnen:
Interpretation
Was ist an den fünf identifizierten Fällen konkret auffällig?
- Fall 6: unterdurchschnittliche Lesekompetenz bei gleichzeitig durchschnittlichem IQ und durchschnittlicher Matheleistung
- Fall 9: Sehr hohe Werte in IQ, Lese- und Matheleistung
- Fall 33: Unterdurchschnittliche Leseleistung “trotz” eher durchschnittlicher Intelligenz
- Fall 80: Sehr niedriger IQ bei gleichzeitig durchschnittlicher Lesekompetenz
- Fall 99: Sehr niedrige Werte in IQ, Lesekompetenz und Mathematik
Die Entscheidung, ob Ausreißer oder auffällige Datenpunkte aus Analysen ausgeschlossen werden, ist schwierig und kann nicht pauschal beantwortet werden. Im vorliegenden Fall wäre z.B. zu überlegen, ob die Intelligenztestwerte der Fälle 80 und 99, die im Bereich von Lernbehinderung oder sogar geistiger Behinderung liegen, in einer Stichprobe von Schülern/innen aus Regelschulen als glaubwürdige Messungen interpretiert werden können oder als Hinweise auf mangelndes Commitment bei der Beantwortung hinweisen.
Einfluss von Hebelwert und Cook’s Distanz
Was wäre nun gewesen, wenn die Hebelwerte oder Cook’s Distanz extreme Werte angezeigt hätten? Um dieser Frage auf den Grund zu gehen, schauen wir uns für eine Kombination der beiden Koeffizienten den Effekt auf eine Regressionsgerade an. Die vier Grafiken zeigen jeweils die Regressionsgerade in schwarz ohne den jeweiligen Ausreißer, während die Gerade in blau die Regressionsanalyse (Y ~ 1 + X) inklusive des Ausreißers symbolisiert.
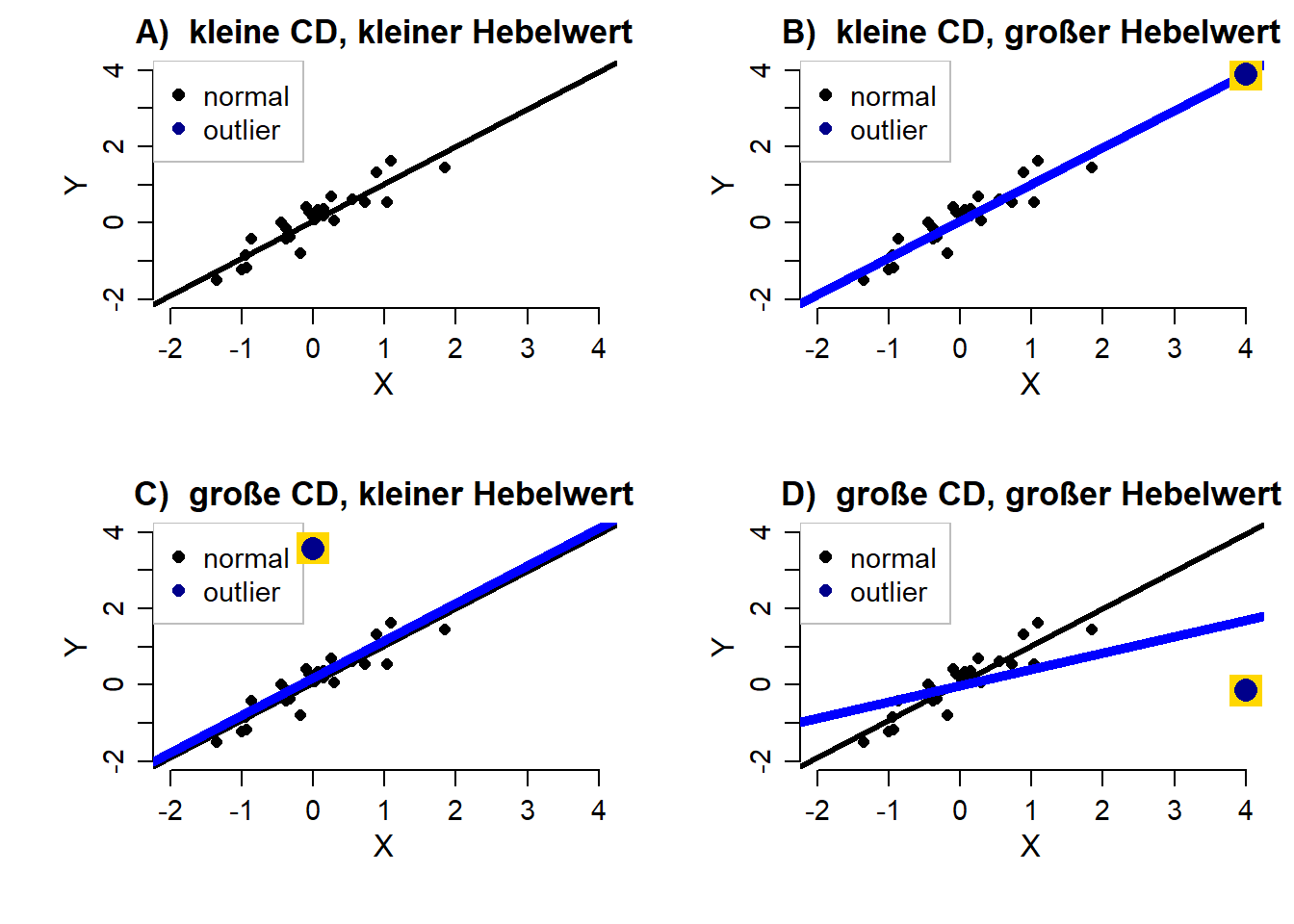
In A) ist die Regression ohne Ausreißer dargestellt. B) zeigt den Effekt, wenn nur der Hebelwert groß ist. Es ist kaum ein Einfluss auf die Regressionsgerade auszumachen. Der Mittelwert der Variable X wird stark nach rechts verschoben. Dies bedeutet, dass ein großer Hebelwert nur den Mittelwert dieser Variable in Richtung des Ausreißers “hebelt”, nicht aber zwangsweise die Regressionsgerade! C) zeigt eine große Cook’s Distanz bei gleichzeitig kleinem Hebelwert. die Gerade ist etwas nach oben verschoben und auch die Steigung hat sich leicht verändert. Insgesamt ist mit dem bloßen Auge allerdings noch kein extremer Effekt auf die Gerade auszumachen. Dieser Effekt wird nur in D) deutlich. Hier ist sowohl Cook’s Distanz als auch der Hebelwert extrem. Dadurch verändert sich die Regressionsgerade stark. Hier könnten wir davon sprechen, dass die Gerade durch den Ausreißer nach unten “gehebelt” wird. Die hier dargestellte Erhebung hat auch auch die größte Mahalanobisdistanz, da sie sowohl in X als auch in Y Richtung extrem ist (siehe dazu nächsten Abschnitt). Insgesamt zeigt diese Grafik, dass nicht ein Koeffizient alleine ausreicht, um einen Effekt auf eine Regressionsanalyse zu untersuchen und dass Werte besonders dann extreme Auswirkungen haben, wenn mehrere Koeffizienten groß sind!
Mahalanobisdistanz
Die Mahalanobisdistanz (siehe z.B. Eid et al., 2017, ab Seite 707) ist ein Werkzeug, welches zur Überprüfung der Normalverteilung und zur Identifikation von multidimensionalen Ausreißern verwendet werden kann. Mit Hilfe der Mahalanobisdistanz wird die Entfernung vom zentralen Zentroiden bestimmt und mit Hilfe der Kovarianzmatrix gewichtet. Im Grunde kann man sagen, dass die Entfernung vom gemeinsamen Mittelwert über alle Variablen an der Variation in den Daten relativiert wird. Im eindimensionalen Fall ist die Mahalanobisdistanz nichts anderes als der quadrierte \(z\)-Wert, denn wir bestimmen dann die Mahalanobisdistanz einer Person \(i\) via \[MD_i=\frac{(X_i-\bar{X})^2}{\sigma_X^2}=\left(\frac{X_i-\bar{X}}{\sigma_X}\right)^2=z^2.\] Wir erkennen, dass wir hier den Personenwert relativ zur Streuung in den Daten betrachten. Nutzen wir nun mehrere Variablen und wollen multivariate Ausreißer interpretieren, so ist die Mahalanobisdistanz folgendermaßen definiert: \[MD_i=(\mathbf{X}_i-\bar{\mathbf{X}})'\Sigma^{-1}(\mathbf{X}_i-\bar{\mathbf{X}}).\] Der Vektor der Mittelwertsdifferenzen \(\mathbf{X}_i-\bar{\mathbf{X}}\) wird durch die Kovarianzmatrix der Daten \(\Sigma\) gewichtet. Sind zwei Variablen \(X_1\) und \(X_2\) positiv korreliert, so treten große Werte (und auch kleine Werte) auf beiden Variablen gemeinsam häufig auf, allerdings sind große \(X_1\) und kleine \(X_2\)-Werte (gleichzeitig und auch umgekehrt) unwahrscheinlich. Dies lässt sich anhand der Mahalanobisdistanz untersuchen. Wann ist nun ein Mahalanobisdistanzwert extrem? Dies können wir uns an einem zweidimensionalen Beispiel klarer machen. Dazu tragen wir in ein Diagramm die Ellipsen (Kurven) gleicher Mahalanobisdistanz ein, also jene Linien, welche laut Mahalanobisdistanz gleich weit vom Zentroiden entfernt liegen. Je dunkler die Kurven, desto weiter entfernt liegen diese Punkte vom Zentroiden (hier \((0,0)\)) und desto unwahrscheinlicher sind diese Punkte in den Daten zu beobachten. In diesem Beispiel nehmen wir an, dass die Variablen positiv korreliert sind:
## Warning: Paket 'ellipse' wurde unter R Version 4.1.1 erstellt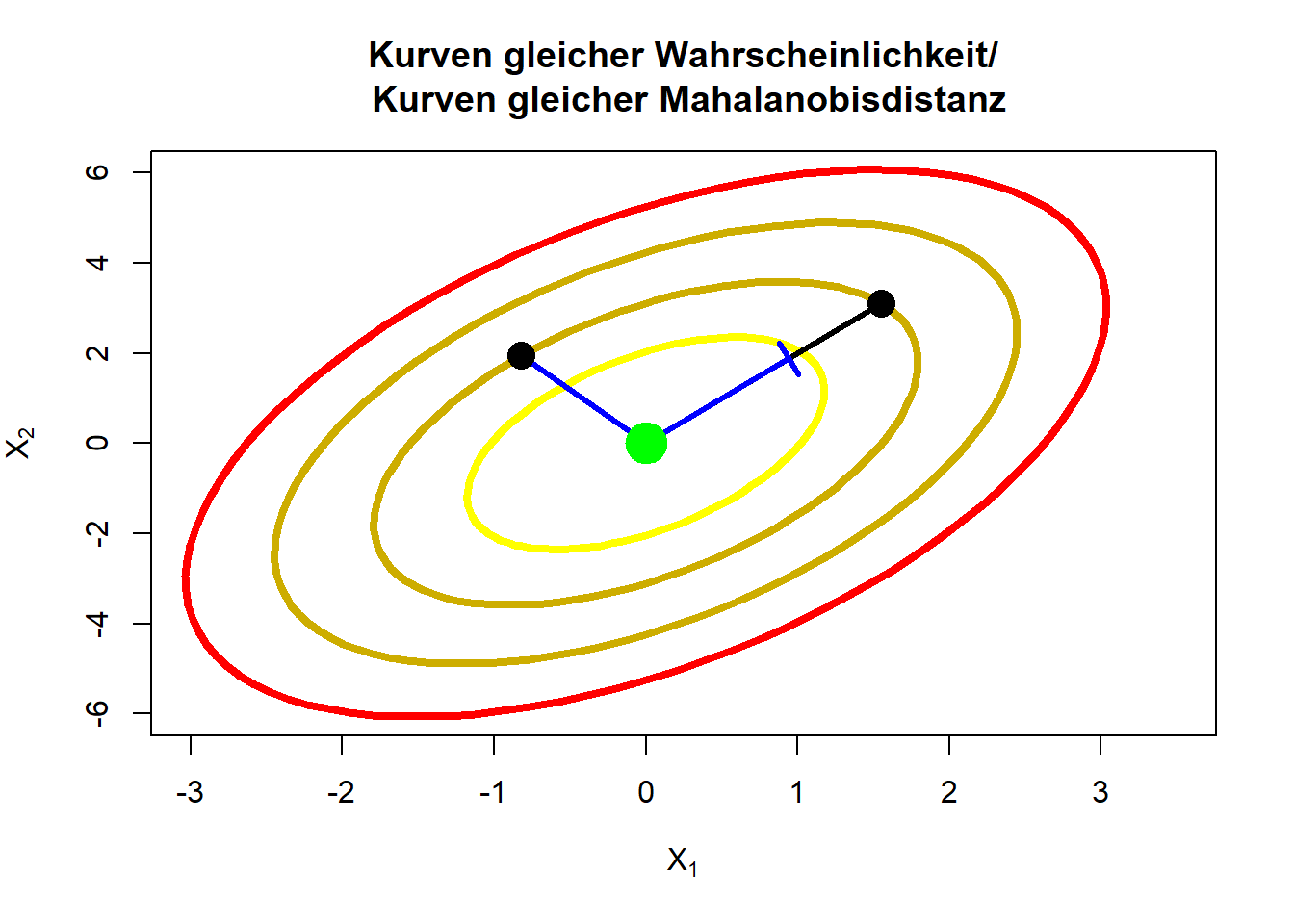 Hier ist
\[\begin{align*}
\mathbf{X}&=\begin{pmatrix}X_1\\ X_2\end{pmatrix}\\
\bar{\mathbf{X}}&=\begin{pmatrix}\bar{X}_1\\ \bar{X}_2\end{pmatrix}=\begin{pmatrix}0\\ 0\end{pmatrix}
\end{align*}\]
Der Zentroid ist hier in Hellgrün dargestellt. Außerdem sind zwei Punkte (in schwarz) eingezeichnet, die die gleiche Mahalanobisdistanz haben. Allerdings sehen wir, repräsentiert durch die blaue Linie, ebenfalls die euklidische Distanz. Die euklidische Distanz ist jene, welche wir nutzen, wenn wir ein Maßband anlegen würden (um bspw. ein Zimmer zu vermessen oder eben die Distanz auf dem Bildschirm der beiden Punkte vom hellgrünen Zentroiden). Dies bedeutet, dass wenn wir in dieser Grafik (und damit in den Daten) die Kovariation der Variablen ignorieren würden, so würden wir den linken schwarzen Punkt und die blaue Linie als äquidistant (also gleich weit entfernt) annehmen. Berücksichtigen wir allerdings die positive Korrelation der Variablen, dann erkennen wir, dass die beiden schwarzen Punkte gleich wahrscheinlich sind und damit im Schnitt gleich häufig auftreten. Dies lässt sich folgendermaßen erklären: wenn zwei Variablen positiv korreliert sind, sind extreme positive und extreme negative Werte auf beiden Variablen gleichzeitig recht wahrscheinlich, während es sehr unwahrscheinlich ist, dass die eine Variable eine hohe und die andere gleichzeitig eine niedrige Ausprägung aufweist (und umgekehrt). Entsprechend haben Wertekonstellationen, die sehr unwahrscheinlich sind (gegeben der Struktur in den Daten) eine große Mahalanobisdistanz - in der Grafik wächst also die Mahalanobisdistanz je dunkler die Kurve.
Hier ist
\[\begin{align*}
\mathbf{X}&=\begin{pmatrix}X_1\\ X_2\end{pmatrix}\\
\bar{\mathbf{X}}&=\begin{pmatrix}\bar{X}_1\\ \bar{X}_2\end{pmatrix}=\begin{pmatrix}0\\ 0\end{pmatrix}
\end{align*}\]
Der Zentroid ist hier in Hellgrün dargestellt. Außerdem sind zwei Punkte (in schwarz) eingezeichnet, die die gleiche Mahalanobisdistanz haben. Allerdings sehen wir, repräsentiert durch die blaue Linie, ebenfalls die euklidische Distanz. Die euklidische Distanz ist jene, welche wir nutzen, wenn wir ein Maßband anlegen würden (um bspw. ein Zimmer zu vermessen oder eben die Distanz auf dem Bildschirm der beiden Punkte vom hellgrünen Zentroiden). Dies bedeutet, dass wenn wir in dieser Grafik (und damit in den Daten) die Kovariation der Variablen ignorieren würden, so würden wir den linken schwarzen Punkt und die blaue Linie als äquidistant (also gleich weit entfernt) annehmen. Berücksichtigen wir allerdings die positive Korrelation der Variablen, dann erkennen wir, dass die beiden schwarzen Punkte gleich wahrscheinlich sind und damit im Schnitt gleich häufig auftreten. Dies lässt sich folgendermaßen erklären: wenn zwei Variablen positiv korreliert sind, sind extreme positive und extreme negative Werte auf beiden Variablen gleichzeitig recht wahrscheinlich, während es sehr unwahrscheinlich ist, dass die eine Variable eine hohe und die andere gleichzeitig eine niedrige Ausprägung aufweist (und umgekehrt). Entsprechend haben Wertekonstellationen, die sehr unwahrscheinlich sind (gegeben der Struktur in den Daten) eine große Mahalanobisdistanz - in der Grafik wächst also die Mahalanobisdistanz je dunkler die Kurve.
Beschäftigen wir uns zunächst mit der Testung auf multivariate Normalverteilung. Wenn diese gegeben ist, sind die Daten der Mahalanobisdistanz \(\chi^2(df=p)\) verteilt ist, wobei \(p\) die Anzahl an Variablen ist. Der Vorteil hiervon ist, dass wir eine eindimensionale Verteilung untersuchen können, um ein Gefühl für multivariate Daten zu erhalten. Bspw. kann dann ein Histogramm oder ein Q-Q-Plot verwendet werden, um die Daten auf Normalverteilung zu untersuchen, bzw. es kann bspw. der Kolmogorov-Smirnov Test durchgeführt werden, um zu prüfen, ob die Mahalanobisdistanz \(\chi^2(df=p)\) verteilt ist.
Der Befehl in R für die Mahalanobisdistanz ist mahalanobis. Wir nehmen einfach die Leseleistung und die Mathematikleistung via Schulleistungen$... als unsere zwei Variablen auf und fassen diese zusammen zu einer Datenmatrix X mit cbind (column-bind), welches die übergebenen Variablen als Spaltenvektoren zusammenfasst (siehe dazu auch Appendix B in der Einführungssitzung). mahalanobis braucht 3 Argumente: x = X die Daten, den gemeinsamen Mittelwert der Daten, den wir hier mit colMeans bestimmen (es wird jeweils der Mittelwert für die Spalten gebildet) sowie die Kovarianzmatrix der Daten mit cov(X) (cor gibt die Korrelationsmatrix aus; hier wird allerdings die Kovarianzmatrix gebraucht- anhand der Korrelationsmatrix lässt sich jedoch die Beziehung der Variablen besser einordnen), an welcher die Struktur relativiert werden soll:
X <- cbind(Schulleistungen$reading, Schulleistungen$math) # Datenmatrix mit Leseleistung in Spalte 1 und Matheleistung in Spalte 2
colMeans(X) # Spaltenmittelwerte (1. Zahl = Mittelwert der Leseleistung, 2. Zahl = Mittelwert der Matheleistung)## [1] 496.0660 561.4645cov(X) # Kovarianzmatrix von Leseleistung und Matheleistung## [,1] [,2]
## [1,] 11333.071 4653.679
## [2,] 4653.679 13639.296cor(X) # zum Vergleich: die Korrelationsmatrix (die Variablen scheinen mäßig zu korrelieren, was unbedingt in die Ausreißerdiagnostik involviert werden muss)## [,1] [,2]
## [1,] 1.0000000 0.3743059
## [2,] 0.3743059 1.0000000MD <- mahalanobis(x = X, center = colMeans(X), cov = cov(X))Außerdem widerspricht die Verteilung der Mahalanobisdistanz nicht (zu sehr) der Annahme auf multivariate Normalverteilung, da das Histogramm einigermaßen zur Dichte der \(\chi^2(df=2)\)-Verteilung passt:
hist(MD, freq = F, breaks = 15)
xWerte <- seq(from = min(MD), to = max(MD), by = 0.01)
lines(x = xWerte, y = dchisq(x = xWerte, df = 2), lwd = 3)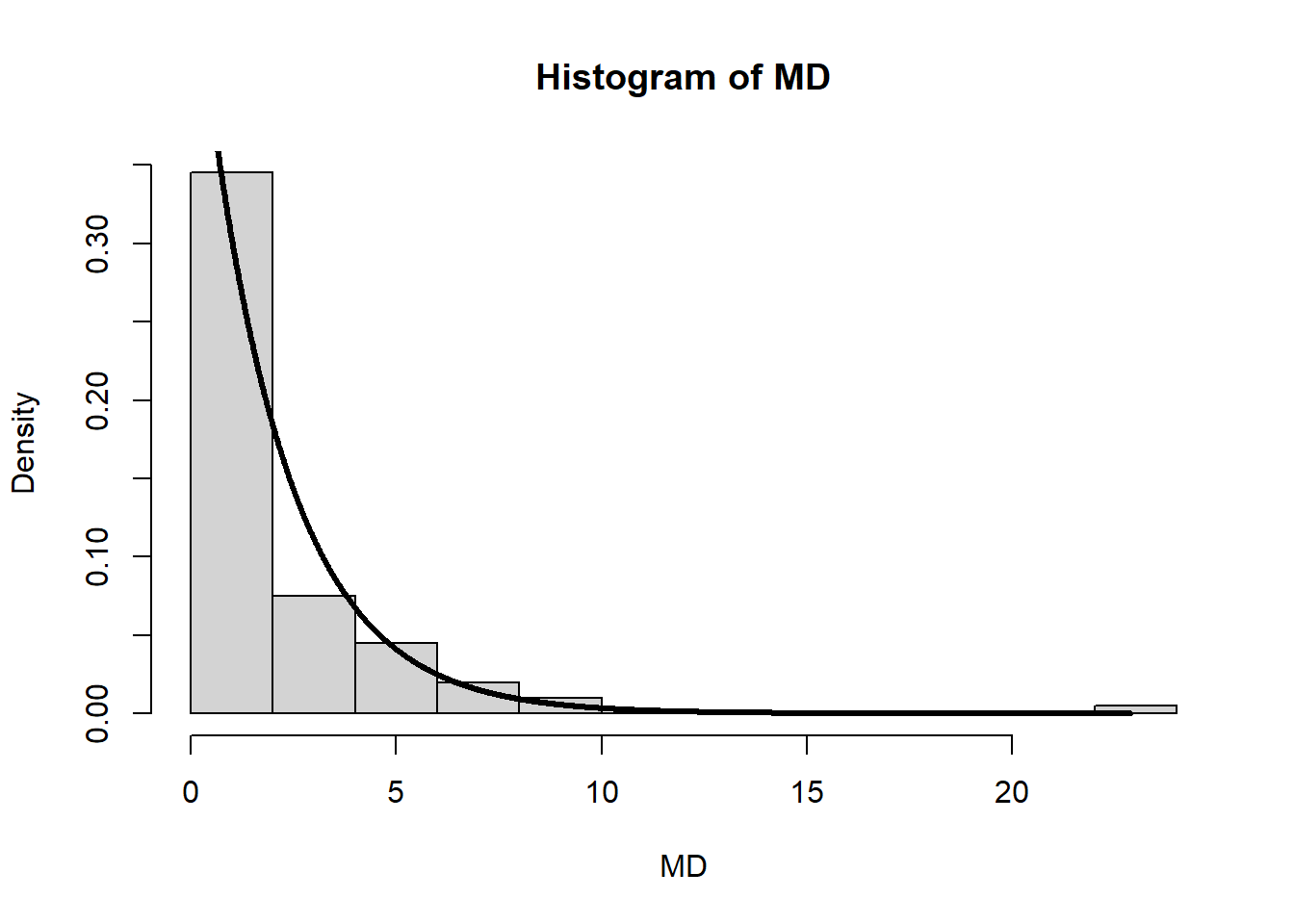
qqPlot(x = MD,distribution = "chisq", df = 2, pch = 16)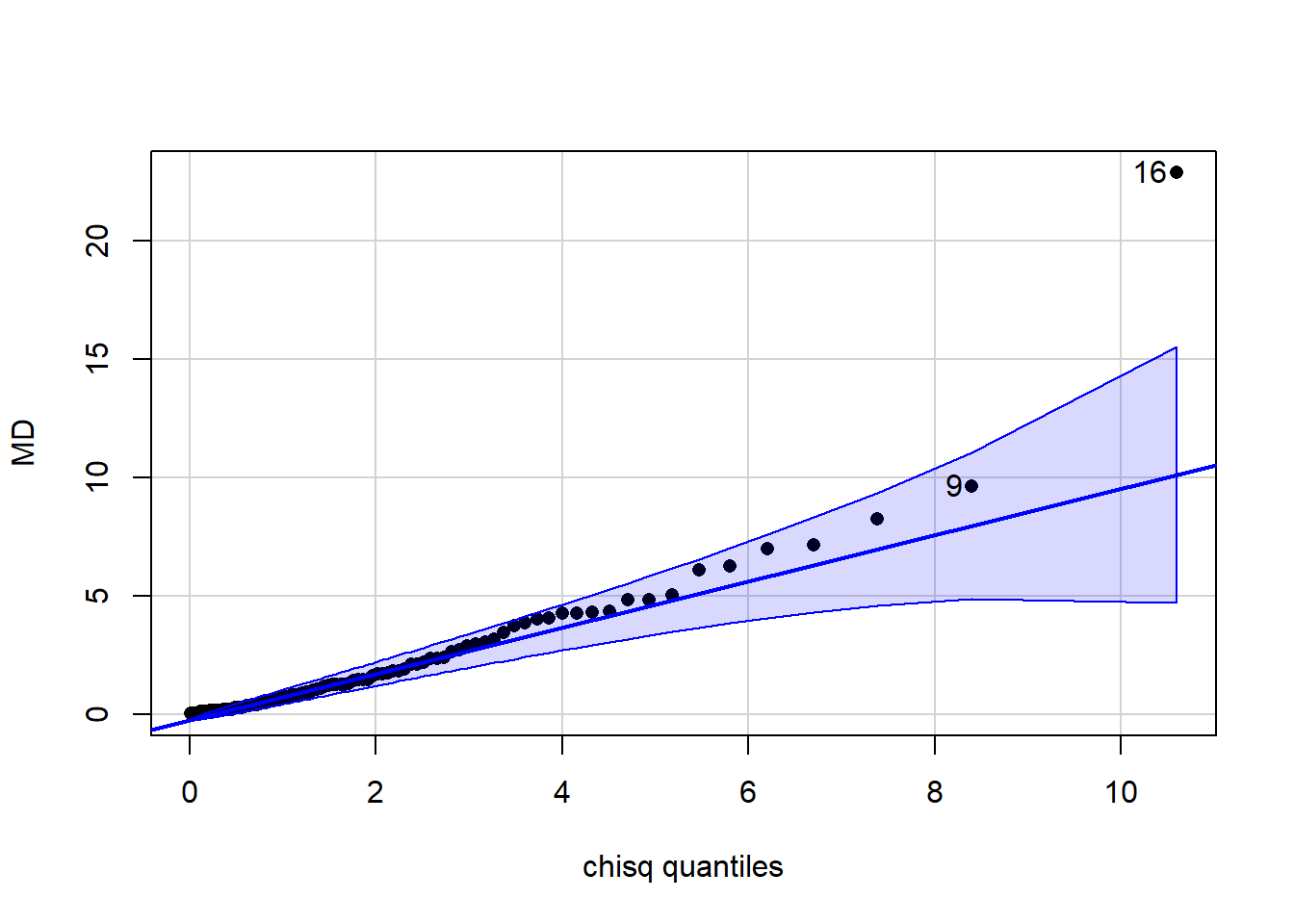
## [1] 16 9Gleiches gilt auch für den Q-Q-Plot, der hier ebenfalls gegen die \(\chi^2(df=2)\)-Verteilung abgetragen wurde. Der Q-Q-Plot hat außerdem als Output die Fallnummer (Pbn-Nr) der extremeren Werte - hier die Nummern 9 und 16. Insgesamt ist also zu sagen, dass die Mahalanobisdistanz nicht der multivariaten Normalverteilungsannahme widerspricht. Der Ellipsenplot oben zeigt zwei multivariat-normalverteilte Variablen. Die Normalverteilungsdichte können wir uns dort wie einen Hügel vorstellen, der aus dem Bildschirm wächst, wobei hellere Kurven für eine größere Höhe des Hügels sprechen.
Nach der Testung der multivariaten Normalverteilung, können wir uns nun den Ausreißern zuwenden. Zum Bestimmen der kritischen Distanz nehmen wir die \(\chi^2\) Verteilung heran. Wir bestimmen mit qchisq den kritischen Wert, wobei als \(p\)-Wert hier meist ein \(\alpha\)-Niveau von .01 oder .001 herangezogen wird, damit wir nicht fälschlicherweise zu viele Werte aussortieren. Hierbei übergeben wir dem Argument p das \(\alpha\)-Niveau, lower.tail = F besagt, dass wir damit die obere Grenze meinen (also mit p gerade die Wahrscheinlichkeit meinen, einen extremeren Wert zu finden), df = 2 stellt die Freiheitsgrade ein (hier = 2, da 2 Variablen):
qchisq(p = .01, lower.tail = F, df = 2) # alpha = 1%## [1] 9.21034qchisq(p = .001, lower.tail = F, df = 2) # alpha = 0.1%## [1] 13.81551Nun können wir die Mahalanobisdistanz untersuchen:
MD## [1] 0.88733518 0.21566377 2.41458442 0.13177919 1.27927630 4.28965128
## [7] 0.03451167 0.33661264 9.62502968 1.22925996 3.44404519 1.06648706
## [13] 1.47842392 1.83106572 4.29879647 22.89143112 0.21555167 0.49834275
## [19] 2.65724843 1.81949191 2.12850866 1.31066833 0.14334729 0.15748576
## [25] 1.71430321 1.74615142 1.00646134 0.17988251 5.04513115 7.14678462
## [31] 0.16464887 7.01861572 4.85177478 1.19043337 2.72462931 4.34611563
## [37] 0.52793945 0.80019408 0.74740206 0.57368396 0.28016792 6.27035221
## [43] 4.07657057 0.04345642 0.81076314 2.97056512 2.10323417 1.08103246
## [49] 0.60950451 2.90704907 0.10986424 6.09359260 0.94728160 2.35683503
## [55] 0.03620534 0.40241153 1.62654030 1.72697393 3.75760606 1.46629223
## [61] 0.71323544 0.41142182 0.39965918 0.61656862 0.37536805 0.22094404
## [67] 3.04527077 0.27381745 0.79358572 3.86151524 3.19370574 0.19468438
## [73] 0.29354422 2.35326071 0.95634005 0.07240648 1.92700957 1.44365936
## [79] 0.12899197 4.04487099 0.42931176 0.05394028 4.83779128 0.15390664
## [85] 2.20766936 1.27108100 0.37456784 0.20895062 1.47329914 1.26292357
## [91] 1.25364137 0.67270315 0.85951141 4.25348827 0.28156897 0.18421086
## [97] 0.47650958 0.13256756 8.26520343 0.18224578Hier alle Werte durch zugehen ist etwas lästig. Natürlich können wir den Vergleich mit den kritischen Werten auch automatisieren und z.B. uns nur diejenigen Mahalanobisdistanzwerte ansehen, die größer als der kritische Wert zum \(\alpha\)-Niveau von 1% sind. Wenn wir den which Befehl nutzen, so erhalten wir auch noch die Probandennummer der möglichen Ausreißer.
MD[MD > qchisq(p = .01, lower.tail = F, df = 2)] # Mahalanobiswerte > krit. Wert## [1] 9.62503 22.89143which(MD > qchisq(p = .01, lower.tail = F, df = 2)) # Pbn-Nr.## [1] 9 16Auf dem \(\alpha\)-Niveau von 1% gäbe es 2 Ausreißer (Pbn-Nr = 9, 16), auf dem von 0.1% nur noch einen (Pbn-Nr = 16). ***
Appendix
Appendix A
Regression “zu Fuß”
Wir schauen uns nun ein weiteres Beispiel an und berechnen alle Koeffizienten zunächst mit lm und anschließend “zu Fuß”. Wir wollen folgendes Modell schätzen
\[y_{i,math} = b_0 +b_{reading}x_{i,reading} + b_{IQ}x_{i,IQ} + e_i\] oder in Matrixform:
\[\begin{align} \begin{pmatrix} y_1\\y_2\\y_3\\y_4\\...\\y_{100}\end{pmatrix} = b_{0} * \begin {pmatrix}1\\1\\1\\1\\...\\1\end{pmatrix} + b_{reading} * \begin {pmatrix}x_{reading1}\\x_{reading2}\\x_{reading3}\\x_{reading4}\\...\\y_{reading100}\end{pmatrix} + b_{IQ} * \begin {pmatrix}x_{IQ1}\\x_{IQ2}\\x_{IQ3}\\x_{IQ4}\\...\\x_{IQ100}\end{pmatrix} + \begin {pmatrix}e_1\\e_2\\e_3\\e_4\\...\\e_{100}\end{pmatrix} \end{align}\]
Mit lm kommen wir zu folgendem Ergebnis; das Modell nennen wir phantasievoll our_next_model:
our_next_model <- lm(math ~ reading + IQ, data = Schulleistungen)
our_next_model##
## Call:
## lm(formula = math ~ reading + IQ, data = Schulleistungen)
##
## Coefficients:
## (Intercept) reading IQ
## 58.17167 -0.03585 5.30982Nun schätzen wir diese Parameter zu Fuß:
# Vektor Y
y <- Schulleistungen$math
head(y)## [1] 451.9832 589.6540 509.3267 560.4300 659.4524 602.8577# Matrix X vorbereiten (Spalten mit beiden Prädiktoren + Spalte mit Einsen anfügen)
X <- as.matrix(Schulleistungen[,c("reading", "IQ")])
X <- cbind(rep(1,nrow(X)), X)
head(X)## reading IQ
## [1,] 1 449.5884 81.77950
## [2,] 1 544.8495 106.75898
## [3,] 1 331.3466 99.14033
## [4,] 1 531.5384 111.91499
## [5,] 1 604.3759 116.12682
## [6,] 1 308.7457 106.14127\[\begin{align}y = \begin{pmatrix}451,98\\589,65\\509,33\\560,43\\...\\603,18\end{pmatrix}\end{align}\]
\[\begin{align}X = \begin{pmatrix}1 & 449,59 & 81,78\\1 & 544,85 & 106,76\\1 & 331,35 & 99,14\\1 & 531,54 & 111,91\\ ... & ... & ... \\1 & 487,22 & 106,13\end{pmatrix}\end{align}\]
Vorgehen bei der Berechnung der Regressionsgewichte:
- Berechnung der Kreuzproduktsumme (X’X)
- Berechnung der Inversen der Kreuzproduktsumme (\((X'X)^{-1}\))
- Berechnung des Kreuzproduksummenvektors (X’y)
- Berechnung des Einflussgewichtsvektors
1. Berechnung der Kreuzproduktsumme (\(X’X\))
Die Kreuzproduktsumme (X’X) wird berechnet, indem die transponierte Matrix X (X’) mit der Matrix X multipliziert wird. Die transponierte Matrix X’ erhalten Sie durch den Befehl t(X).
\[\begin{align} \dfrac{}{X'\begin{pmatrix} 1 & 1 & 1 & 1 & ... & 1\\449,58 & 544,85 & 331,35 & 531,54 & ... & 487,22\\81,78 & 106,76 & 99,14 & 111,91 & ... & 106,13\end{pmatrix}} \dfrac{\begin{pmatrix}1 & 449,59 & 81,78\\1 & 544,85 & 106,76\\1 & 331,35 & 99,14\\1 & 531,54 & 111,91\\... & ... & ... \\1 & 487,22 & 106,13\end{pmatrix}X} {\begin{pmatrix}100,00 & 49606,60 & 9813,43\\49606,61 & 25730126,10 & 4962448,08\\9813,43 & 4962448,10 & 987595,82\end{pmatrix}X'X}\end{align}\]
# Berechnung der Kreuzproduktsumme X’X in R
XX <- t(X) %*% X # X' erhalten Sie durch t(X)
XX## reading IQ
## 100.000 49606.6 9813.425
## reading 49606.605 25730126.1 4962448.077
## IQ 9813.425 4962448.1 987595.8242. Berechnung der Inversen der Kreuzproduktsumme \((X'X)^{-1}\)
Die Inverse der Kreuzproduktsumme kann in R durch den solve-Befehl berechnet werden.
# Berechnung der Inversen (mit Regel nach Sarrus) in R
solve(XX)## reading IQ
## 0.4207610612 -1.568521e-04 -3.392822e-03
## reading -0.0001568521 1.316437e-06 -5.056210e-06
## IQ -0.0033928220 -5.056210e-06 6.013228e-05\[\begin{align}(X'X)^{-1}= \begin{pmatrix}0,42 & -1,56e^{-04} & -3,39^{-03}\\-0,00 & 1,32e^{-06} & -5,06e^{-06}\\-0,00 & -5,06e^{-06} & 6,01e^{-05}\end{pmatrix}\end{align}\]
3. Berechnung des Kreuzproduksummenvektors (X’y)
Der Kreuzproduktsummenvektor (X’y) wird durch die Multiplikation der transponierten X Matrix (X’) und des Vektors y berechnet.
\[\begin{align} \dfrac{}{X'\begin{pmatrix}1 & 1 & 1 & 1 & ... & 1\\449,58 & 544,85 & 331,35 & 531,54 & ... & 487,22\\81,78 & 106,76 & 99,14 & 111,91 & ... & 106,13\end{pmatrix}} \dfrac{\begin{pmatrix}451,98\\589,65\\509,33\\560,43\\...\\603,18\end{pmatrix}y} {\begin{pmatrix}56146,45\\28313059,77\\5636931,00\end{pmatrix}X'y} \end{align}\]
#Berechnung des Kreuzproduksummenvektors X`y in R
Xy <- t(X) %*% y
head(Xy)## [,1]
## 56146.45
## reading 28313059.77
## IQ 5636931.004. Berechnung des Einflussgewichtsvektors
Die geschätzten Regressionsgewichte nach dem Kriterium der kleinsten Quadrate werden berechnet, indem die Inverse der Kreuzproduktsumme \((X'X)^{-1}\) mit dem Kreuzproduksummenvektor (X’y) multipliziert wird. Den Vektor mit den vorhergesagten Werte von y (\(\hat{y}\)) können Sie durch die Multiplikation der X-Matrix mit den Regressionsgewichten (\(\hat{b}\)) berechnen.
\[\begin{align} \dfrac{}{(X'X)^{-1}\begin{pmatrix}0,42 & -1,56e^{-04} & -3,39^{-03}\\-0,00 & 1,32e^{-06} & -5,06e^{-06}\\-0,00 & -5,06e^{-06} & 6,01e^{-05}\end{pmatrix}} \dfrac{\begin{pmatrix}56146,45\\28313059,77\\5636931,00\end{pmatrix}X'y} {\begin{pmatrix}58,17\\-0,04\\5,31\end{pmatrix}\hat{b}} \end{align}\]
# Berechnung des Einflussgewichtsvektors in R
b_hat <- solve(XX) %*% Xy # Vektor der geschätzten Regressionsgewichte
b_hat## [,1]
## 58.17167003
## reading -0.03584686
## IQ 5.30981976y_hat <- as.vector(X %*% b_hat) # Vorhersagewerte
head(y_hat)## [1] 476.2897 605.5115 572.7112 633.3661 653.1192 610.6951\[\begin{align} \hat{y}_{math} = \begin{pmatrix}476,29\\605,51\\572,71\\633,37\\...\\604,22\end{pmatrix} \end{align}\]
Tatsächlich kommen wir zum selben Ergebnis wie mit lm. Dies liegt daran, dass lm im Grunde diese Matrixoperationen für uns durchführt!
Berechnung der standardisierten Regressionsgewichte
Bisher wurden aber nur die unstandardisierten Regressionsgewichte berechnet. Diese haben den Vorteil, leichter interpretierbar zu sein. So wird das unstandardisierte Regressionsgewicht folgendermaßen interpretiert: wenn sich die unabhängige Variable um eine Einheit verändert, verändert sich die abhängige Variable um den unstandardisierten Koeffizienten. Der Nachteil dieser unstandardisierten Regressionsgewichte ist jedoch, dass die Regressionsgewichte nicht vergleichbar sind. Demzufolge kann anhand der Größe der Regressionsgewichte nicht gesagt werden, welcher Regressionskoeffizient eine stärkere Erklärungskraft hat.
Daher werden die Regressionsgewichte häufig standardisiert. Durch die Standardisierung sind die Regressionsgewichte nicht mehr von der ursprünglichen Skala abhängig und haben daher den Vorteil, dass sie miteinander verglichen werden können. Allerdings sind die standardisierten Regressionsgewichte nicht mehr so leicht zu interpretieren. Die Interpretation der standardisierten Regressionsgewichte lautet: wenn sich die unabhängige Variable um eine Standardabweichung erhöht (und unter Kontrolle weiterer unabhängiger Variablen), so beträgt die erwartete Veränderung in der abhängigen Variable \(\beta\) Standardabweichungen (das standardisierte Interzept ist Null).
Die standardisierten Regressionsgewichte können über den standardisierten y Vektor und die standardisierte Matrix (dazu scale-Befehl in R) ermittelt werden.
#Berechnung der standardisierten Regressionsgewichte
y_s <- scale(y) # Standardisierung y
X_s <- scale(X) # Standardisierung X
X_s[,1] <- 1 # Einsenvektor ist nach Standardisierung zunächst NaN, muss wieder gefüllt werden
b_hat_s <- solve(t(X_s)%*% X_s) %*% t(X_s)%*%y_s #Regressionsgewichte aus den standardisierten Variablen
round(b_hat_s, 3)## [,1]
## 0.000
## reading -0.033
## IQ 0.716Der lm Befehl mit dem zusatz lm.beta aus dem gleichnamigen Paket führt zu:
lm.beta(our_next_model)##
## Call:
## lm(formula = math ~ reading + IQ, data = Schulleistungen)
##
## Standardized Coefficients::
## (Intercept) reading IQ
## 0.0000000 -0.0326760 0.7161496Berechnung des globalen Signifikanztests
Determinationskoeffizient \(R^2\)
Der Determinationskoeffizient \(R^2\) gibt an, wie viel Varianz in der abhängigen Variable durch die unabhängigen Variablen erklärt werden kann:
\(R^2= \dfrac{Q_d}{Q_d + Q_e}\)
# Determinationskoeffizient R2
Q_d <- sum((y_hat - mean(y))^2) # Regressionsquadratsumme
Q_e <- sum((y - y_hat)^2) # Fehlerquadratsumme
R2 <- Q_d / (Q_d + Q_e) # Determinationskoeffizient R2\(R^2= \dfrac{Q_d}{Q_d + Q_e} = \dfrac{6.5805169\times 10^{5}}{6.5805169\times 10^{5} + 6.9223863\times 10^{5}} = 0.49\)
\(F\)-Wert
Der F-Wert dient zur Überprüfung der Gesamtsignifikanz des Modells.
# F-Wert
n <- length(y) # Fallzahl (n=100)
m <- ncol(X)-1 # Zahl der Prädiktoren (m=2)
F_omn <- (R2/m) / ((1-R2)/(n-m-1)) # F-Wert
F_krit <- qf(.95, df1=m, df2=n-m-1) # kritischer F-Wert (alpha=5%)
p <- 1-pf(F_omn, m, n-m-1) # p-Wert\(F_{omn} = \dfrac{\dfrac{R^2}{m}}{\dfrac{1-R^2}{n-m-1}} = \dfrac{\dfrac{0.49}{2}}{\dfrac{1-0.49}{100-2-1}} = 46.1\)
\(df_1 = 2, df_1 = n-m-1 = 100-2-1 =97\)
\(F_{krit}(\alpha=.05, df_1=2, df_2= 97)= 3.09\)
\(p=0.00000000000000844\)
Appendix B
Regressionsmodell
Folgende Befehle führen zum gleichen Ergebnis wie:
lm(reading ~ 1 + female + IQ, data = Schulleistungen)##
## Call:
## lm(formula = reading ~ 1 + female + IQ, data = Schulleistungen)
##
## Coefficients:
## (Intercept) female IQ
## 88.209 38.470 3.944Das Interzept kann explizit mit angegeben werden (falls Sie 0 + schreiben, setzen Sie das Interzept auf 0, was sich entsprechend auf die Parameterschätzungen auswirken wird, falls das Interzept von 0 verschieden ist!):
lm(reading ~ 0 + female + IQ, data = Schulleistungen)##
## Call:
## lm(formula = reading ~ 0 + female + IQ, data = Schulleistungen)
##
## Coefficients:
## female IQ
## 45.187 4.785Dem Output ist zu entnehmen, dass die Parameterschätzungen sich drastisch geändert haben!
Lassen wir das Interzept in der Schreibweise weg, so wird es per Default mitgeschätzt.
lm(reading ~ female + IQ, data = Schulleistungen)##
## Call:
## lm(formula = reading ~ female + IQ, data = Schulleistungen)
##
## Coefficients:
## (Intercept) female IQ
## 88.209 38.470 3.944Mit formula benutzen wir nicht die Position in der Funktion, sondern das Argument für die Formel:
lm(formula = 1 + reading ~ female + IQ, data = Schulleistungen) ##
## Call:
## lm(formula = 1 + reading ~ female + IQ, data = Schulleistungen)
##
## Coefficients:
## (Intercept) female IQ
## 89.209 38.470 3.944Wir können also auch einfach die Reihenfolge umdrehen, solange wir Argumente benutzen:
lm(data = Schulleistungen, formula = 1 + reading ~ female + IQ) ##
## Call:
## lm(formula = 1 + reading ~ female + IQ, data = Schulleistungen)
##
## Coefficients:
## (Intercept) female IQ
## 89.209 38.470 3.944Die Formel kann auch in Anführungszeichen geschrieben werden:
lm("reading ~ 1 + female + IQ", data = Schulleistungen) ##
## Call:
## lm(formula = "reading ~ 1 + female + IQ", data = Schulleistungen)
##
## Coefficients:
## (Intercept) female IQ
## 88.209 38.470 3.944Wir können auf die Datensatzspezifizierung verzichten, indem wir die Variablen direkt ansprechen (es ändern sich entsprechend die Namen der Koeffizienten im Output):
lm(Schulleistungen$reading ~ 1 + Schulleistungen$female + Schulleistungen$IQ) ##
## Call:
## lm(formula = Schulleistungen$reading ~ 1 + Schulleistungen$female +
## Schulleistungen$IQ)
##
## Coefficients:
## (Intercept) Schulleistungen$female Schulleistungen$IQ
## 88.209 38.470 3.944Wir können auch neue Variablen definieren, um diese dann direkt anzusprechen (es ändern sich entsprechend die Namen der Koeffizienten):
AV <- Schulleistungen$reading
UV1 <- Schulleistungen$female
UV2 <- Schulleistungen$IQ
lm(AV ~ 1 + UV1 + UV2)##
## Call:
## lm(formula = AV ~ 1 + UV1 + UV2)
##
## Coefficients:
## (Intercept) UV1 UV2
## 88.209 38.470 3.944Selbstverständlich gibt es auch noch weitere Befehle, die zum selben Ergebnis kommen. Sie sehen, dass Sie in R in vielen Bereichen mit leicht unterschiedlichem Code zum selben Ergebnis gelangen!
Appendix C
ggplot2
Im folgenden Block sehen wir den Code für ein Histogramm in ggplot2-Notation (das Paket muss natürlich installiert sein: install.packages(ggplot2)). Hier sind einige Zusatzeinstellungen gewählt, die das Histogramm optisch aufbereiten.
library(ggplot2)## Warning: Paket 'ggplot2' wurde unter R Version 4.1.2 erstelltdf_res <- data.frame(res) # als Data.Frame für ggplot
ggplot(data = df_res, aes(x = res)) +
geom_histogram(aes(y =..density..),
bins = 15, # Wie viele Balken sollen gezeichnet werden?
colour = "blue", # Welche Farbe sollen die Linien der Balken haben?
fill = "skyblue") + # Wie sollen die Balken gefüllt sein?
stat_function(fun = dnorm, args = list(mean = mean(res), sd = sd(res)), col = "darkblue") + # Füge die Normalverteilungsdiche "dnorm" hinzu und nutze den empirischen Mittelwert und die empirische Standardabweichung "args = list(mean = mean(res), sd = sd(res))", wähle dunkelblau als Linienfarbe
labs(title = "Histogramm der Residuen mit Normalverteilungsdichte", x = "Residuen") # Füge eigenen Titel und Achsenbeschriftung hinzu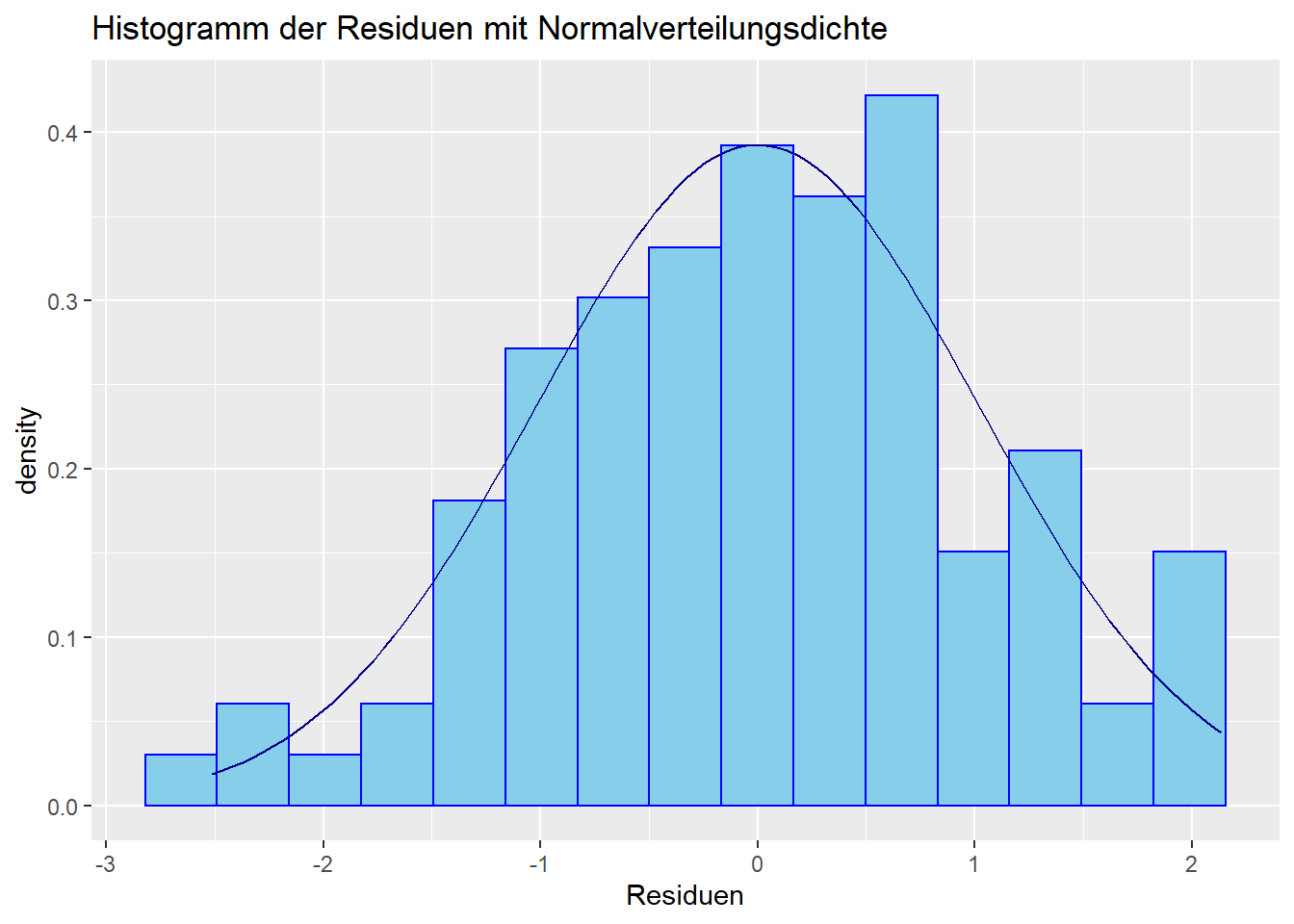
Nutzen wir nur die Defaulteinstellung des Histogramms (bis auf bins = 15 - für die Vergleichbarkeit der beiden Grafiken), sieht es so aus:
# hier nochmal nur das Nötigste:
ggplot(data = df_res, aes(x = res)) +
geom_histogram(aes(y =..density..), bins = 15) +
stat_function(fun = dnorm, args = list(mean = mean(res), sd = sd(res)))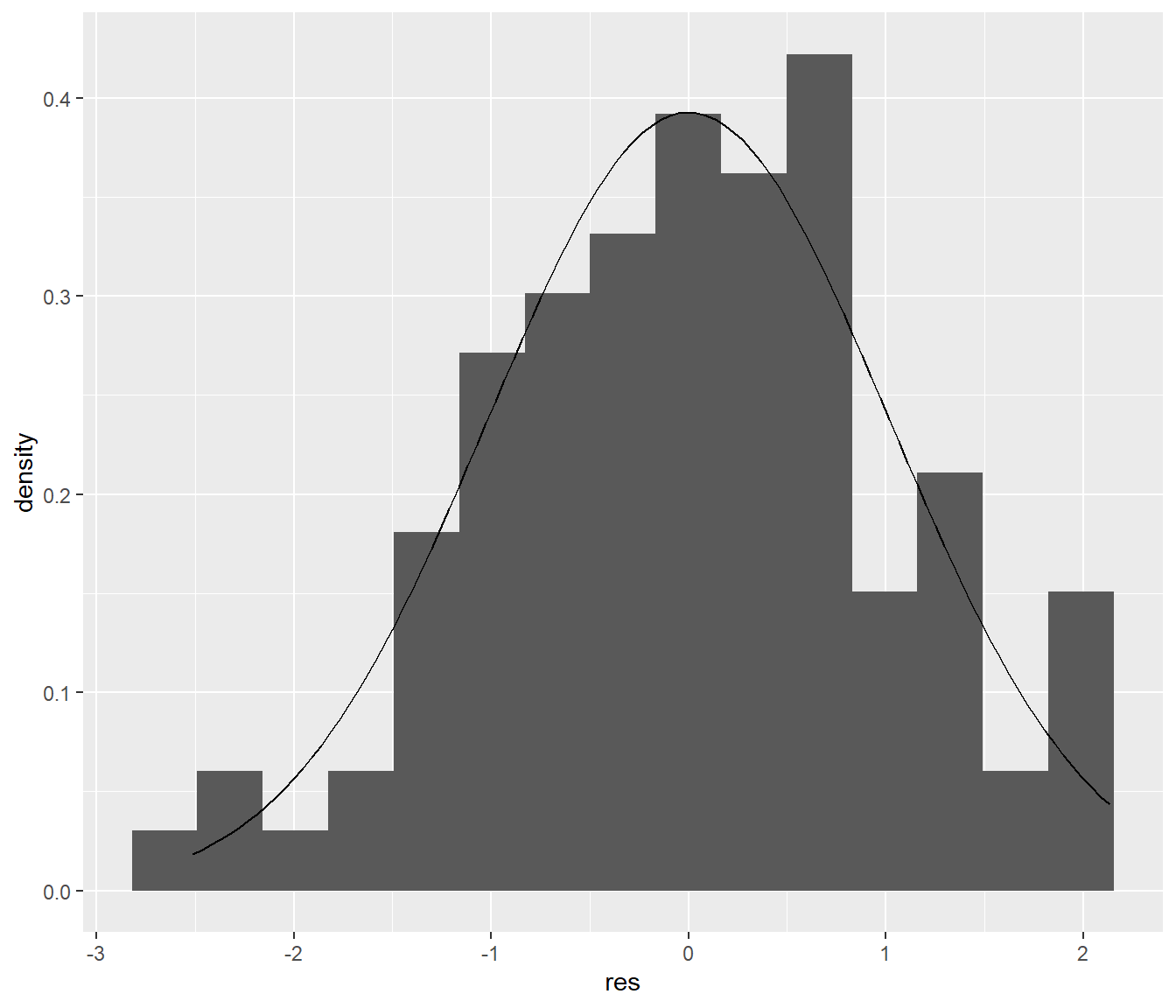
Hier sind auch noch die weiteren Histogramme dieser Sitzung mit ggplot aufbereitet:
Hat-Values
n <- length(residuals(our_model))
h <- hatvalues(our_model) # Hebelwerte
df_h <- data.frame(h) # als Data.Frame für ggplot
ggplot(data = df_h, aes(x = h)) +
geom_histogram(aes(y =..density..), bins = 15)+
geom_vline(xintercept = 4/n, col = "red") # Cut-off bei 4/n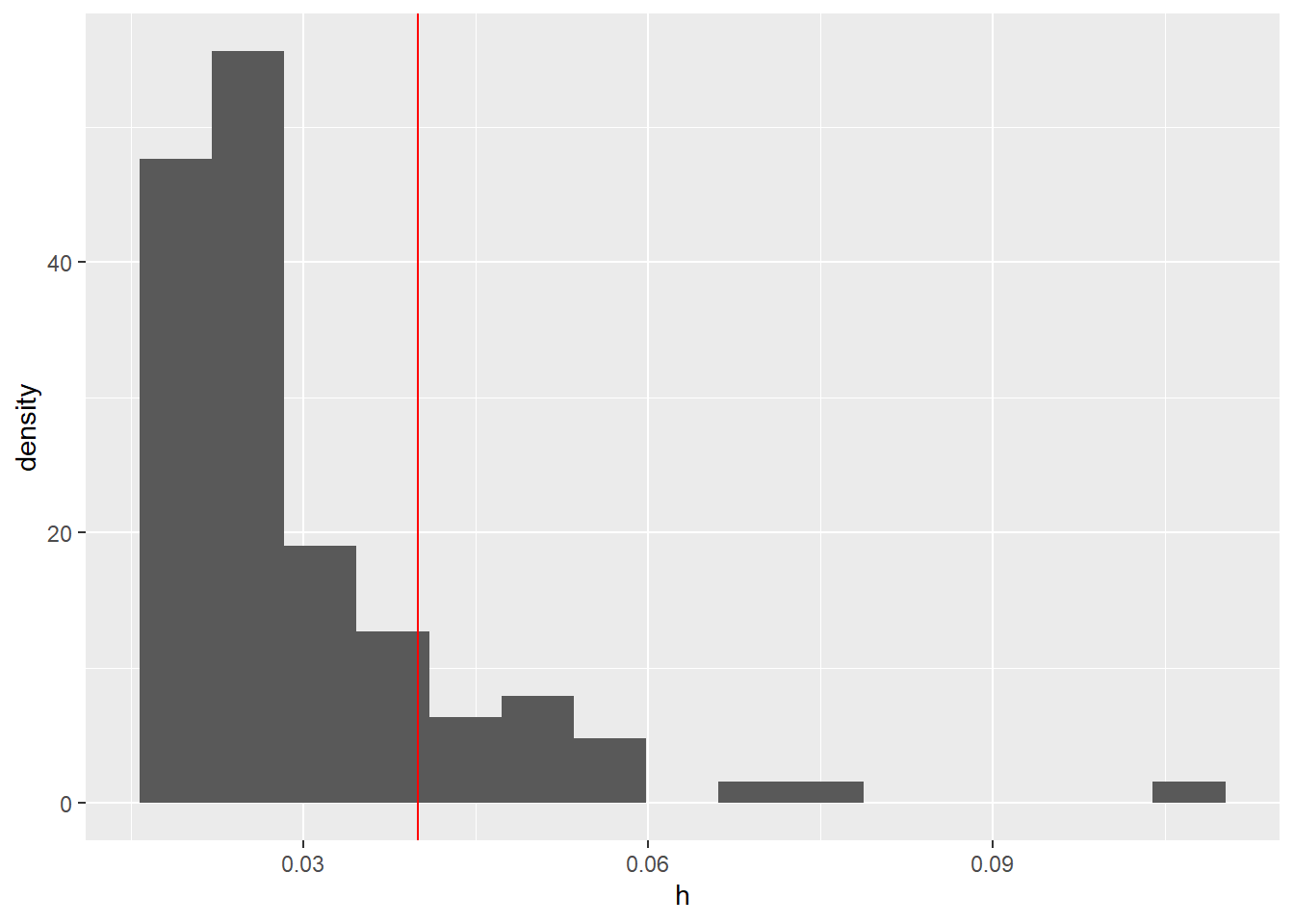
Cooks-Distanz:
# Cooks Distanz
CD <- cooks.distance(our_model) # Cooks Distanz
df_CD <- data.frame(CD) # als Data.Frame für ggplot
ggplot(data = df_CD, aes(x = CD)) +
geom_histogram(aes(y =..density..), bins = 15)+
geom_vline(xintercept = 1, col = "red") # Cut-Off bei 1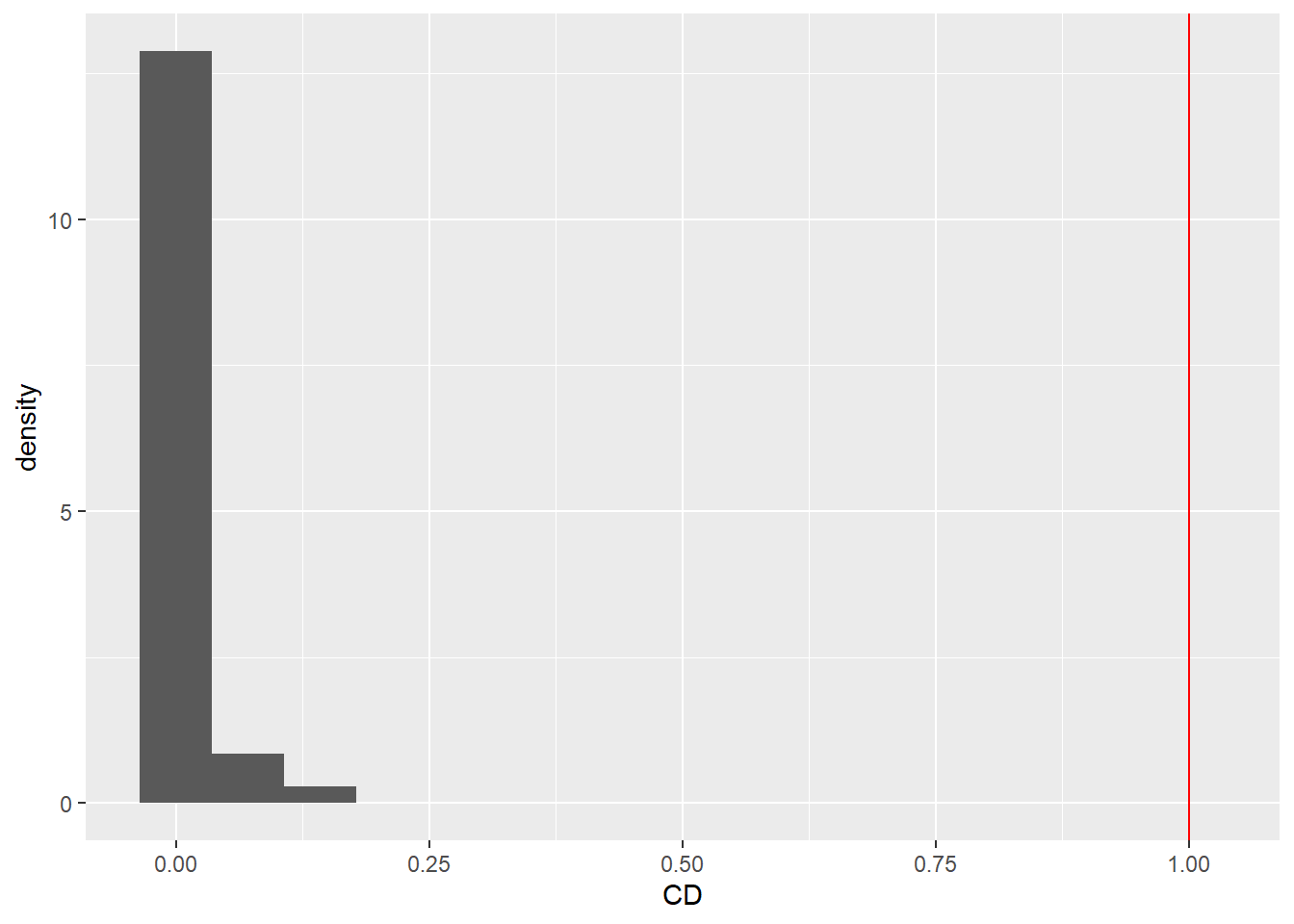
Appendix D
Multikollinearität und Standardfehler
Dies ist der Appendix A der Bachelor Sitzung zu Voraussetzungen der Regression von Julien Irmer.
Im Folgenden stehen \(\beta\)s für unstandardisierte Regressionskoeffizienten.
Für eine einfache Regressionsgleichung mit \[Y_i=\beta_0 + \beta_1x_{i1} + \beta_2x_{i2} + \varepsilon_i\] kann die selbe Gleichung auch in Matrixnotation formuliert werden \[Y = X\beta + \varepsilon.\] Hier ist \(X\) die Systemmatrix, welche die Zeilenvektoren \(X_i=(1, x_{i1}, x_{i2})\) enthält. Die Standardfehler, welche die Streuung der Parameter \(\beta:=(\beta_0,\beta_1,\beta_2)\) beschreiben, lassen sich wie folgt ermitteln. Wir bestimmen zunächst die Matrix \(I\) wie folgt \[I:=(X'X)^{-1}\hat{\sigma}^2_e,\] wobei \(\hat{\sigma}^2_e\) die Residualvarianz unserer Regressionsanalyse ist (also der nicht-erklärte Anteil an der Varianz von \(Y\)). Aus der Matrix \(I\) erhalten wir die Standardfehler sehr einfach: Sie stehen im Quadrat auf der Diagonalen. Das heißt, die Standardfehler sind \(SE(\beta)=\sqrt{\text{diag}(I)}\) (Wir nehmen mit \(\text{diag}\) die Diagonalelemente aus \(I\) und ziehen aus diesen jeweils die Wurzel: der erste Eintrag ist der \(SE\) von \(\beta_0\); also \(SE(\beta_0)=\sqrt{I_{11}}\); der zweite von \(\beta_1\);\(SE(\beta_1)=\sqrt{I_{22}}\); usw.). Was hat das nun mit der Kollinearität zu tun? Wir wissen, dass in \(X'X\) die Information über die Kovariation im Datensatz steckt (dafür muss nur noch durch die Stichprobengröße geteilt werden und das Vektorprodukt der Mittelwerte abgezogen werden; damit wir eine Zentrierung um den Mittelwert sowie eine Normierung an der Stichprobengröße vorgenommen). Beispielsweise lässt sich die empirische Kovarianzmatrix \(S\) zweier Variablen \(z_1\) und \(z_2\) sehr einfach bestimmen mit \(Z:=(z_1, z_2)\): \[ S=\frac{1}{n}Z'Z - \begin{pmatrix}\overline{z}_1\\\overline{z}_2 \end{pmatrix}\begin{pmatrix}\overline{z}_1&\overline{z}_2 \end{pmatrix}.\] Weitere Informationen hierzu (Kovarianzmatrix und Standardfehler) sind im Appendix B (sowie auch in einigen Kapiteln von Eid et al. (2017) Unterpunkt 5.2-5.4 bzw. ab Seite 1058) nachzulesen.
Insgesamt bedeutet dies, dass die Standardfehler von der Inversen der Kovarianzmatrix unserer Daten sowie von der Residualvarianz abhängen. Sie sind also groß, wenn die Residualvarianz groß ist (damit ist die Vorhersage von \(Y\) schlecht) oder wenn die Inverse der Kovarianzmatrix groß ist (also wenn die Variablen stark redundant sind und somit hoch miteinander korrelieren). Nehmen wir dazu der Einfachheit halber an, dass \(\hat{\sigma}_e^2=1\) (es geht hier nur um eine numerische Präsentation der Effekte, nicht um ein sinnvolles Modell) sowie \(n = 100\) (Stichprobengröße). Zusätzlich gehen wir von zentrierten Variablen (Mittelwert von 0) aus. Dann lässt sich aus \(X'X\) durch Division durch \(100\) die Kovarianzmatrix der Variablen bestimmen. Wir gucken uns drei Fälle an:
\[\begin{align*} \text{Fall 1: } X'X&=\begin{pmatrix}100&0&0\\0&100&0\\0&0&100\end{pmatrix},\\ \text{Fall 2: } X'X&=\begin{pmatrix}100&0&0\\0&100&99\\0&99&100\end{pmatrix}, \\ \text{Fall 3: } X'X&=\begin{pmatrix}100&0&0\\0&100&100\\0&100&100\end{pmatrix}. \end{align*}\]
Hierbei ist zu beachten, dass \(X\) die Systemmatrix ist, welche auch die \(1\) des Interzepts enthält. Natürlich ist eine Variable von einer Konstanten unabhängig, weswegen die erste Zeile und Spalte von \(X'X\) jeweils der Vektor \((100, 0, 0)\) ist. Die zugehörigen Korrelationsmatrizen können durch Divison durch 100 berechnet werden (da wir zentrierte Variablen haben, die Stichprobengröße gleich 100 ist und die Varianzen der Variablen gerade 100 sind!). Wir betrachten nur die Minormatrizen, aus welchen die 1. Zeile und die 1. Spalte gestrichen wurden. Diese teilen wir durch 100 und erhalten die Korrelationsmatrix der Variablen:
\[\begin{align*} \text{Fall 1: }\Sigma_1&=\begin{pmatrix}1&0\\0&1\end{pmatrix},\\ \text{Fall 2: }\Sigma_2&=\begin{pmatrix}1&.99\\.99&1\end{pmatrix},\\ \text{Fall 3: } \Sigma_3&=\begin{pmatrix}1&1\\1&1\end{pmatrix}. \end{align*}\]
Im Fall 1 sind die zwei Variablen unkorreliert. Die Inverse ist leicht zu bilden.
XX_1 <- matrix(c(100,0,0,
0,100,0,
0,0,100),3,3)
XX_1 # Die Matrix X'X im Fall 1## [,1] [,2] [,3]
## [1,] 100 0 0
## [2,] 0 100 0
## [3,] 0 0 100I_1 <- solve(XX_1)*1 # I (*1 wegen Residualvarianz = 1)
I_1## [,1] [,2] [,3]
## [1,] 0.01 0.00 0.00
## [2,] 0.00 0.01 0.00
## [3,] 0.00 0.00 0.01sqrt(diag(I_1)) # Wurzel aus den Diagonalelementen der Inverse = SE, wenn sigma_e^2=1## [1] 0.1 0.1 0.1Die Standardfehler sind nicht sehr groß: alle liegen bei \(0.1\).
Im Fall 2 sind die zwei Variablen fast perfekt (zu \(.99\)) korreliert - es liegt hohe Multikollinearität vor. Die Inverse ist noch zu bilden. Die Standardfehler sind deutlich erhöht im Vergleich zu Fall 1.
XX_2 <- matrix(c(100,0,0,
0,100,99,
0,99,100),3,3)
XX_2 # Die Matrix X'X im Fall 2## [,1] [,2] [,3]
## [1,] 100 0 0
## [2,] 0 100 99
## [3,] 0 99 100I_2 <- solve(XX_2)*1 # I (*1 wegen Residualvarianz = 1)
I_2## [,1] [,2] [,3]
## [1,] 0.01 0.0000000 0.0000000
## [2,] 0.00 0.5025126 -0.4974874
## [3,] 0.00 -0.4974874 0.5025126sqrt(diag(I_2)) # SEs im Fall 2## [1] 0.1000000 0.7088812 0.7088812sqrt(diag(I_1)) # SEs im Fall 1## [1] 0.1 0.1 0.1Die Standardfehler des Fall 2 sind sehr groß im Vergleich zu Fall 1 (mehr als sieben Mal so groß - was de facto schon gigantisch ist!); nur der Standardfehler des Interzepts bleibt gleich. Die Determinante von \(X'X\) in Fall 2 liegt deutlich näher an \(0\) im Vergleich zu Fall 1; hier: \(10^6\).
det(XX_2) # Determinante Fall 2## [1] 19900det(XX_1) # Determinante Fall 1## [1] 1e+06Im Fall 3 sind die zwei Variablen perfekt korreliert - es liegt perfekte Multikollinearität vor. Die Inverse kann nicht gebildet werden (da \(\text{det}(X'X) = 0\)). Die Standardfehler können nicht berechnet werden. Eine Fehlermeldung wird ausgegeben.
XX_3 <- matrix(c(100,0,0,
0,100,100,
0,100,100),3,3)
XX_3 # Die Matrix X'X im Fall 3## [,1] [,2] [,3]
## [1,] 100 0 0
## [2,] 0 100 100
## [3,] 0 100 100det(XX_3) # Determinante on X'X im Fall 3## [1] 0I_3 <- solve(XX_3)*1 # I (*1 wegen Residualvarianz = 1)
I_3
sqrt(diag(I_3)) # Wurzel aus den Diagonalelementen der Inverse = SE, wenn sigma_e^2=1
# hier wird eine Fehlermeldung ausgegeben, wodurch der Code nicht ausführbar ist und I_3 nicht gebildet werden kann:
# Error in solve.default(XX_3) :
# Lapack routine dgesv: system is exactly singular: U[2,2] = 0Der VIF bzw. die Toleranz quantifizieren die Korrelation zwischen den beiden Variablen. Der VIF wäre in diesen Analysen im 1. Fall für beide Variablen 1, im 2. Fall für beide Variabeln 50.25 und im 3. Fall nicht berechenbar (bzw. \(\infty\)). Entsprechend wäre die Toleranz im 1. Fall 1 und 1, im 2. Fall 0.02 und 0.02 sowie im 3. Fall 0 und 0.
Dieser Exkurs zeigt, wie sich die Multikollinearität auf die Standardfehler und damit auf die Präzision der Parameterschätzung auswirkt. Inhaltlich bedeutet dies, dass die Prädiktoren redundant sind und nicht klar ist, welchem Prädiktor die Effekte zugeschrieben werden können.
Die Matrix \(I\) ist im Zusammenhang mit der Maximum-Likelihood-Schätzung die Inverse der Fischer-Information und enthält die Informationen der Kovariationen der Parameterschätzer (diese Informationen enthält sie hier im Übrigen auch!).
Literatur
Agresti, A, & Finlay, B. (2013). Statistical methods for the social sciences. (Pearson new international edition, 4th edition). Harlow, Essex : Pearson Education Limited.
Eid, M., Gollwitzer, M., & Schmitt, M. (2017). Statistik und Forschungsmethoden (5. Auflage, 1. Auflage: 2010). Weinheim: Beltz.
Pituch, K. A. & Stevens, J. P. (2016). Applied Multivariate Statistics for the Social Sciences (6th ed.). New York: Taylor & Francis.
- Blau hinterlegte Autor:innenangaben führen Sie direkt zur universitätsinternen Ressource.
